Ensamblaje del genoma fúngico de novo
Características del servicio
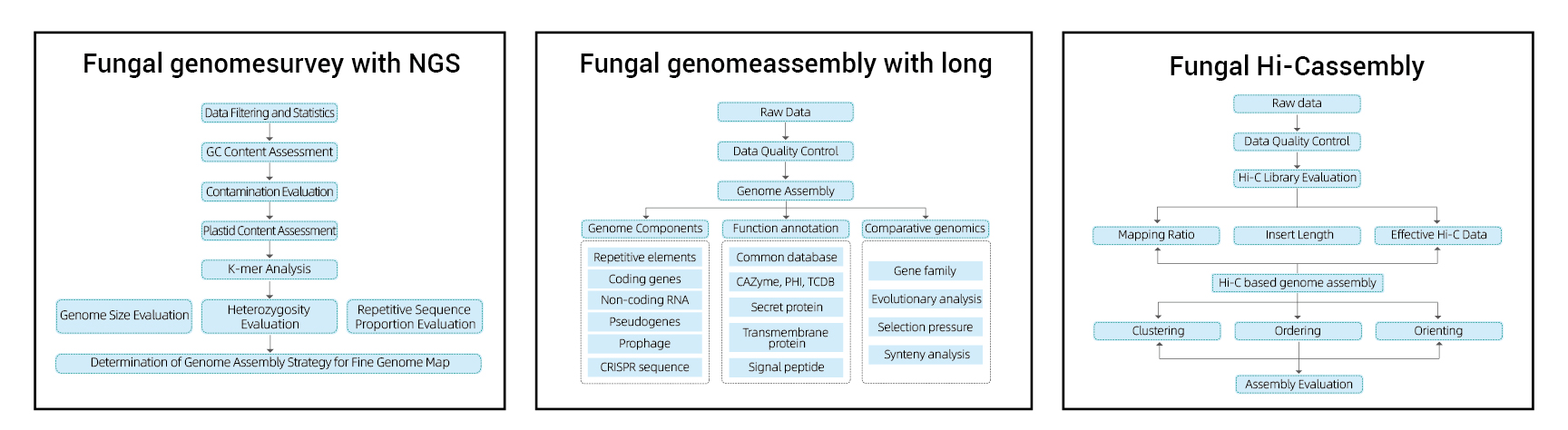
Con tres opciones posibles para elegir según el grado deseado de integridad del genoma:
● Opción de borrador del genoma: secuenciación de lectura corta con Illumina NovaSeq PE150.
● Opción de genoma fino de hongos:
Estudio del genoma: Illumina NovaSeq PE150.
Ensamblaje del genoma: PacBio Revio (lecturas HiFi) o Nanopore PromethION 48.
● Genoma fúngico a nivel cromosómico:
Estudio del genoma: Illumina NovaSeq PE150.
Ensamblaje del genoma: PacBio Revio (lecturas HiFi) o Nanopore PromethION 48.
Anclaje Contig con montaje Hi-C.
Ventajas del servicio
●Múltiples estrategias de secuenciación disponibles: Para diferentes objetivos de investigación y requisitos de integridad del genoma
●Flujo de trabajo completo de bioinformática:Esto incluye el ensamblaje del genoma y la predicción de múltiples elementos genómicos, la anotación de genes funcionales y el anclaje de contig.
●Amplia experiencia: Con más de 12 000 genomas microbianos reunidos, aportamos más de una década de experiencia, un equipo de análisis altamente capacitado, contenido completo y excelente soporte posventa.
●Soporte Postventa:Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Especificaciones de servicio
| Servicio | Estrategia de secuenciación | Control de calidad |
| Proyecto de genoma | Iluminación PE150 100x | Q30≥85% |
| Genoma fino | Estudio del genoma: Illumina PE150 50 x Montaje: PacBio HiFi 30x o Nanopore 100x | contig N50 ≥1Mb(Pacbio Unicelular) contig N50 ≥2Mb(ONT Unicelular) contig N50 ≥500kb(Otros) |
| Genoma a nivel cromosómico | Estudio del genoma: Illumina PE150 50 x Montaje: PacBio HiFi 30x o Nanopore 100x Montaje Hi-C 100x | Relación de anclaje Contig>90%
|
Requisitos de servicio
| Concentración (ng/μL) | Cantidad total (μg) | Volumen (μL) | DE260/280 | OD260/230 | |
| PacBio | ≥20 | ≥2 | ≥20 | 1.7-2.2 | ≥1,6 |
| nanoporo | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1.0-3.0 |
| iluminar | ≥1 | ≥0,06 | ≥20 | - | - |
Hongo unicelular: ≥3.5x1010 células
Hongo macro: ≥10 g
Flujo de trabajo del servicio

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de datos

Servicios postventa
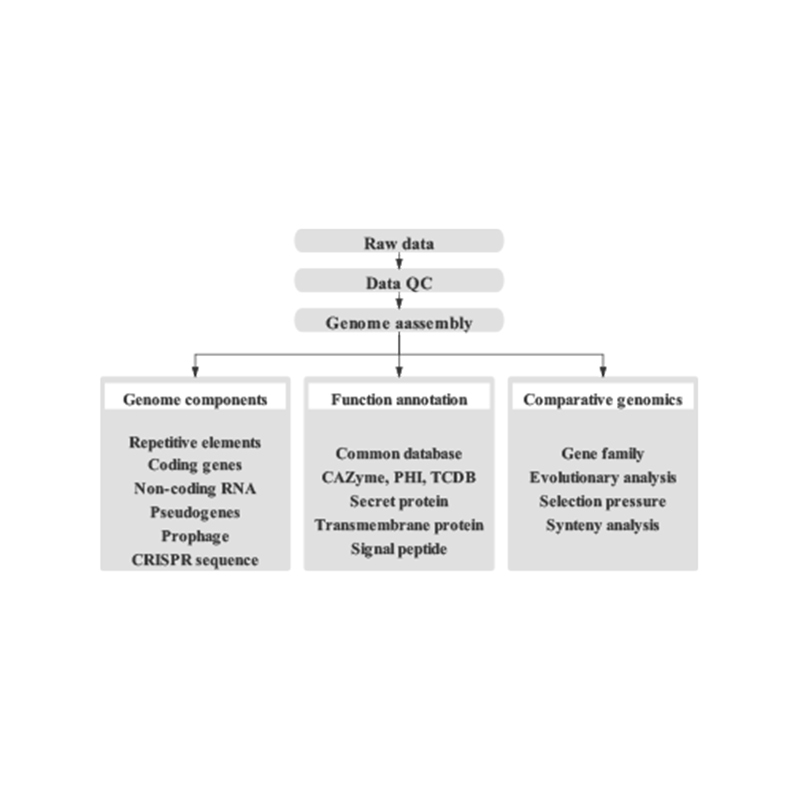
Incluye el siguiente análisis:
Encuesta del genoma:
- Control de calidad de datos de secuenciación.
- Estimación del genoma: tamaño, heterocigosidad, elementos repetitivos.
Ensamblaje fino del genoma:
- Control de calidad de datos de secuenciación
- De novoAsamblea
- Análisis de componentes del genoma: predicción de CDS y múltiples elementos genómicos
- Anotación funcional con múltiples bases de datos generales (GO, KEGG, etc.) y bases de datos avanzadas (CARD, VFDB, etc.)
Conjunto Hi-C:
- Evaluación de biblioteca Hi-C.
- Contigs anclando agrupaciones, ordenando y orientando
- Evaluación del ensamblaje HI-C: basado en genoma de referencia y mapa de calor
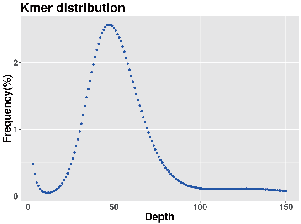
Encuesta del genoma: distribución de k-mer
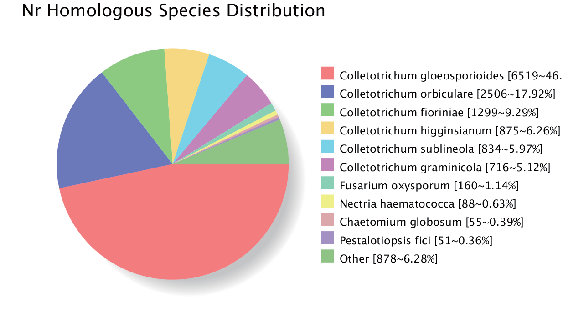
Ensamblaje del genoma: anotación de genes homólogos (base de datos NR)
Ensamblaje del genoma: anotación genética funcional (GO)
Explore los avances facilitados por los servicios de ensamblaje del genoma fúngico de BMKGene a través de una colección seleccionada de publicaciones.
Hao, J. y col. (2023) 'Perfil ómico integrado del hongo medicinal Inonotus obliquus en condiciones sumergidas',Genómica BMC, 24 (1), págs. 1-12. doi: 10.1186/S12864-023-09656-Z/FIGURES/3.
Lu, L. y col. (2023) 'La secuenciación del genoma revela la evolución y los mecanismos patogénicos del patógeno de la mancha ocular aguda del trigo Rhizoctonia cerealis',El diario de cultivos, 11 (2), págs. 405–416. doi: 10.1016/J.CJ.2022.07.024.
Zhang, H. y col. (2023) 'Recursos genómicos de cuatro especies de Clarireedia que provocan la mancha del dólar en diversos céspedes',Enfermedad de las plantas, 107 (3), págs. 929–934. doi: 10.1094/PDIS-08-22-1921-A
Zhang, SS y cols. (2023) 'Evidencia genética y molecular de un sistema de apareamiento tetrapolar en el hongo comestible Grifola frondosa',Revista de hongos, 9(10), pág. 959. doi: 10.3390/JOF9100959/S1.