Secuenciación de ARNm de longitud completa: nanoporos
Características
● Captura de ARNm poli-A seguida de síntesis de ADNc y preparación de bibliotecas.
● Secuenciación de las transcripciones completas.
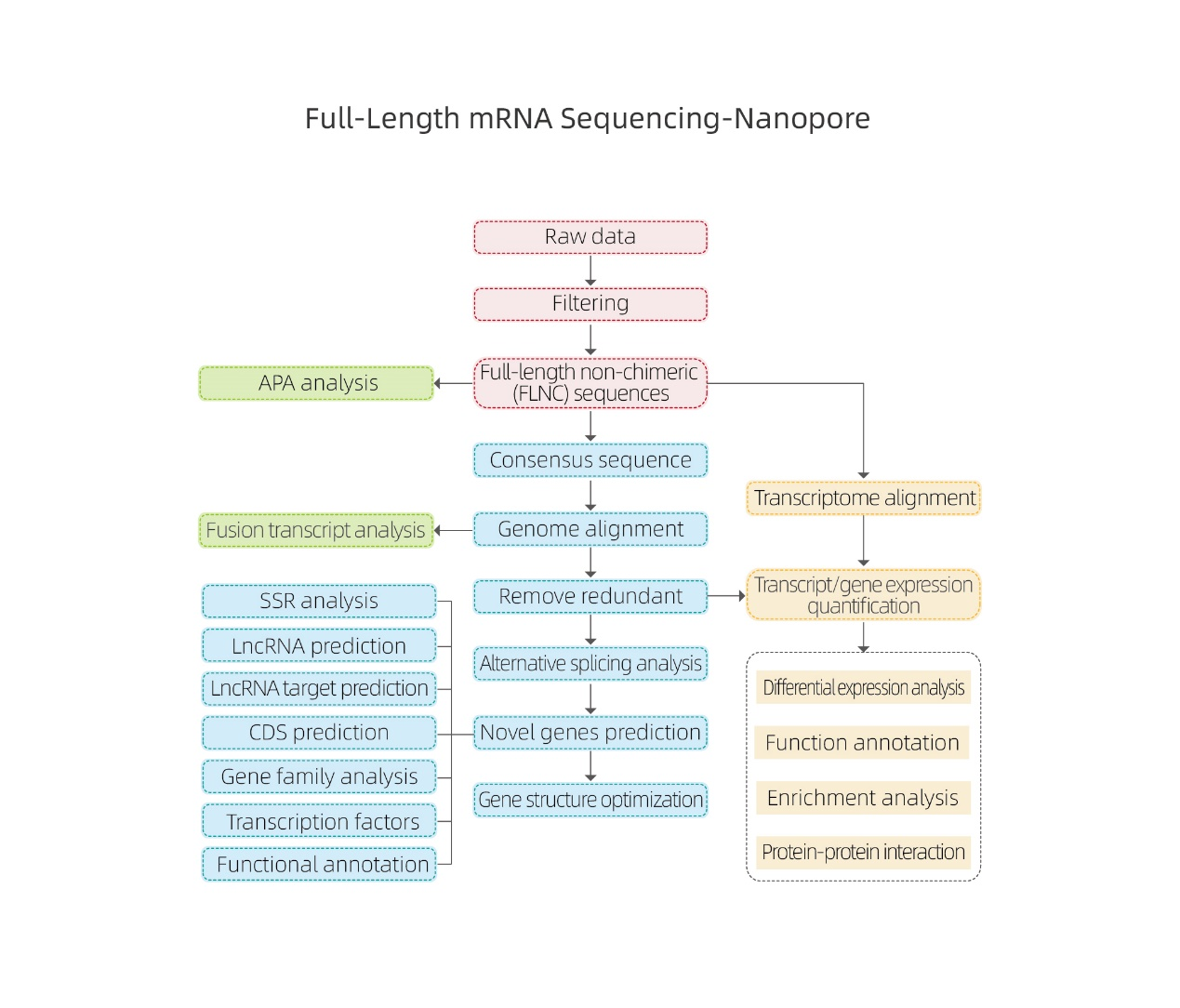
● Análisis bioinformático basado en el alineamiento con un genoma de referencia.
● El análisis bioinformático incluye no sólo la expresión a nivel de genes e isoformas, sino también el análisis de lncRNA, fusiones de genes, poliadenilación y estructura genética.
Ventajas del servicio
●Cuantificación de la expresión a nivel de isoformas.: permite un análisis de expresión detallado y preciso, revelando cambios que pueden quedar enmascarados al analizar la expresión genética completa
●Demandas de datos reducidas:En comparación con la secuenciación de próxima generación (NGS), la secuenciación de nanoporos presenta requisitos de datos más bajos, lo que permite niveles equivalentes de saturación de cuantificación de la expresión genética con datos más pequeños.
●Mayor precisión de la cuantificación de la expresión.: tanto a nivel genético como de isoforma
●Identificación de información transcriptómica adicional.: poliadenilación alternativa, genes de fusión y lcnRNA y sus genes diana
●Amplia experiencia: Nuestro equipo aporta una gran experiencia a cada proyecto, habiendo completado más de 850 proyectos de transcriptoma completo de Nanopore y procesado más de 8000 muestras.
●Soporte postventa: nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Requisitos de muestra y entrega
| Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| Poli A enriquecido | Iluminación PE150 | 6/12GB | Puntuación de calidad media: Q10 |
Requisitos de muestra:
Nucleótidos:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 100 | ≥ 1,0 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | Para plantas: RIN≥7,0; Para animales: RIN≥7,5; 5,0≥28S/18S≥1,0; elevación basal limitada o nula |
● Plantas:
Raíz, Tallo o Pétalo: 450 mg
Hoja o Semilla: 300 mg
Fruta: 1,2 gramos
● Animales:
Corazón o Intestino: 300 mg
Vísceras o Cerebro: 240 mg
Músculo: 450 mg
Huesos, Cabello o Piel: 1g
● Artrópodos:
Insectos: 6g
Crustáceos: 300 mg
● Sangre entera: 1 tubo
● Células: 106 células
Entrega de muestra recomendada
Envase: tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3; B1, B2, B3.
Envío:
1. Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
2. Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio
Nucleótidos:

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de datos

Servicios postventa
Flujo de trabajo del servicio
Tejido:

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de datos

Servicios postventa
● Procesamiento de datos sin procesar
● Identificación de transcripción
● Empalme alternativo
● Cuantificación de expresión a nivel de gen y nivel de isoforma.
● Análisis de expresión diferencial
● Anotación y enriquecimiento de funciones (DEG y DET)
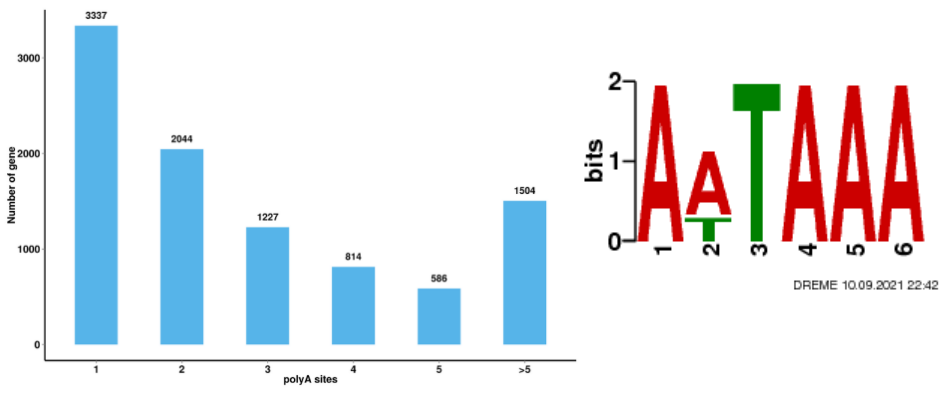
Análisis de empalme alternativo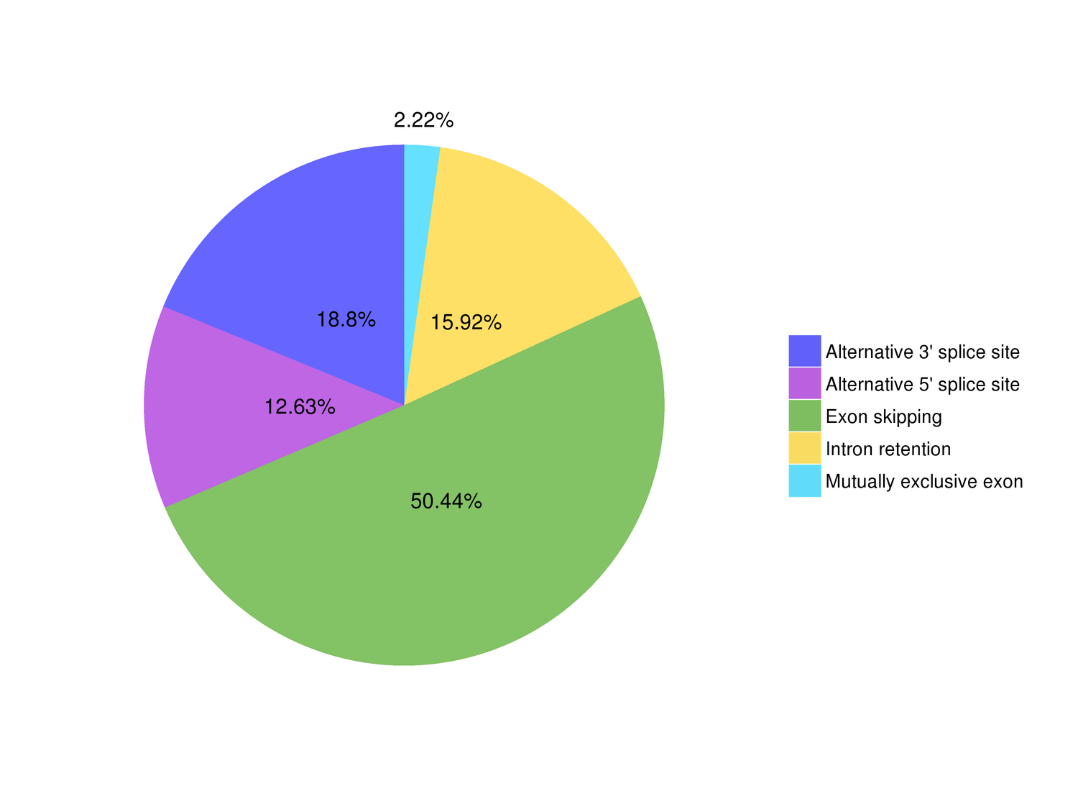 Análisis de poliadenilación alternativa (APA)
Análisis de poliadenilación alternativa (APA)
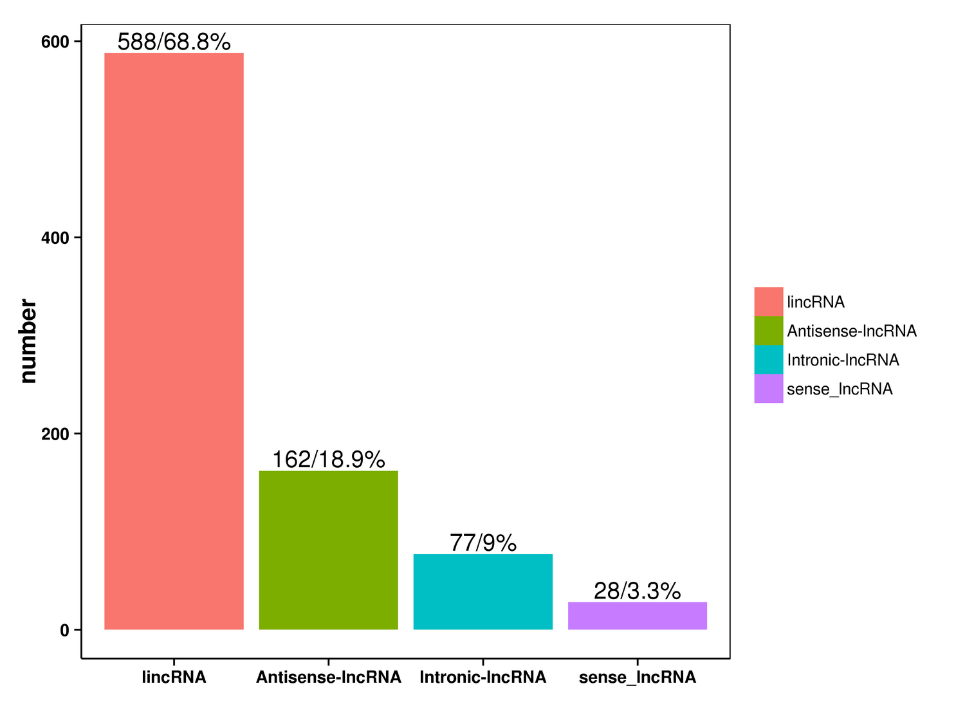
predicción de ARNc
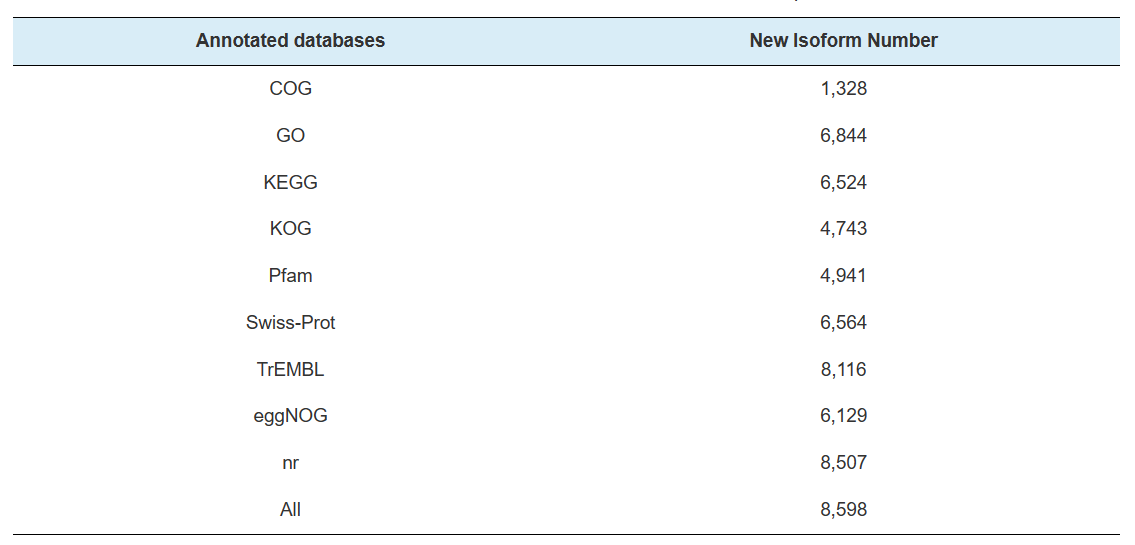
Anotación de genes nuevos.
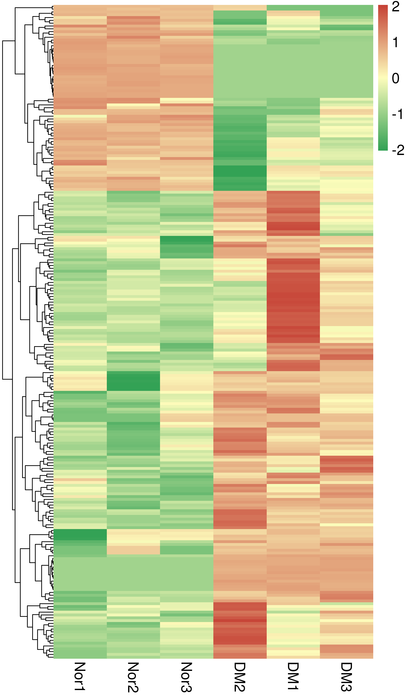
Agrupación de DET
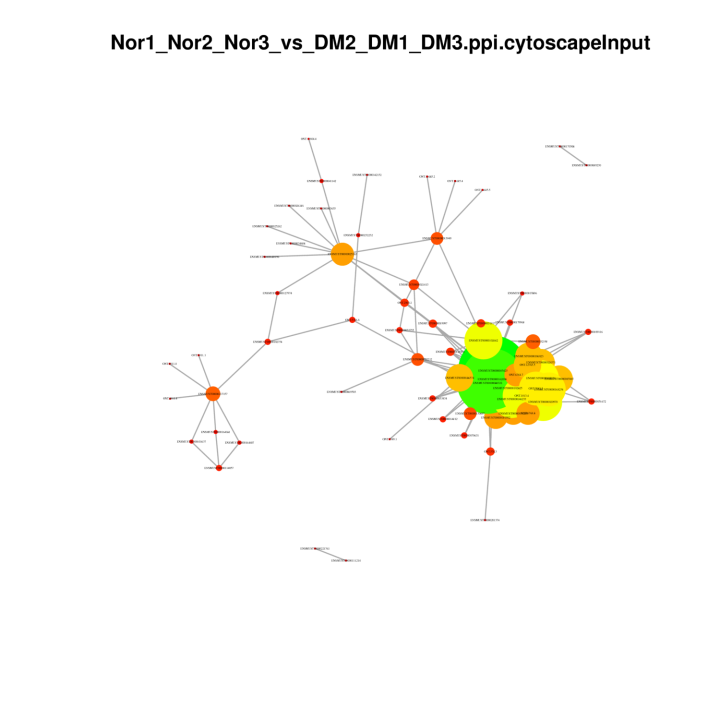
Redes proteína-proteína en DEG
Explore los avances facilitados por los servicios de secuenciación de ARNm completo Nanopore de BMKGene a través de una colección seleccionada de publicaciones.
Gong, B. y col. (2023) 'Activación epigenética y transcripcional de la quinasa secretora FAM20C como oncogén en glioma', Journal of Genetics and Genomics, 50 (6), págs. doi: 10.1016/J.JGG.2023.01.008.
Él, Z. et al. (2023) 'La secuenciación completa del transcriptoma de linfocitos que responden al IFN-γ revela una respuesta inmune sesgada por Th1 en la platija (Paralichthys olivaceus)', Fish & Shellfish Immunology, 134, pág. 108636. doi: 10.1016/J.FSI.2023.108636.
Ma, Y. et al. (2023) 'Análisis comparativo de los métodos de secuenciación de ARN de PacBio y ONT para la identificación del veneno de Nemopilema Nomurai', Genomics, 115(6), pág. 110709. doi: 10.1016/J.YGENO.2023.110709.
Yu, D. y col. (2023) 'El análisis Nano-seq revela una tendencia funcional diferente entre exosomas y microvesículas derivadas de hUMSC', Stem Cell Research and Therapy, 14 (1), págs. doi: 10.1186/S13287-023-03491-5/TABLAS/6.