Transcriptoma espacial BMKMANU S1000
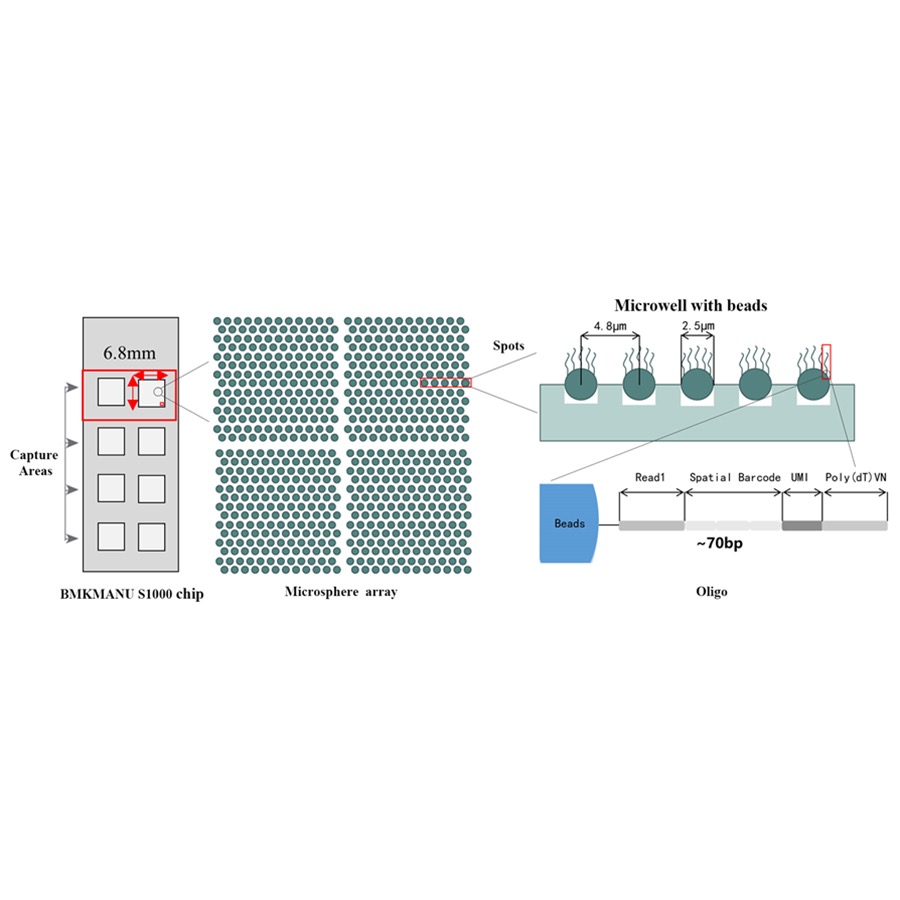
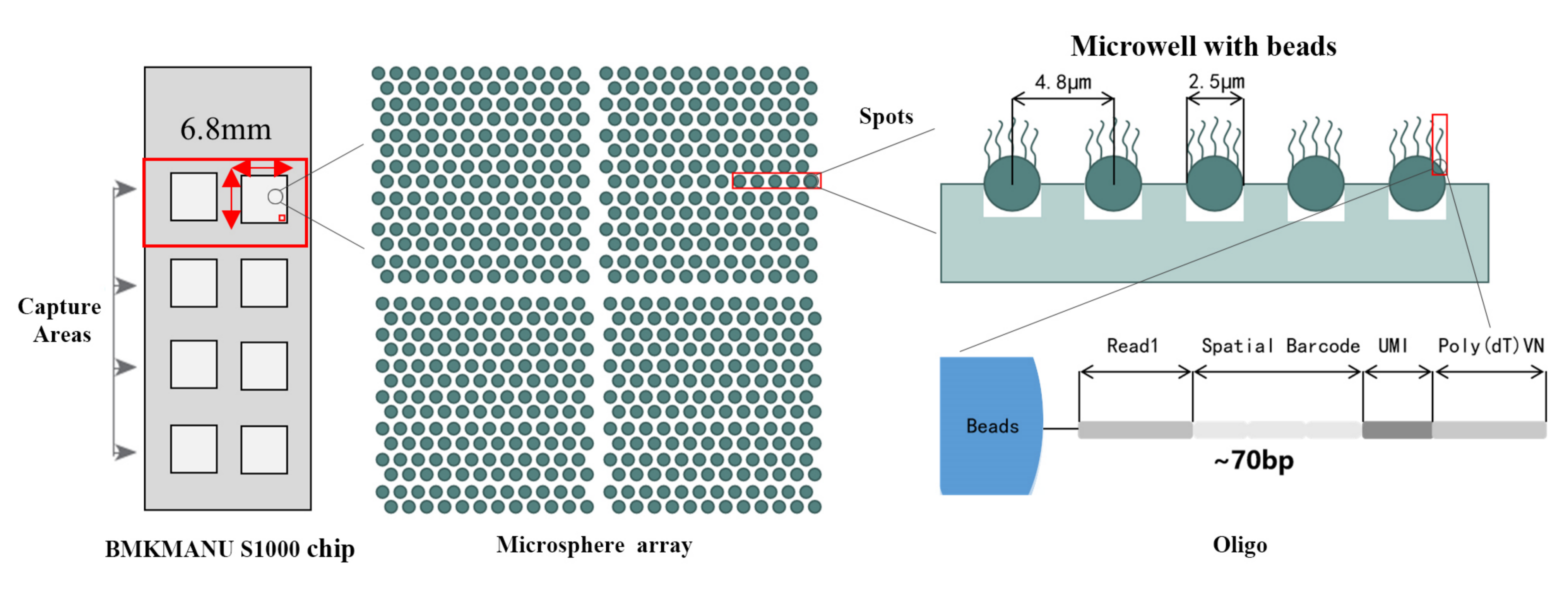
Esquema técnico del transcriptoma espacial BMKMANU S1000
Características
● Resolución: 5 µM
● Diámetro del punto: 2,5 µM
● Número de anuncios: aproximadamente 2 millones
● 3 formatos de área de captura posibles: 6,8 mm * 6,8 mm, 11 mm * 11 mm o 15 mm * 20 mm
● Cada cuenta con código de barras está cargada con cebadores compuestos de 4 secciones:
cola poli(dT) para cebado de ARNm y síntesis de ADNc
Identificador molecular único (UMI) para corregir el sesgo de amplificación
código de barras espacial
Secuencia de unión del cebador de secuenciación de lectura parcial 1
● H&E y tinción fluorescente de secciones
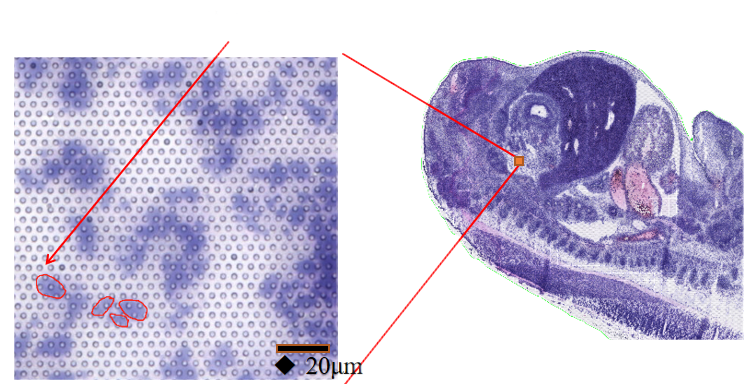
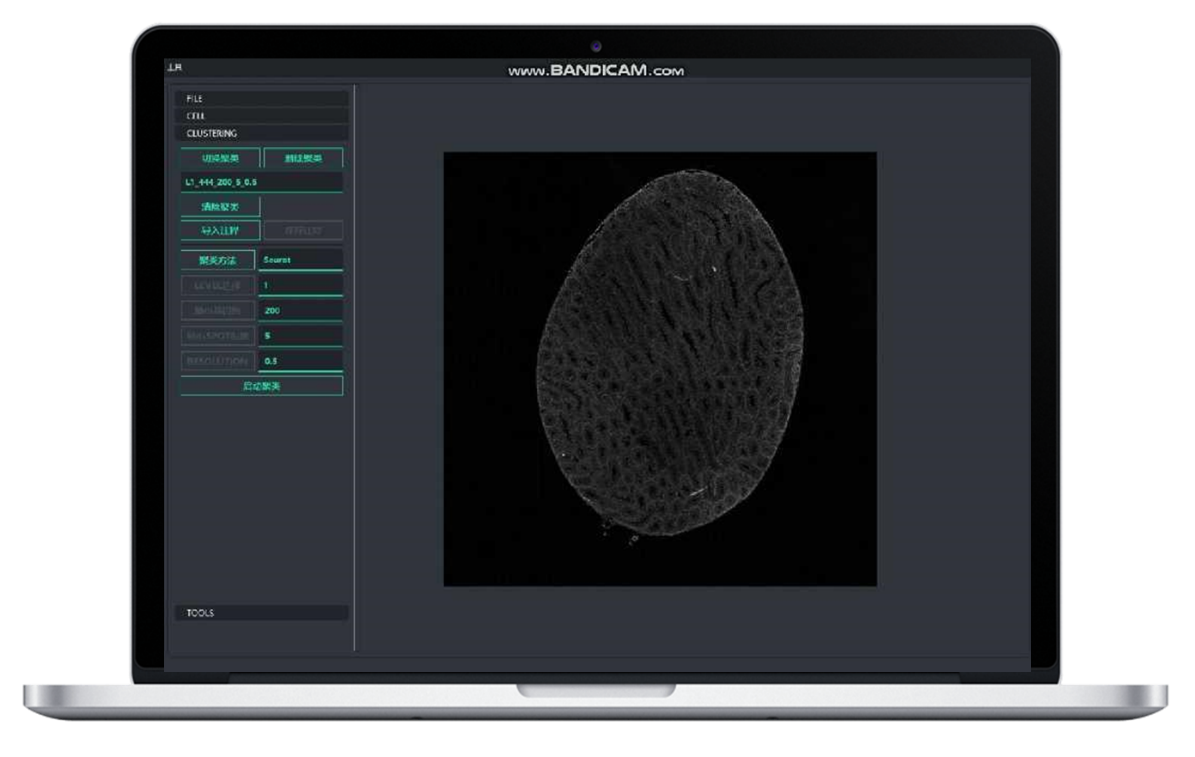
● Posibilidad de utilizartecnología de segmentación celular: integración de tinción H&E, tinción fluorescente y secuenciación de ARN para determinar los límites de cada célula y asignar correctamente la expresión genética a cada célula.
Ventajas de BMKMANU S1000
●Resolución subcelular: Cada área de captura contenía >2 millones de puntos espaciales con códigos de barras con un diámetro de 2,5 µm y un espacio de 5 µm entre los centros de los puntos, lo que permite el análisis del transcriptoma espacial con resolución subcelular (5 µm).
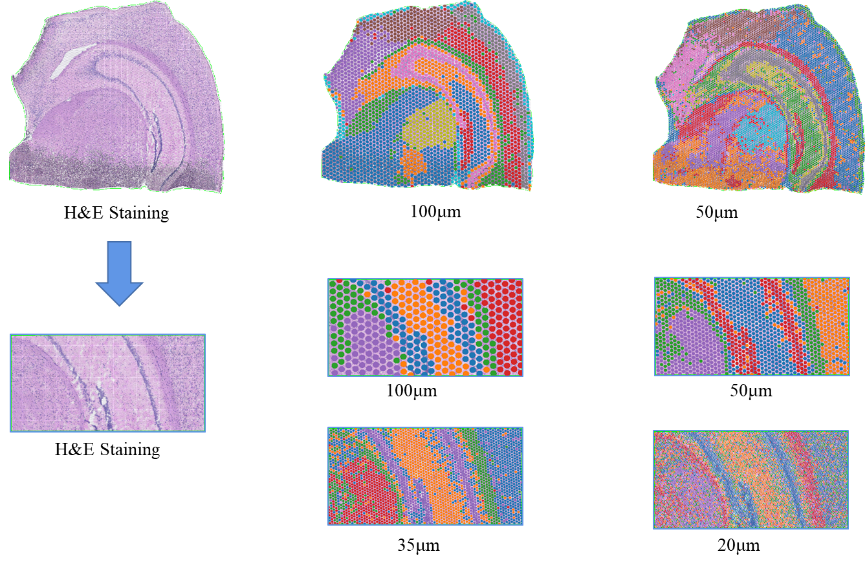
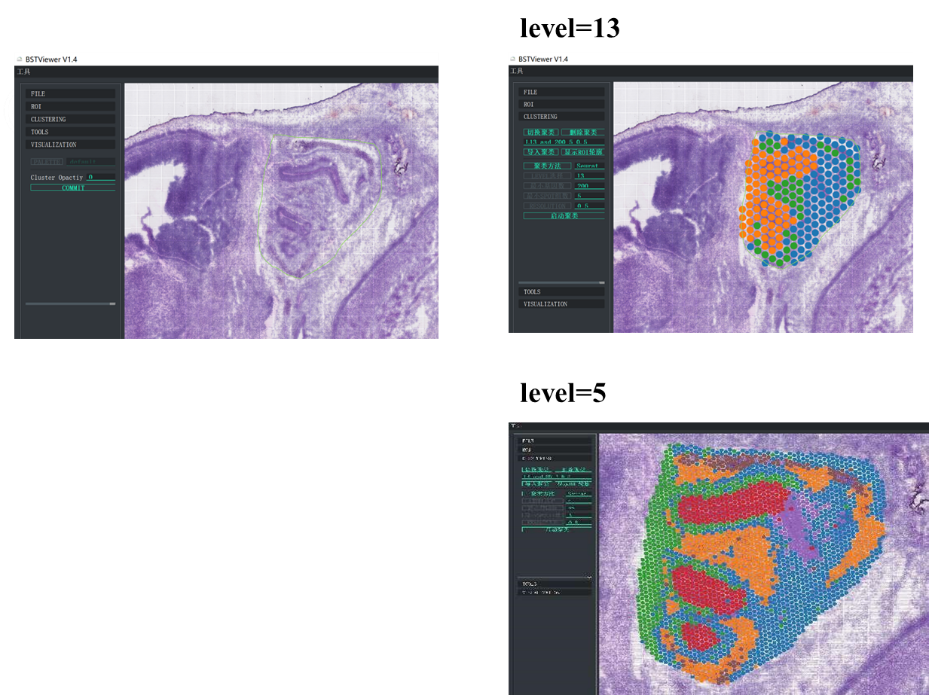
●Análisis de resolución multinivel:Análisis flexible de niveles múltiples que van desde 100 μm a 5 μm para resolver diversas características del tejido con una resolución óptima.
●Posibilidad de utilizar la tecnología de segmentación celular “tres en una diapositiva”:Al combinar tinción de fluorescencia, tinción H&E y secuenciación de ARN en un solo portaobjetos, nuestro algoritmo de análisis "tres en uno" permite la identificación de límites celulares para la transcriptómica celular posterior.
●Compatible con múltiples plataformas de secuenciación: Tanto NGS como secuenciación de lectura larga están disponibles.
●Diseño flexible de 1 a 8 áreas de captura activa: El tamaño del área de captura es flexible, siendo posible utilizar 3 formatos (6,8 mm * 6,8 mm., 11 mm * 11 mm y 15 mm * 20 mm)
●Servicio integral: Integra todos los pasos basados en la experiencia y las habilidades, incluida la criosección, la tinción, la optimización de tejidos, los códigos de barras espaciales, la preparación de bibliotecas, la secuenciación y la bioinformática.
●Bioinformática Integral y Visualización de Resultados Fácil de Usar:El paquete incluye 29 análisis y más de 100 cifras de alta calidad, combinados con el uso de software desarrollado internamente para visualizar y personalizar la división celular y la agrupación puntual.
●Análisis y visualización de datos personalizados: disponible para diferentes solicitudes de investigación
●Equipo técnico altamente calificado: con experiencia en más de 250 tipos de tejidos y más de 100 especies, incluidos humanos, ratones, mamíferos, peces y plantas.
●Actualizaciones en tiempo real de todo el proyecto: con control total del progreso experimental.
●Análisis conjunto opcional con secuenciación de ARNm unicelular
Especificaciones de servicio
|
Muestra Requisitos
| Biblioteca |
Estrategia de secuenciación
| Datos recomendados | Control de calidad |
| Muestras criogénicas integradas en OCT, 3 bloques por muestra | Biblioteca de ADNc S1000 | Illumina PE150 (otras plataformas disponibles) | 100.000 lecturas de PE por 100 uM (60-150 GB) | RIN>7 |
Para obtener más detalles sobre la guía de preparación de muestras y el flujo de trabajo del servicio, no dude en hablar con unExperto en BMKGENE
Flujo de trabajo del servicio
En la fase de preparación de la muestra, se realiza una prueba inicial de extracción de ARN en masa para garantizar que se pueda obtener ARN de alta calidad. En la etapa de optimización del tejido, las secciones se tiñen y visualizan y se optimizan las condiciones de permeabilización para la liberación de ARNm del tejido. Luego, el protocolo optimizado se aplica durante la construcción de la biblioteca, seguido de la secuenciación y el análisis de datos.
El flujo de trabajo completo del servicio implica actualizaciones en tiempo real y confirmaciones del cliente para mantener un ciclo de retroalimentación receptivo, lo que garantiza una ejecución fluida del proyecto.
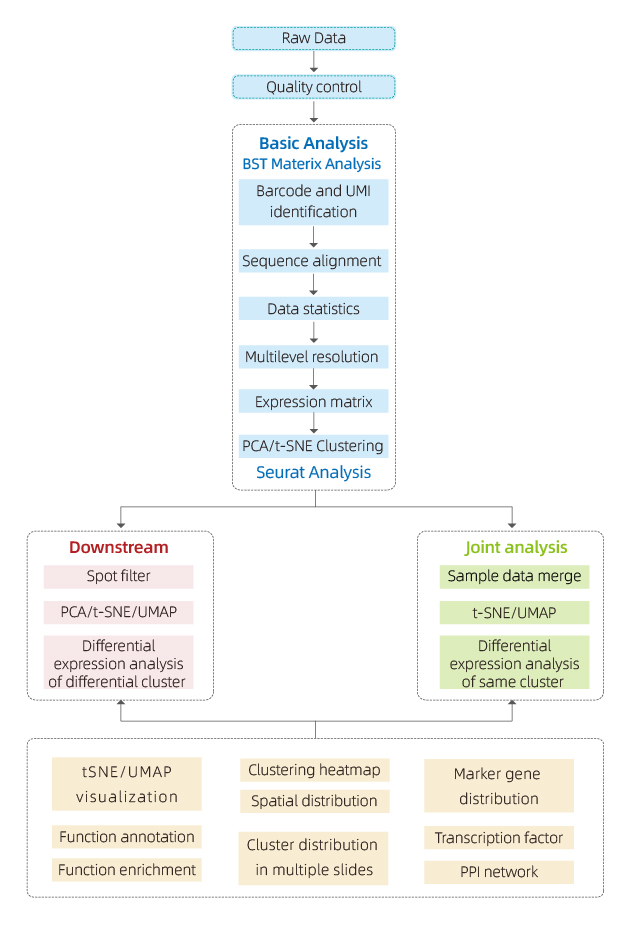
Los datos generados por BMKMANU S1000 se analizan utilizando el software "BSTMatrix", diseñado de forma independiente por BMKGENE, generando una matriz de expresión genética. A partir de ahí, se genera un informe estándar que incluye control de calidad de los datos, análisis de muestras internas y análisis entre grupos.
● Control de calidad de datos:
Salida de datos y distribución de puntuación de calidad.
Detección de genes por punto
Cobertura de tejido
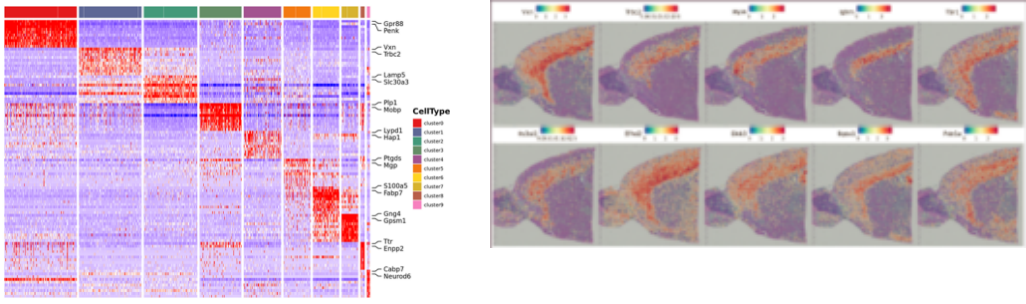
● Análisis de muestra interna:
Riqueza genética
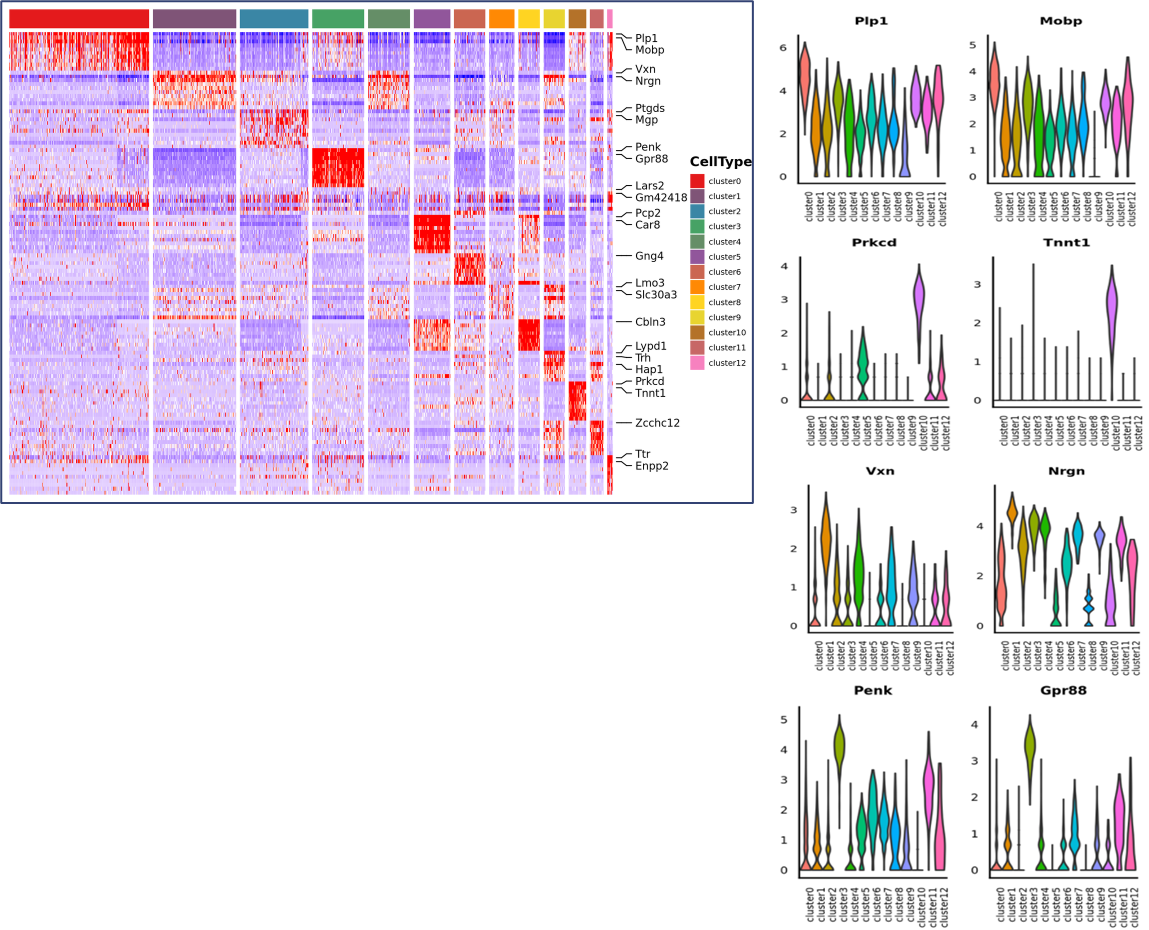
Agrupación puntual, incluido el análisis de dimensiones reducidas
Análisis de expresión diferencial entre clusters: identificación de genes marcadores.
Anotación funcional y enriquecimiento de genes marcadores.
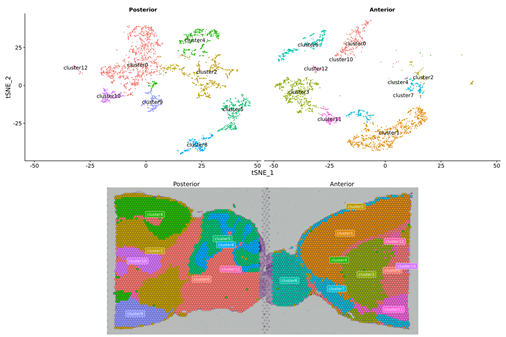
● Análisis intergrupal:
Recombinación de manchas de ambas muestras (p. ej., enferma y control) y reagrupación
Identificación de genes marcadores para cada grupo.
Anotación funcional y enriquecimiento de genes marcadores.
Expresión diferencial del mismo cluster entre grupos
Además, BMKGENE desarrolló "BSTViewer", una herramienta fácil de usar que permite al usuario visualizar la expresión genética y detectar agrupaciones en diferentes resoluciones.
BMKGene desarrolló un software para una visualización fácil de usar
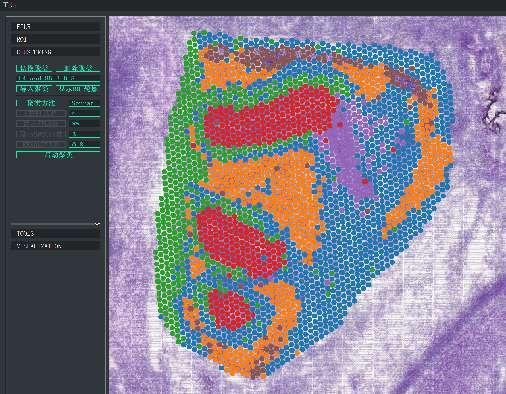
Agrupación de puntos de BSTViewer con resolución multinivel
BSTCellViewer: división celular automática y manual
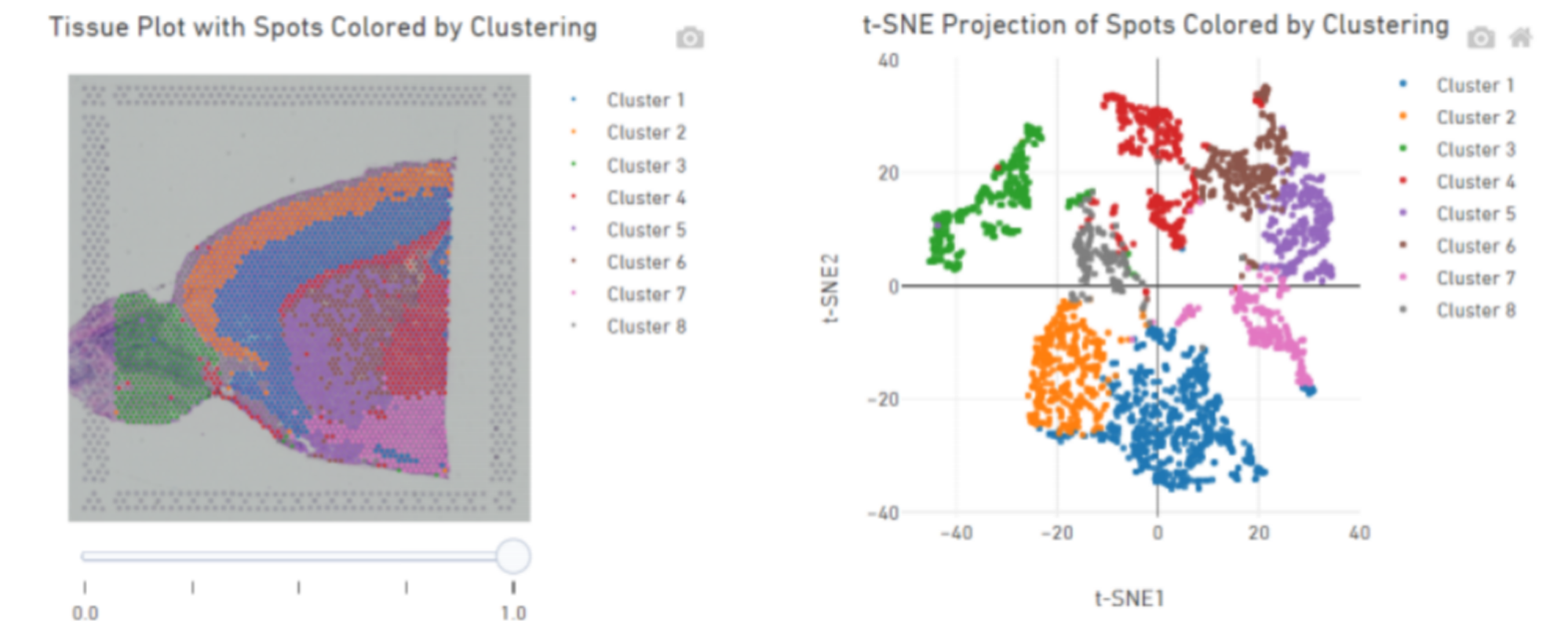
Análisis de muestra interna
Agrupación puntual:
Identificación de genes marcadores y distribución espacial:
Análisis intergrupal
Combinación de datos de ambos grupos y reagrupación:
Genes marcadores de nuevos grupos:
Explore los avances facilitados por los servicios de transcriptómica espacial de BMKGene con la tecnología BMKManu S1000 en esta publicación destacada:
Canción, X. et al. (2023) 'La transcriptómica espacial revela células de clorénquima inducidas por la luz implicadas en la promoción de la regeneración de brotes en callos de tomate',Actas de la Academia Nacional de Ciencias de los Estados Unidos de América, 120(38), pág. e2310163120. doi: 10.1073/pnas.2310163120
Usted, Y. et al. (2023) 'Comparación sistemática de métodos transcriptómicos espaciales basados en secuenciación',bioRxiv, pag. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.