Transcriptoma espacial de Visium de genómica 10x
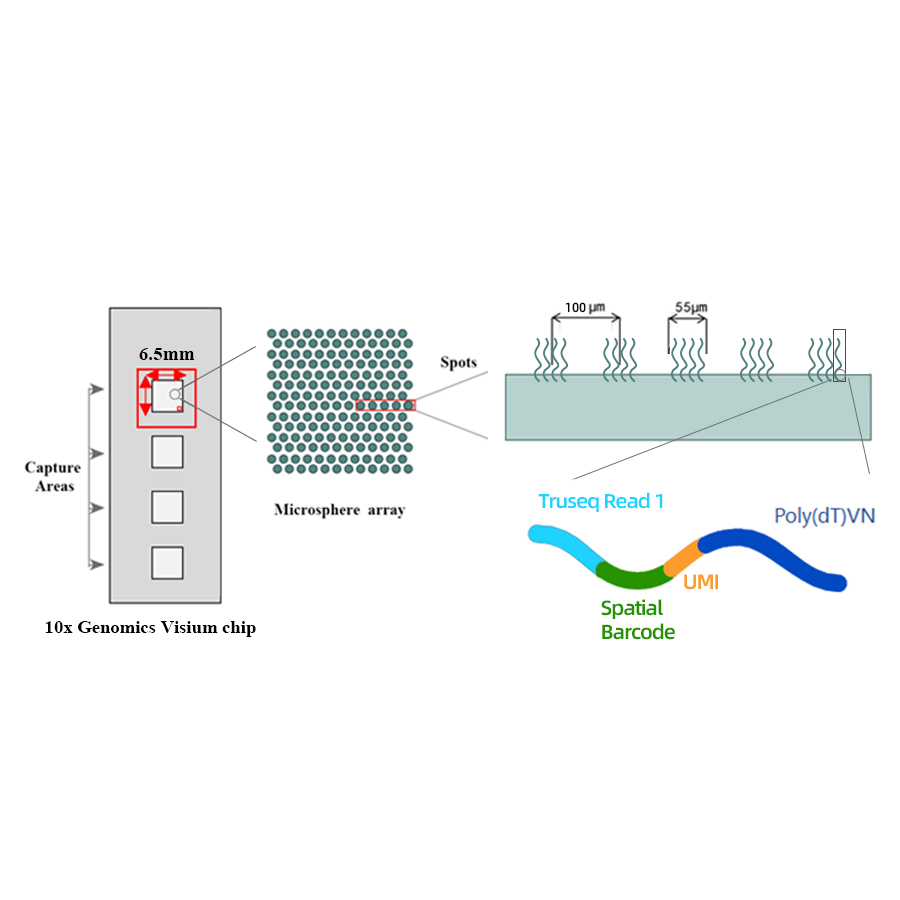
Esquema técnico

Características
● Resolución: 100 µm
● Diámetro de la mancha: 55 µm
● Número de puntos: 4992
● Área de captura: 6.5 x 6.5 mm
● Cada punto código de barras está cargado con cebadores compuestos por 4 secciones:
- cola de poli (DT) para cebado de ARNm y síntesis de ADNc
- Identificador molecular único (UMI) para corregir el sesgo de amplificación
- Código de barras espacial
- secuencia de unión de la votación de secuenciación de lectura parcial 1
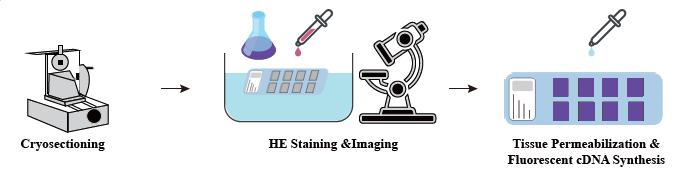
● Tinción de secciones H&E
Ventajas
●Servicio único: Integra toda la experiencia y los pasos basados en habilidades, incluida la crioconción, tinción, optimización de tejidos, código de barras espacial, preparación de la biblioteca, secuenciación y bioinformática.
● Equipo técnico altamente calificado: Con experiencia en más de 250 tipos de tejidos y más de 100 especies, incluidos humanos, ratones, mamíferos, peces y plantas.
●Actualización en tiempo real de todo el proyecto: con el control total del progreso experimental.
●Bioinformática estándar integral:El paquete incluye 29 análisis y más de 100 cifras de alta calidad.
●Análisis y visualización de datos personalizados: Disponible para diferentes solicitudes de investigación.
●Análisis conjunto opcional con secuenciación de ARNm de células individuales
Presupuesto
| Requisitos de muestra | Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| Muestras de crio embebido en octubre (Diámetro óptimo: aprox. 6x6x6 mm³) 2 bloques por muestra | Biblioteca de ADNc de 10x Visium | Illumina PE150 | 50k PE lecturas por lugar (60 GB) | Rin> 7 |
Para obtener más detalles sobre la orientación de preparación de muestras y el flujo de trabajo de servicio, no dude en hablar con unExperto en bmkgene
Flujo de trabajo de servicio
En la fase de preparación de la muestra, se realiza una prueba de extracción de ARN a granel inicial para garantizar que se pueda obtener un ARN de alta calidad. En la etapa de optimización del tejido, las secciones se tiñen y visualizan y las condiciones de permeabilización para la liberación de ARNm del tejido están optimizadas. El protocolo optimizado se aplica luego durante la construcción de la biblioteca, seguido de secuenciación y análisis de datos.
El flujo de trabajo de servicio completo implica actualizaciones en tiempo real y confirmaciones del cliente para mantener un ciclo de retroalimentación receptivo, asegurando la ejecución sin problemas del proyecto.

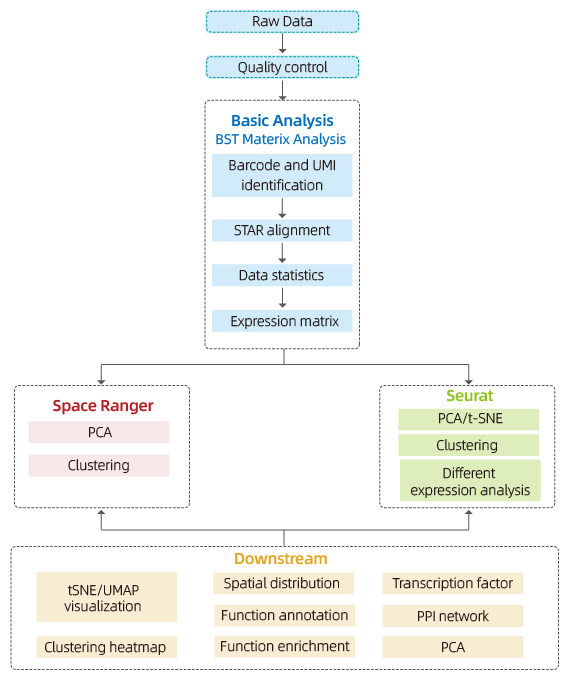
Incluye el siguiente análisis:
Control de calidad de datos:
o Distribución de salida de datos y puntaje de calidad
o Detección de genes por lugar
o cobertura de tejido
Análisis de muestra interna:
o Riqueza de genes
o Agrupación de manchas, incluido el análisis de dimensión reducida
o Análisis de expresión diferencial entre grupos: identificación de genes marcadores
o Anotación funcional y enriquecimiento de genes marcadores
Análisis entre grupos
o Re-combinación de puntos de ambas muestras (por ejemplo, enfermo y control) y reubique
o Identificación de genes marcadores para cada clúster
o Anotación funcional y enriquecimiento de genes marcadores
o Expresión diferencial del mismo clúster entre grupos
Análisis de muestra interna
Clustering spot

Identificación de genes marcadores y distribución espacial


Análisis entre grupos
Combinación de datos de ambos grupos y reubique
Genes marcadores de nuevos grupos
Explore los avances facilitados por el servicio de transcriptómica espacial de Bmkgene por 10x Visium en estas publicaciones destacadas:
Chen, D. et al. (2023) 'MTHL1, un potencial homólogo de Drosophila de GPCR de adhesión de mamíferos, está involucrado en reacciones antitumorales a las células oncogénicas inyectadas en las moscas',Actas de la Academia Nacional de Ciencias de los Estados Unidos de América, 120 (30), p. E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'El acero permite la delineación de alta resolución de los datos transcriptómicos espacio-temporales',Informes en bioinformática, 24 (2), págs. 1-10. doi: 10.1093/bib/bbad068.
Liu, C. et al. (2022) "Un atlas espacio -temporal de la organogénesis en el desarrollo de flores de orquídeas",Investigación de ácidos nucleicos, 50 (17), pp. 9724–9737. doi: 10.1093/nar/gkac773.
Wang, J. et al. (2023) 'La integración de la transcriptómica espacial y la secuenciación de ARN de un solo núcleo revela las estrategias terapéuticas potenciales para el leiomioma uterino',Revista Internacional de Ciencias Biológicas, 19 (8), pp. 2515–2530. doi: 10.7150/ijbs.83510.