Homa Tuta Ekzoma Sekvencado
Servaj Trajtoj
● Du ekzome-paneloj haveblaj surbaze de cela riĉiĝo per enketoj: Sure Select Human All Exon v6 (Agilent) kaj xGen Exome Hybridization Panel v2 (IDT).
● Sekvencado sur Illumina NovaSeq.
● Bioinformatika dukto direktita al analizo de malsano aŭ analizo de tumoroj.
Servaj Avantaĝoj
●Celoj Protein Kodiga Regiono: Kaptante kaj sekvencante proteinajn kodigajn regionojn, hWES estas utiligita por riveli variaĵojn ligitajn al proteinstrukturo.
●Kostefika:hWES donas ĉirkaŭ 85% de la homaj malsan-rilataj mutacioj de 1% de la homa genaro.
●Alta Precizeco: Kun alta sinsekva profundo, hWES faciligas la detekton de kaj oftaj variantoj kaj maloftaj variantoj kun frekvencoj pli malaltaj ol 1%.
●Rigora Kvalita Kontrolo: Ni efektivigas kvin kernajn kontrolpunktojn tra ĉiuj stadioj, de specimena kaj biblioteka preparo ĝis sekvencado kaj bioinformadiko. Ĉi tiu zorgema monitorado certigas la liveron de konstante altkvalitaj rezultoj.
●Ampleksa Bioinformatika Analizo: nia dukto iras preter identigado de varioj al la referenca genaro, ĉar ĝi enkorpigas altnivelan analizon dizajnitan por specife trakti esplordemandojn ligitajn al genetikaj aspektoj de malsanoj aŭ tumoranalizo.
●Postvenda Subteno:Nia devontigo etendiĝas preter la kompletiĝo de la projekto kun 3-monata post-venda servoperiodo. Dum ĉi tiu tempo, ni ofertas projektan sekvadon, helpon pri solvo de problemoj kaj sesiojn pri demandoj kaj respondoj por trakti demandojn rilate al la rezultoj.
Specimenaj Specifoj
| Exon-Kapto-Strategio | Sekvenca Strategio | Rekomendita Datuma Eligo |
| Certe Elektu Human All Exon v6 (Agilent) aŭ xGen Exome Hybridization Panel v2 (IDT)
| Illumina NovaSeq PE150 | 5 -10 Gb Por mendeliaj malordoj/maloftaj malsanoj: > 50x Por tumoraj specimenoj: ≥ 100x |
Ekzemplaj Postuloj
| Specimena Tipo
| Kvanto(Qubit®)
| Koncentriĝo | Volumo
| Pureco (NanoDrop™) |
|
Genoma DNA
| ≥ 50 ng | ≥ 6 ng/μL | ≥ 15 μL | OD260/280=1.8-2.0 neniu degradado, neniu poluado
|
Bioinformadiko

Bioinformatika analizo de hWES-malsana provaĵoj inkluzivas:
● Sekvencaj datumoj QC
● Referenca Genoma Alignigo
● Identigo de SNPs kaj InDels
● Funkcia Komentario de SNPs kaj InDels

Bioinformatika analizo de tumorprovaĵoj inkluzivas:
● Sekvencaj datumoj QC
● Referenca Genoma Alignigo
● Identigo de SNP-oj, InDels kaj somataj variadoj
● Identigo de ĝermliniaj variantoj
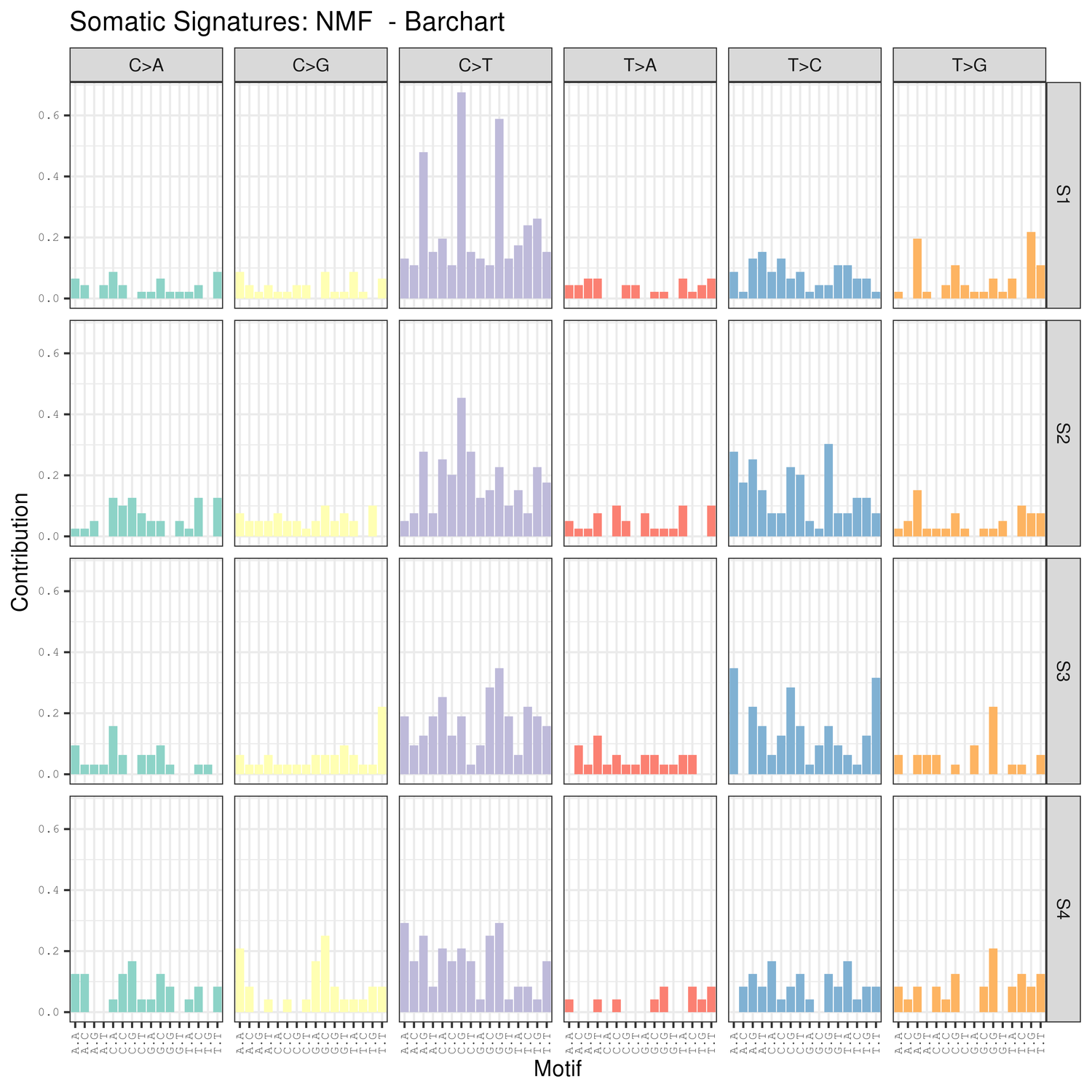
● Analizo de mutaciaj subskriboj
● Identigo de stiraj genoj surbaze de gajno-de-funkciaj mutacioj
● Mutacia komentario je la nivelo de drog-malsaniĝemo
● Analizo de heterogeneco - kalkulo de pureco kaj ploidio
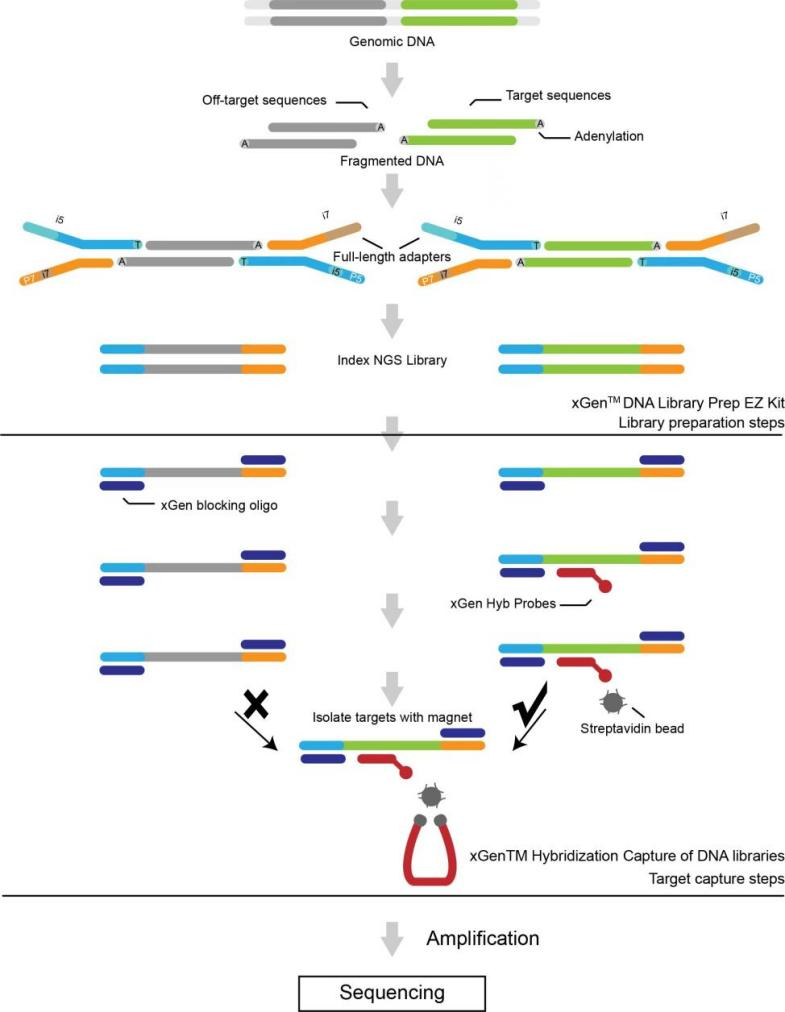
Serva Laborfluo

Specimena livero

DNA-eltiro

Biblioteka konstruado

Sekvencado

Analizo de datumoj

Transdono de datumoj

Post-vendaj servoj
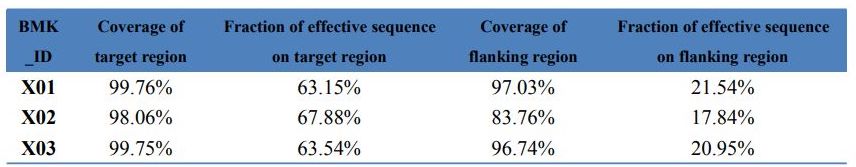
Datumoj QC - Statistiko de Exome-kapto
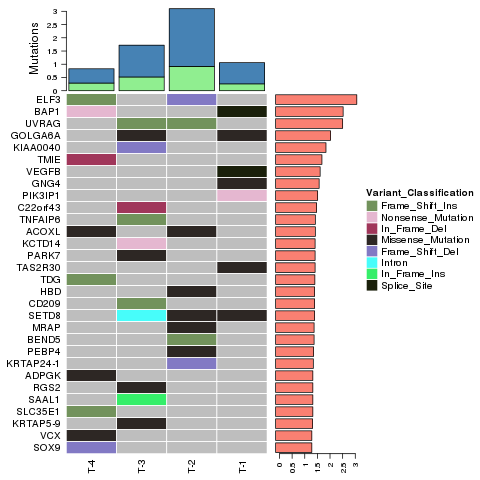
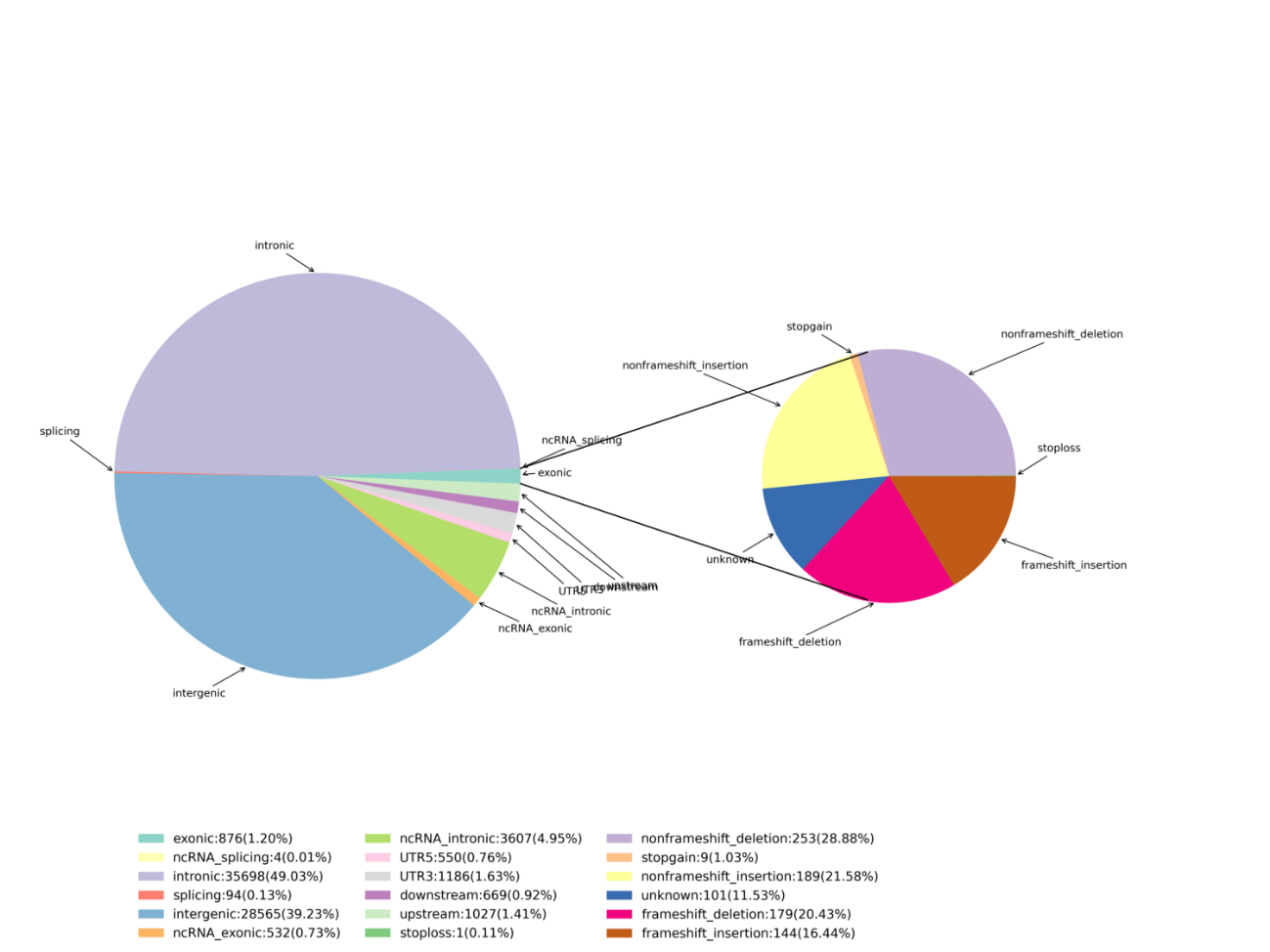
Variantidentigo - InDels
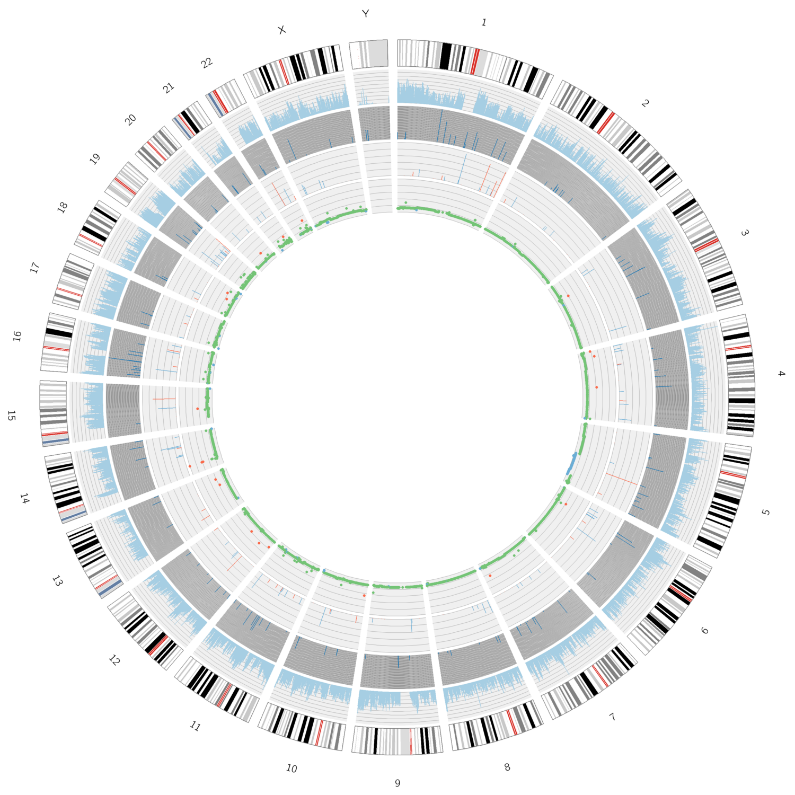
Altnivela analizo: identigo kaj distribuado de malutilaj SNPoj/InDels - Circos-intrigo
Tumoranalizo: identigo kaj distribuado de somataj mutacioj - Circos-intrigo
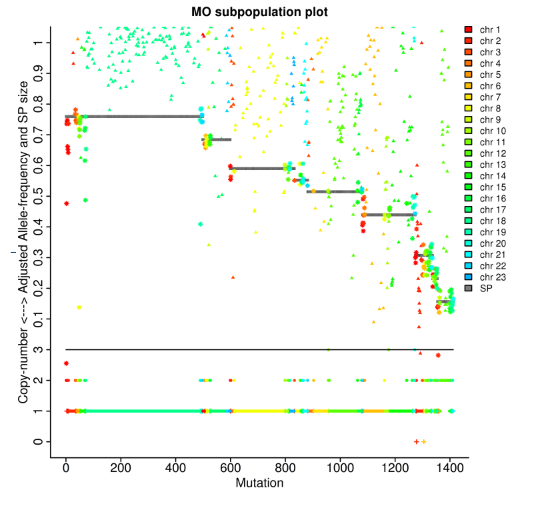
Tumoranalizo: klonaj genlinioj