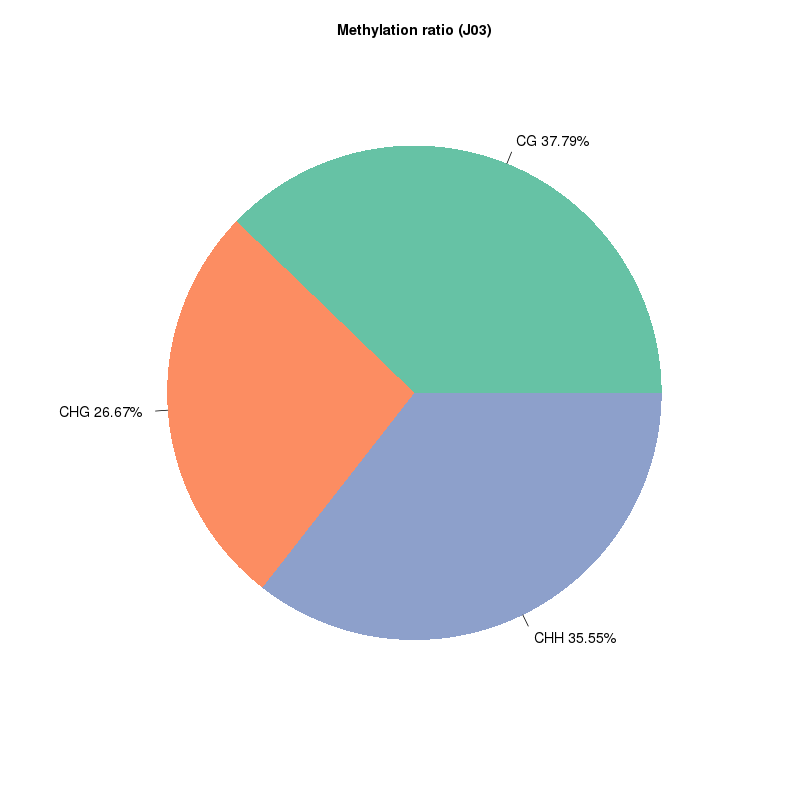Ganzes Genom Bisul -Fixe -Sequenzierung (WGBS)
Servicefunktionen
● Benötigt ein Referenzgenom.
● Die Lambda -DNA wird hinzugefügt, um die Effizienz der Bisulfite -Umwandlung zu überwachen.
● Sequenzierung auf Illumina Novaseq.
Servicevorteile
●Goldstandard für die DNA -Methylierungsforschung: Diese reife Methylierungsumwandlungsverarbeitungstechnologie hat eine hohe Genauigkeit und eine gute Reproduzierbarkeit.
●Breite Abdeckung und Einzelbasisauflösung:Nachweis von Methylierungsstellen auf genomweiter Ebene.
●Komplette Plattform:Bieten Sie einen One-Stop-hervorragenden Service von Probenverarbeitung, Bibliothekskonstruktion, Sequenzierung bis hin zur Bioinformatikanalyse.
●Umfangreiches Fachwissen: Mit WGBS-Sequenzierungsprojekten, die in einer Vielzahl von Arten erfolgreich abgeschlossen sind, bringt BMKgene über ein Jahrzehnt Erfahrung, ein hochqualifiziertes Analyseteam, umfassende Inhalte und exzellente Unterstützung nach dem Verkauf.
●Möglichkeit, sich der Transkriptomik -Analyse anzuschließen: Ermöglichen Sie die integrierte Analyse von WGBs mit anderen OMICS-Daten wie RNA-Seq.
Beispielspezifikationen
| Bibliothek | Sequenzierungsstrategie | Empfohlene Datenausgabe | Qualitätskontrolle |
| Bisulfit behandelt | Illumina PE150 | 30x Tiefe | Q30 ≥ 85% Bisulfitumwandlung> 99% |
Stichprobenanforderungen
| Konzentration (ng/µl) | Gesamtmenge (µg) | Zusätzliche Anforderungen | |
| Genomische DNA | ≥ 5 | ≥ 400 ng | Begrenzte Verschlechterung oder Kontamination |
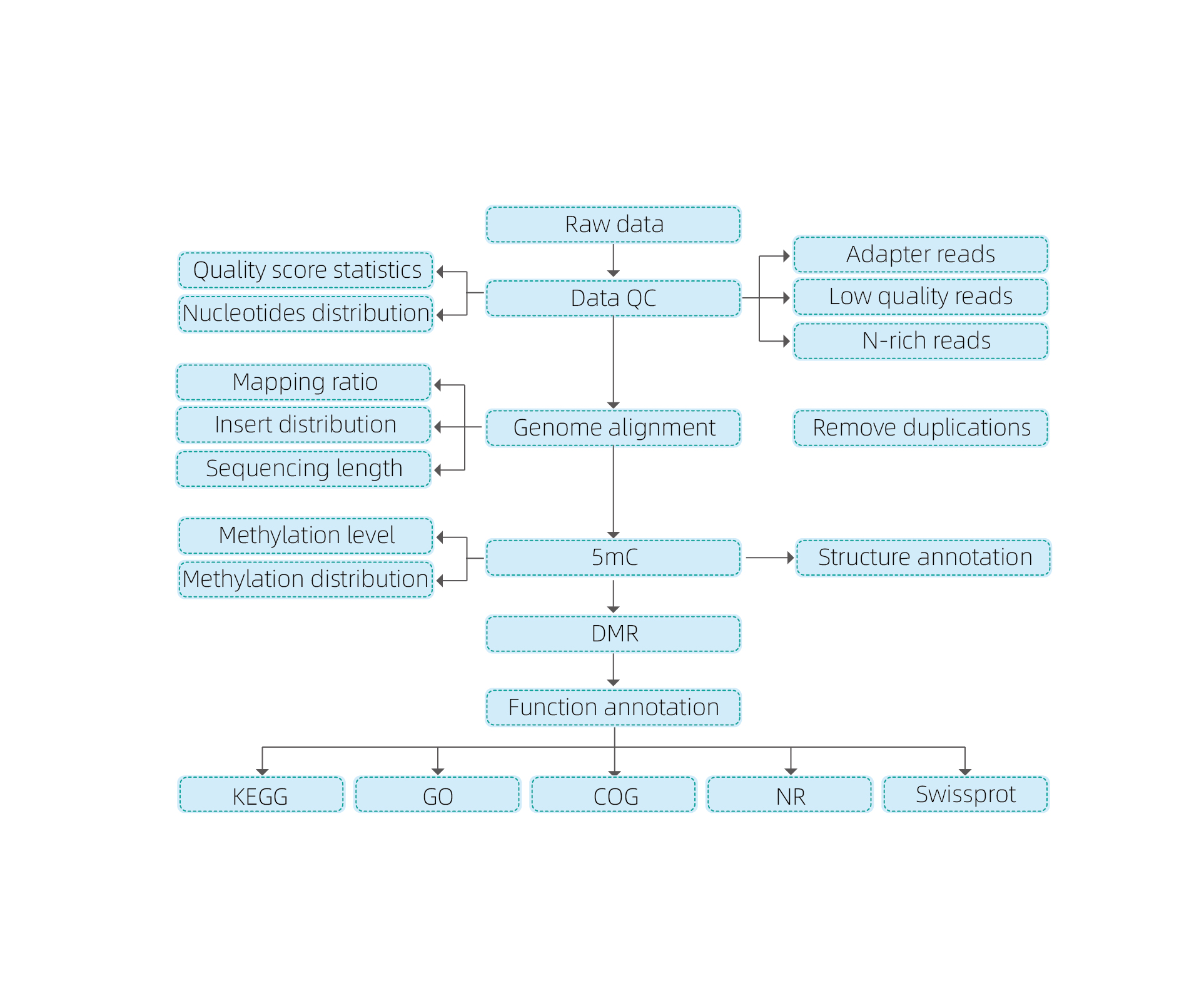
Service Work Flow

Probenabgabe

DNA -Extraktion

Bibliothekskonstruktion

Sequenzierung

Datenanalyse

Datenerbringung
Enthält die folgende Analyse:
● RAW -Sequenzierungsqualitätskontrolle;
● Mapping auf Referenzgenom;
● Nachweis von 5mc methylierten Basen;
● Analyse der Methylierungsverteilung und Anmerkung;
● Analyse differentiell methylierter Regionen (DMRs);
● Funktionelle Annotation von Genen, die mit DMRs verbunden sind.
5mc Methylierungserkennung: Arten von methylierten Stellen
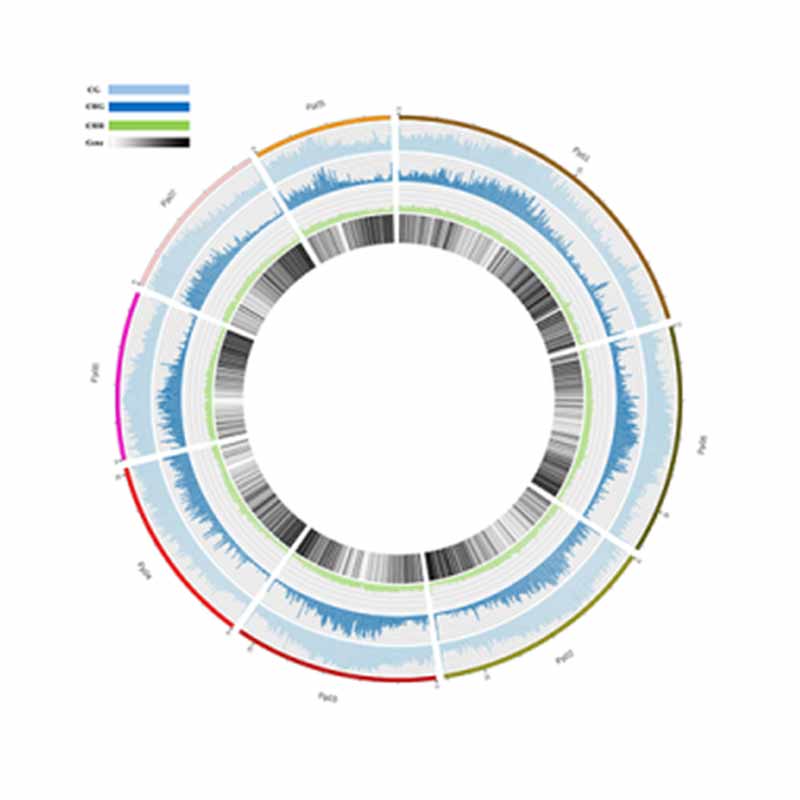
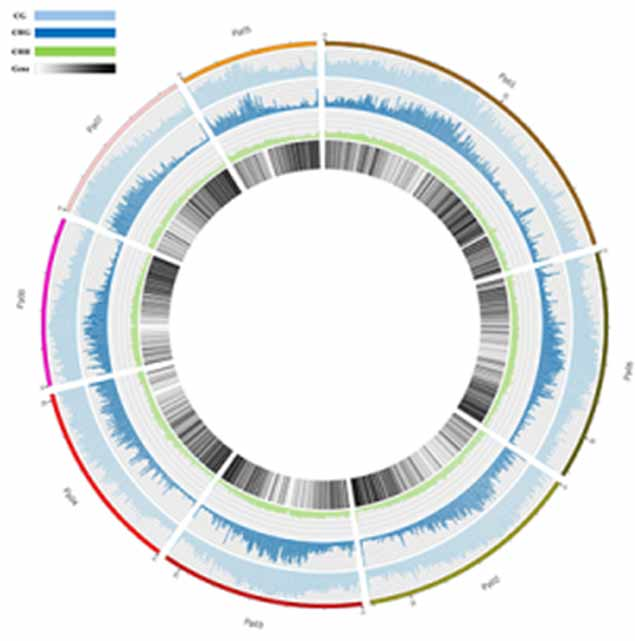
Methylierungskarte. 5mc Methylierungsgenomweite Verteilung
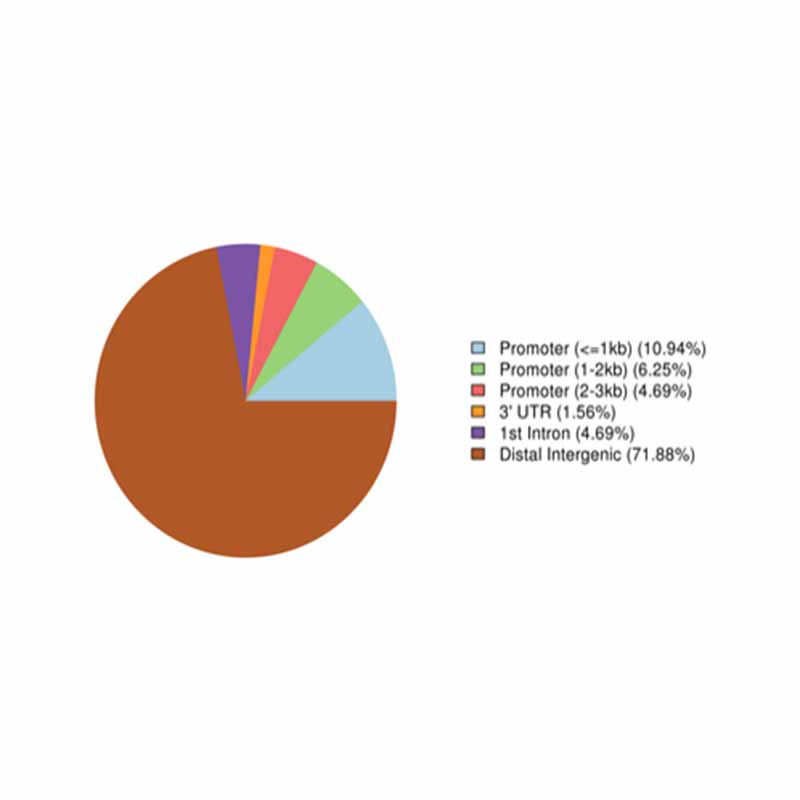
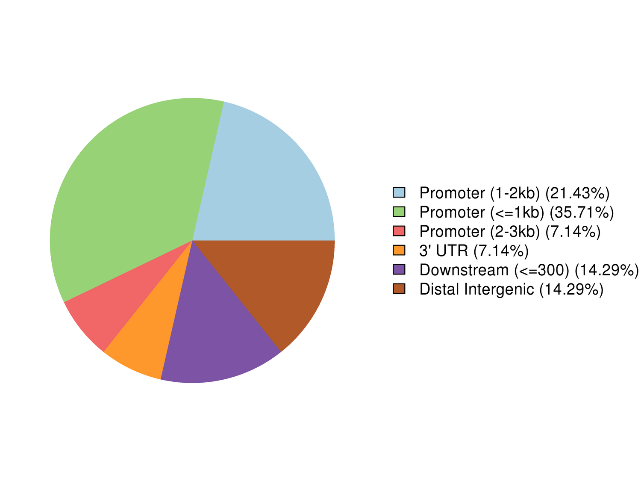
Annotation von stark methylierten Regionen
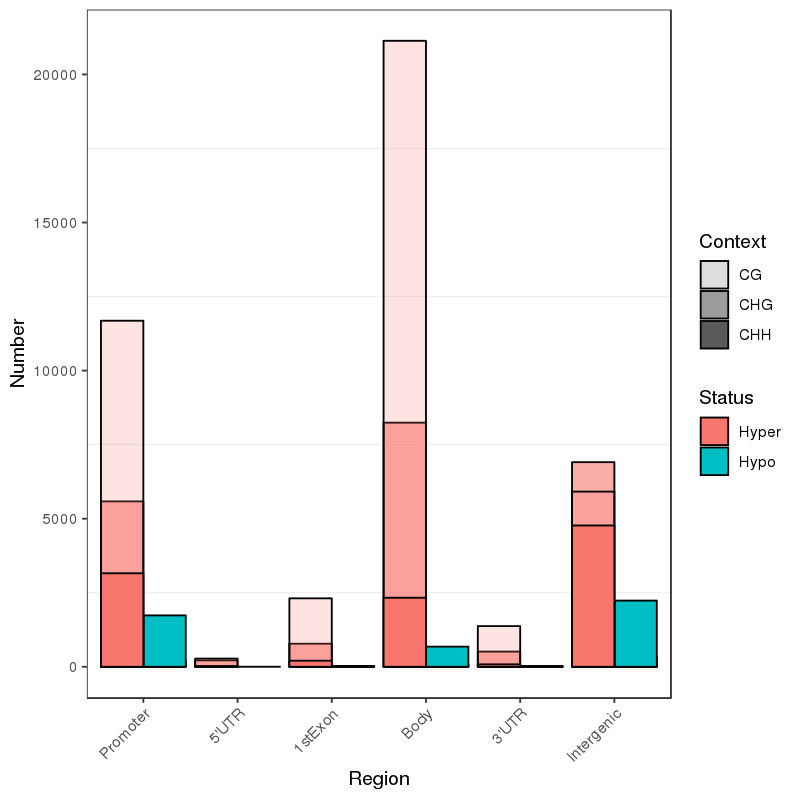
Unterschiedlich methylierte Regionen: assoziierte Gene
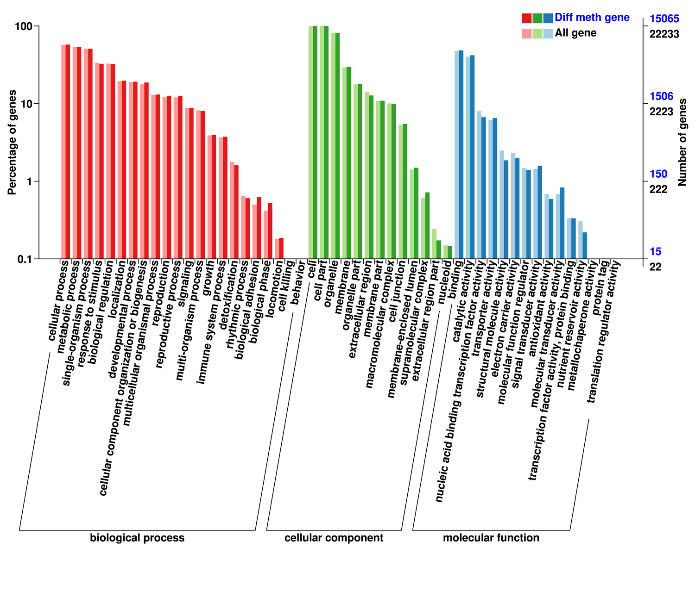
Differentiell methylierte Regionen: Annotation assoziierter Gene (Gen -Ontologie)
Untersuchen Sie die Forschungsergebnisse, die durch die gesamte Genom -Bisulfite -Sequenzierungsdienste von Bmkgene durch eine kuratierte Sammlung von Veröffentlichungen erleichtert wurden.
Fan, Y. et al. (2020) 'Analyse von DNA-Methylierungsprofilen während der Entwicklung des Skelettmuskels unter Verwendung der Bisulfit-Sequenzierung des gesamten Genoms',BMC -Genomik, 21 (1), S. 1–15. doi: 10.1186/s12864-020-6751-5.
Zhao, X. et al. (2022) 'Neue Desoxyribonukleinsäure -Methylierungsstörungen bei Arbeitern, die Vinylchlorid ausgesetzt sind',Toxikologie und industrielle Gesundheit38 (7), S. 377–388. doi: 10.1177/07482337221098600
Zuo, J. et al. (2020) "Beziehungen zwischen Genommethylierung, Spiegel nicht-kodierender RNAs, mRNAs und Metaboliten in Reifentomatenfrüchten",Das Plant Journal, 103 (3), S. 980–994. doi: 10.1111/tpj.14778.