Einzelkern-RNA-Sequenzierung
Technisches Programm
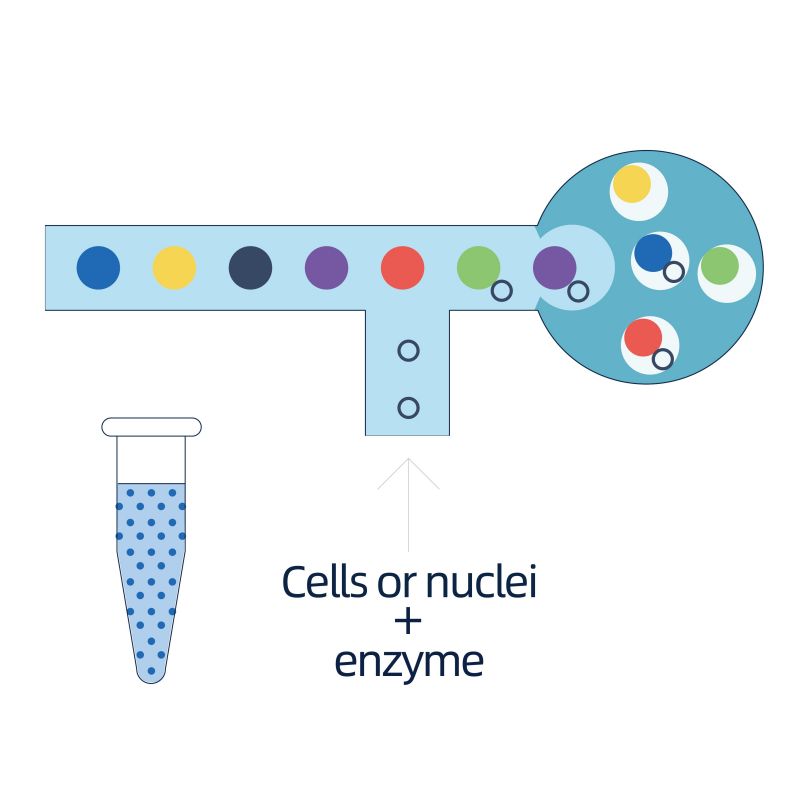
Die Isolierung von Kernen wird durch 10 × Genomics Chromium ™ erreicht, das achtkanales Mikrofluidiksystem mit Doppelübergängen besteht. In diesem System werden eine Gelkügelchen mit Barcodes und Primer, Enzymen und einem einzelnen Kern in einem Ölabfall in Nanolitergröße eingekapselt, wodurch Gel Bead-in-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-In-Insulation). Sobald GEM gebildet ist, werden in jedem Juwel die Zelllyse und Freisetzung von Barcodes durchgeführt. mRNA werden umgekehrt in cDNA -Moleküle mit 10 × Barcodes und UMI transkribiert, die weiter der Standardkonstruktion der Sequenzierungsbibliothek unterliegen.

Merkmale
● Vorbereitung der Einzelnuclei-Suspension aus gefrorenen Geweben
● Bildung von Gel Bead-in-Emulsion (GEM), gefolgt von der cDNA-Synthese
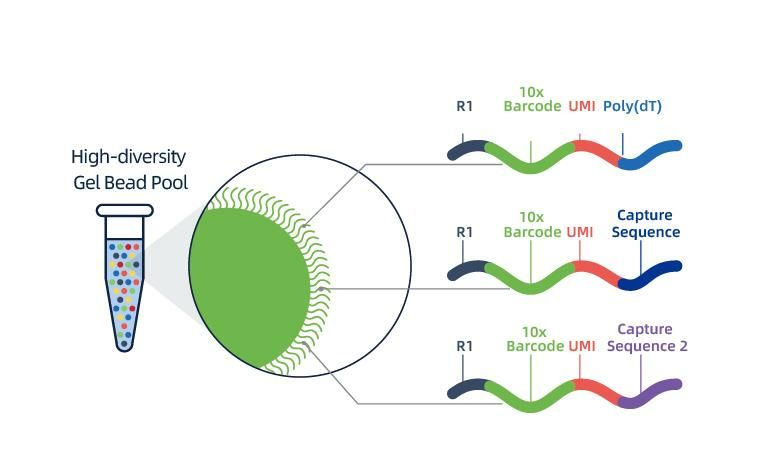
● Jede Perle in einem Edelstein ist mit Primern beladen, die aus 4 Abschnitten bestehen:
Poly (dt) -Schschwanz für mRNA -Priming und cDNA -Synthese,
Eindeutige molekulare Kennung (UMI), um die Verstärkungsverzerrung zu korrigieren
10x Barcode
Bindungssequenz des partiellen Lese -1 -Sequenzierungsprimers
Vorteile
Einzelnukleus-RNA-Sequenzierung umfasst die Einschränkungen der Einzelzell-RNA-Sequenzierung und ermöglicht:
● Die Verwendung von gefrorenen Proben und nicht nur auf frische Proben beschränkt
● Niedrige Stress von gefrorenen Zellen im Vergleich zur enzymatischen Behandlung frischer Zellen, die in den Transkriptomdaten in Form von weniger stressinduzierten Genen spiegeln
● Keine vorherige Entfernung der roten Blutkörperchen erforderlich
● Unbegrenzter Zelldurchmesser
Ur
Proben, die nicht durch Single-Zell-RNA-Sequenzierung analysiert werden können und können für eine einzelne Nuklei-RNA-Sequenzierung berechtigt sind:
| Zelle / Gewebe | Grund |
| Ungespräche gefrorenes Gewebe | Es können keine frischen oder langlebigen Organisationen bekommen |
| Muskelzelle, Megakaryozyte, Fett… | Der Zelldurchmesser ist zu groß, um das Instrument zu betreten |
| Leber… | Zu zerbrechlich, um zu brechen und einzelne Zellen nicht zu unterscheiden |
| Neuronenzelle, Gehirn… | Sensibler, leicht zu belasten, verändert die Sequenzierungsergebnisse |
| Bauchspeicheldrüse, Schilddrüse… | Reich an endogenen Enzymen, die die Produktion der Einzelzellsuspension beeinflussen |
Single-Nucleus vs Single-Cell
| Single-Nucleus | Einzelzell |
| Unbegrenzter Zelldurchmesser | Zelldurchmesser: 10-40 μm |
| Das Material kann gefrorenes Gewebe sein | Das Material muss frisches Gewebe sein |
| Geringer Stress von gefrorenen Zellen | Die Enzymbehandlung kann zu einer Reaktion von Zellstress führen |
| Es müssen keine roten Blutkörperchen entfernt werden | Rote Blutkörperchen müssen entfernt werden |
| Nuklear exprimiert Bioinformation | Die gesamte Zelle exprimiert Bioinformation |
Spezifikationen
| Stichprobenanforderungen | Bibliothek | Sequenzierungsstrategie | Daten empfohlen | Qualitätskontrolle |
| Tiergewebe ≥ 200 mg Pflanzengewebe ≥ 400 mg | 10x Genomics SN -cDNA -Bibliothek | Illumina PE150 | 100K PE liest pro Zelle (100-200 GB) | 700-1200 Kerne/μl und Kernintegrität unter Mikroskop beobachtet |
Weitere Informationen zu den Anleitungen und dem Service -Workflow für Beispiele zur VorbereitungBmkgene Experte
Service Work Flow

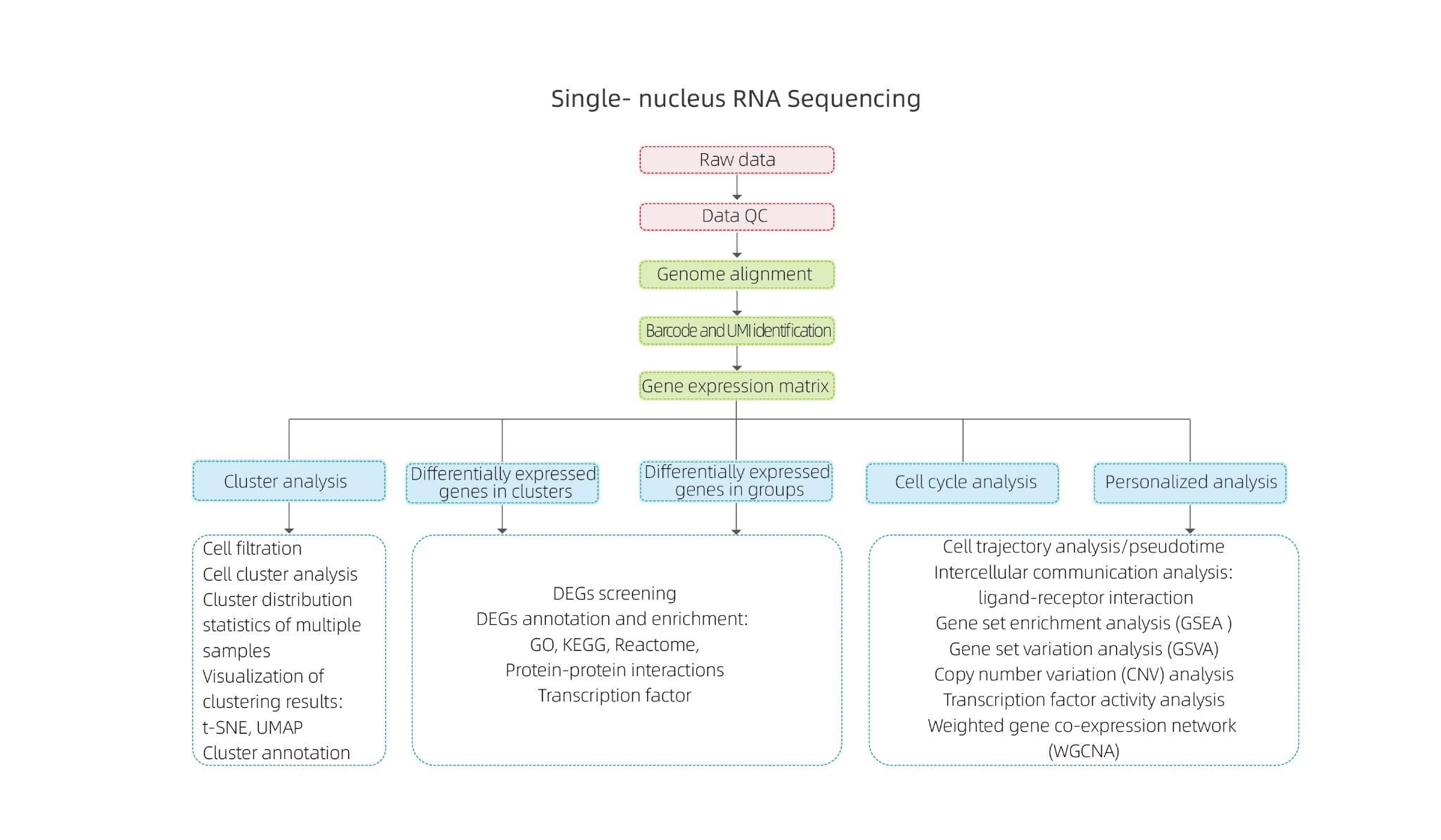
Enthält die folgende Analyse:
● Qualitätskontrolle: Anzahl der Zellen, Gen Nachweis, genaue Identifizierung von Zellen, RNA -Moleküle und Expressionsquantifizierung
● innere Stichprobenanalyse:
Zellclustering und Annotation von Cluster
Differentielle Expressionsanalyse: Identifizierung von DEGs in Clustern
Funktionelle Annotation und Anreicherung von Cluster -DEGs
● Analyse zwischen Gruppen:
Kombination von Daten
Differentiale Expressionsanalyse: Identifizierung von DEGs in Gruppen
Funktionelle Annotation und Anreicherung von Gruppen -DEGs
● Erweiterte Analyse:
Zellzyklusanalyse
Pseudotime -Analyse
Zellkommunikationsanalyse (CellphonedB)
Analyse der Gen -Set -Anreicherung (GSEA)
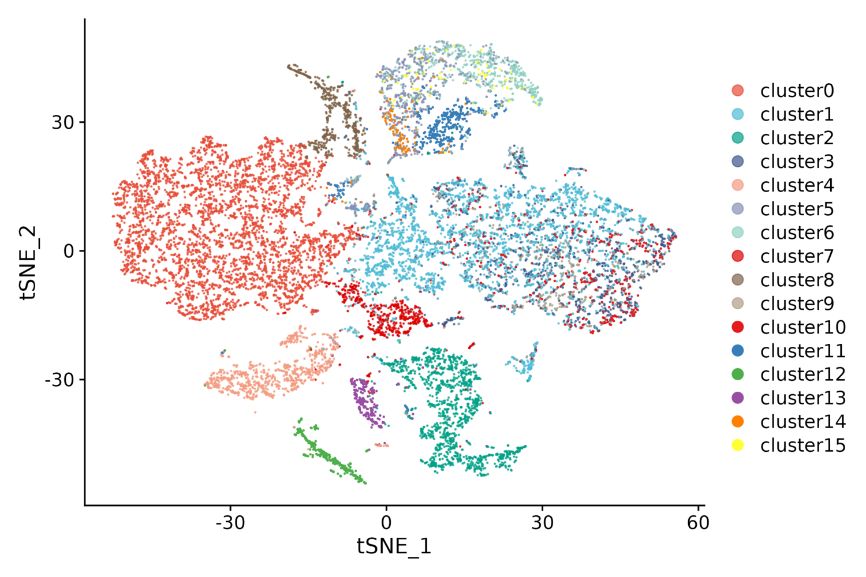
Analyse der inneren Stichprobe
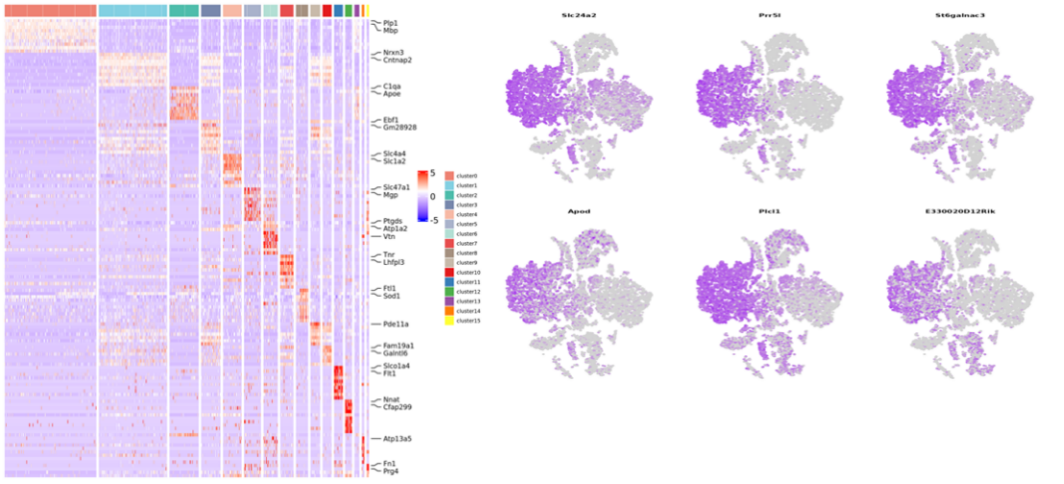
Zellclustering:
Differentielle Expressionsanalyse: Cluster -DEGs
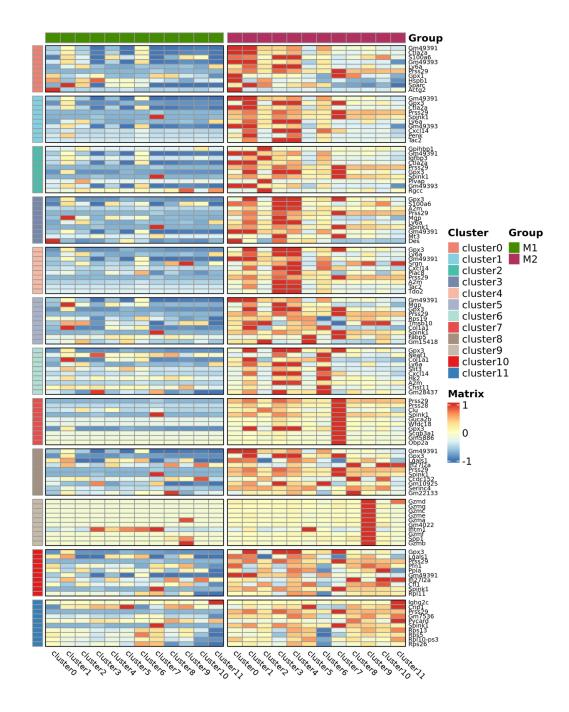
Analyse zwischen Gruppen
Differentielle Expressionsanalyse: Gruppen -DEGs
Erweiterte Analyse:
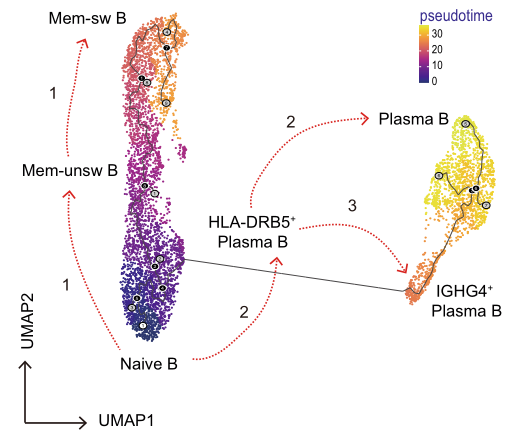
Pseudotime -Analyse:
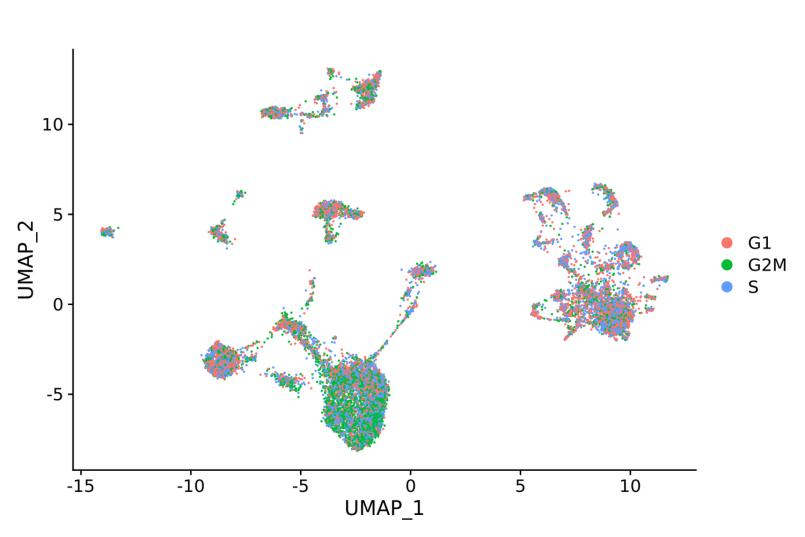
Zellzyklusanalyse:
Untersuchen Sie die Fortschritte, die durch Bmkgene Single-Nucleus-RNA-Sequenzierungsdienste von 10x Chrom in diesen vorgestellten Veröffentlichungen erleichtert wurden:
Wang, L. et al. (2021) 'transkriptomische Einzelzellanalyse zeigt die Immunlandschaft der Lunge in steroidresistenten Asthma-Exazerbation',Verfahren der Nationalen Akademie der Wissenschaften der Vereinigten Staaten von Amerika, 118 (2), p. E2005590118. doi: 10.1073/pnas.2005590118
Zheng, H. et al. (2022) 'ein globales regulatorisches Netzwerk für dysregulierte Genexpression und abnormale metabolische Signalübertragung in Immunzellen in der Mikroumgebung von Gräberkrankheiten und Hashimoto -Thyreoiditis',Grenzen in der Immunologie, 13, p. 879824. Doi: 10.3389/fimmu.2022.879824/bibtex.
Tian, H. et al. (2023) 'Single-Cell-Transkriptom deckt Heterogenität und Immunantworten von Leukozyten nach der Impfung mit inaktiviertem Edwardsiella Tarda in Flunder (Paralichthys Olivaceus)' auf.Aquakultur, 566, p. 739238. Doi: 10.1016/j.aquaculture.2023.739238.
Yu, Y. et al. (2023) 'Photodynamische Therapie verbessert das Ergebnis von Immun-Checkpoint-Inhibitoren durch Umgestaltung der Antitumour-Immunität bei Patienten mit Magenkrebs',Magenkrebs26 (5), S. 798–813. doi: 10.1007/s10120-023-01409-X/Metriken.