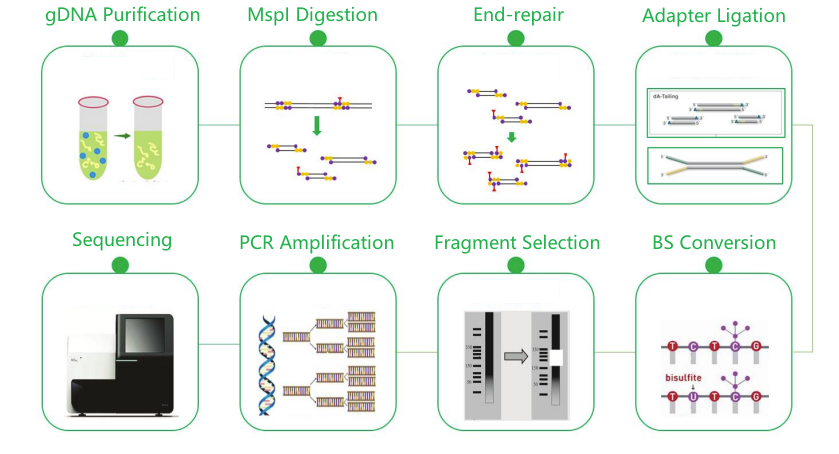
Bisulfit-Sequenzierung mit reduzierter Repräsentation (RRBS)
Servicefunktionen
● Erfordert ein Referenzgenom.
● Lambda-DNA wird zur Überwachung der Bisulfit-Umwandlungseffizienz verwendet.
● Die Effizienz der MspI-Verdauung wird ebenfalls überwacht.
● Doppelter Enzymverdau für Pflanzenproben.
● Sequenzierung auf Illumina NovaSeq.
Servicevorteile
●Kostengünstige und effiziente Alternative zu WGBS: Dadurch kann die Analyse zu geringeren Kosten und mit geringerem Probenbedarf durchgeführt werden.
●Komplette Plattform:bieten exzellenten Service aus einer Hand von der Probenverarbeitung über den Bibliotheksaufbau und die Sequenzierung bis hin zur bioinformatischen Analyse.
●Umfangreiches Fachwissen: Mit erfolgreich abgeschlossenen RRBS-Sequenzierungsprojekten für eine Vielzahl von Arten bringt BMKGENE über ein Jahrzehnt Erfahrung, ein hochqualifiziertes Analyseteam, umfassende Inhalte und exzellenten Post-Sales-Support mit.
Leistungsbeschreibung
| Bibliothek | Sequenzierungsstrategie | Empfohlene Datenausgabe | Qualitätskontrolle |
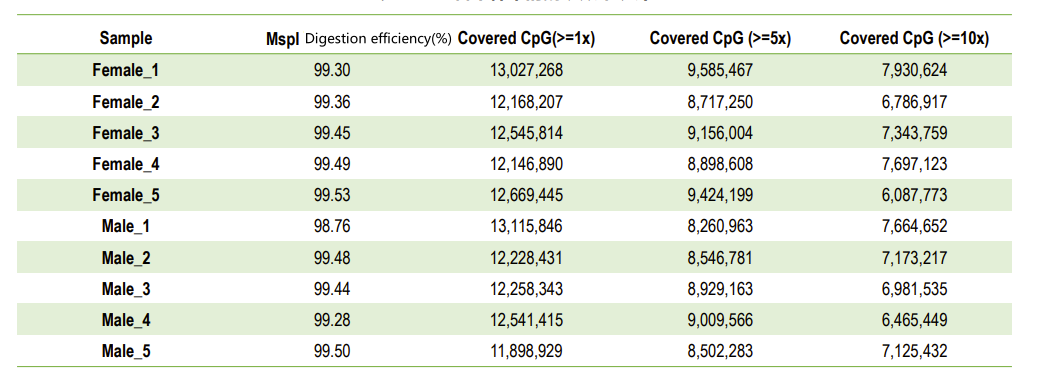
| Mit MspI verdaute und mit Bisulfit behandelte Bibliothek | Illumina PE150 | 8 GB | Q30 ≥ 85 % Bisulfit-Umwandlung > 99 % MspI-Schnitteffizienz > 95 % |
Beispielanforderungen
| Konzentration (ng/µL) | Gesamtmenge (µg) |
| |
| Genomische DNA | ≥ 30 | ≥ 1 | Begrenzte Verschlechterung oder Kontamination |
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse
Kundendienst
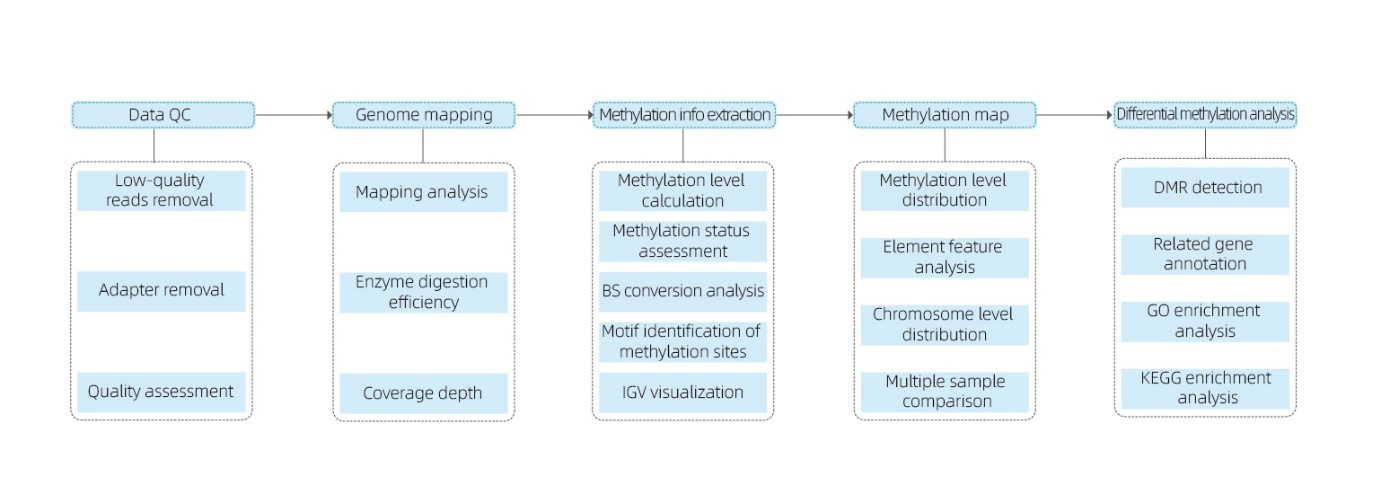
Beinhaltet die folgende Analyse:
● Qualitätskontrolle der Rohsequenzierung;
● Zuordnung zum Referenzgenom;
● Nachweis von 5mC methylierten Basen und Motividentifizierung;
● Analyse der Methylierungsverteilung und Probenvergleich;
● Analyse differentiell methylierter Regionen (DMRs);
● Funktionelle Annotation von Genen, die mit DMRs assoziiert sind.
Qualitätskontrolle: Verdauungseffizienz (bei der Genomkartierung)
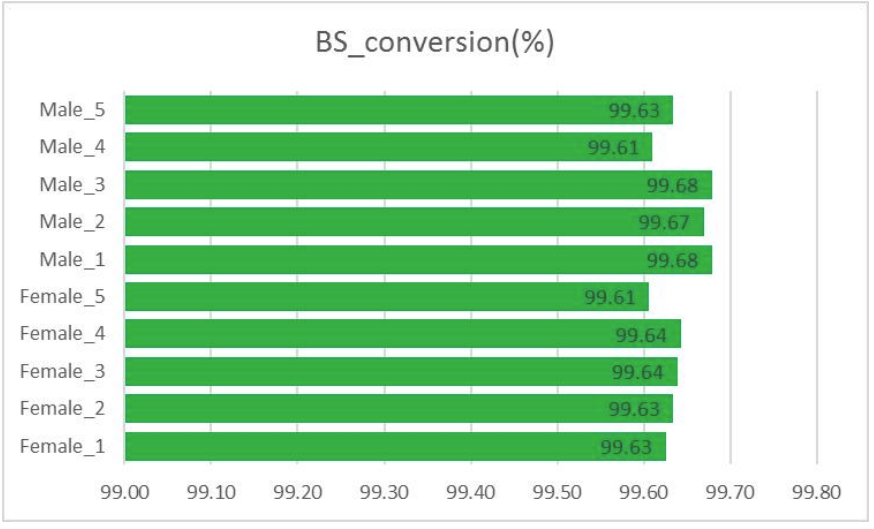
Qualitätskontrolle: Bisulfit-Umwandlung (bei der Extraktion von Methylierungsinformationen)
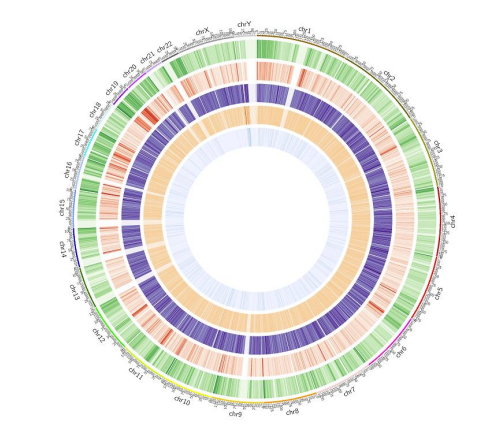
Methylierungskarte: genomweite Verteilung der 5mC-Methylierung
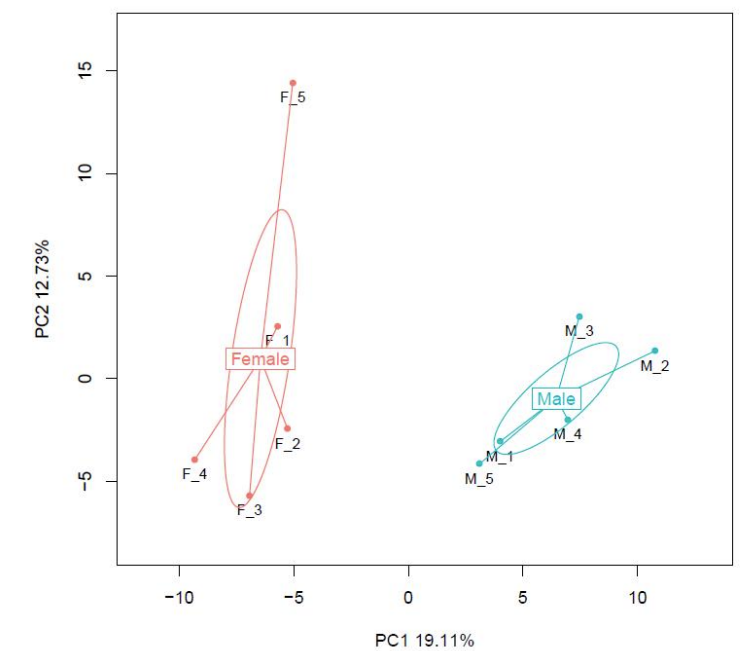
Beispielvergleich: Hauptkomponentenanalyse
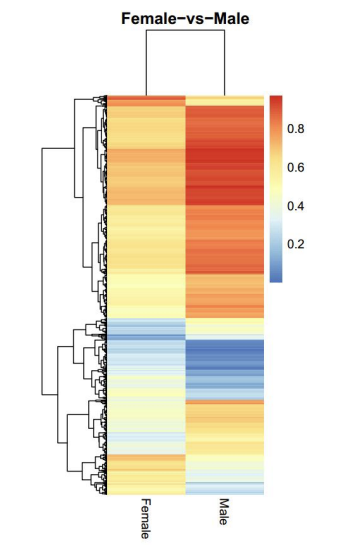
Analyse differenziell methylierter Regionen (DMRs): Heatmap
Entdecken Sie die Forschungsfortschritte, die durch die Bisulfit-Sequenzierungsdienste für das gesamte Genom von BMKGene ermöglicht werden, anhand einer kuratierten Sammlung von Veröffentlichungen.
Li, Z. et al. (2022) „High-Fidelity-Reprogrammierung in Leydig-ähnliche Zellen durch CRISPR-Aktivierung und parakrine Faktoren“,PNAS-Nexus, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. et al. (2023) „Genomweite DNA-Methylierungsanalyse der Körperzusammensetzung bei chinesischen eineiigen Zwillingen“,Europäisches Journal für klinische Untersuchungen, 53(11), S. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) „DNA-Methylierung und Verhältnis von Taille zu Hüfte: eine epigenomweite Assoziationsstudie an chinesischen monozygoten Zwillingen“,Zeitschrift für endokrinologische Untersuchung, 45(12), S. 2365–2376. doi: 10.1007/S40618-022-01878-4.