-

Metagenomische Sequenzierung – NGS
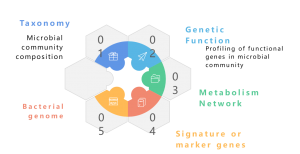
Ein Metagenom ist eine Sammlung des gesamten genetischen Materials einer gemischten Organismengemeinschaft, beispielsweise Umwelt- und menschliche Metagenome. Es enthält Genome sowohl kultivierbarer als auch nicht kultivierbarer Mikroorganismen. Die Shotgun-Metagenomsequenzierung mit NGS ermöglicht die Untersuchung dieser komplexen Genomlandschaften, die in Umweltproben eingebettet sind, indem sie mehr als nur taxonomische Profilerstellung bietet und auch detaillierte Einblicke in die Artenvielfalt, die Abundanzdynamik und komplexe Populationsstrukturen liefert. Über taxonomische Studien hinaus bietet die Shotgun-Metagenomik auch eine funktionale genomische Perspektive und ermöglicht die Erforschung kodierter Gene und ihrer mutmaßlichen Rolle in ökologischen Prozessen. Schließlich trägt die Etablierung von Korrelationsnetzwerken zwischen genetischen Elementen und Umweltfaktoren zu einem ganzheitlichen Verständnis des komplexen Zusammenspiels zwischen mikrobiellen Gemeinschaften und ihrem ökologischen Hintergrund bei. Zusammenfassend lässt sich sagen, dass die metagenomische Sequenzierung ein zentrales Instrument zur Entschlüsselung der genomischen Feinheiten verschiedener mikrobieller Gemeinschaften darstellt und die vielfältigen Beziehungen zwischen Genetik und Ökologie innerhalb dieser komplexen Ökosysteme beleuchtet.
Plattformen: Illumina NovaSeq und DNBSEQ-T7
-

Metagenomische Sequenzierung-TGS
Ein Metagenom ist eine Sammlung des genetischen Materials einer gemischten Organismengemeinschaft, beispielsweise Umwelt- und menschliche Metagenome. Es enthält Genome sowohl kultivierbarer als auch nicht kultivierbarer Mikroorganismen. Die metagenomische Sequenzierung ermöglicht die Untersuchung dieser komplexen Genomlandschaften, die in ökologische Proben eingebettet sind, indem sie mehr als nur taxonomische Profilerstellung bietet. Es bietet auch eine funktionelle Genomik-Perspektive durch die Erforschung der kodierten Gene und ihrer mutmaßlichen Rolle in Umweltprozessen. Während traditionelle Shotgun-Ansätze mit Illumina-Sequenzierung in metagenomischen Studien weit verbreitet sind, hat das Aufkommen der Nanopore- und PacBio-Long-Read-Sequenzierung das Gebiet verändert. Die Nanopore- und PacBio-Technologie verbessert nachgelagerte bioinformatische Analysen, insbesondere die Metagenom-Assemblierung, und gewährleistet so eine kontinuierlichere Assemblierung. Berichte weisen darauf hin, dass die auf Nanoporen und PacBio basierende Metagenomik erfolgreich vollständige und geschlossene Bakteriengenome aus komplexen Mikrobiomen erzeugt hat (Moss, EL, et al., Nature Biotech, 2020). Die Integration von Nanopore-Lesevorgängen mit Illumina-Lesevorgängen bietet einen strategischen Ansatz zur Fehlerkorrektur und mildert die inhärente geringe Genauigkeit von Nanopore. Diese synergetische Kombination nutzt die Stärken jeder Sequenzierungsplattform und bietet eine robuste Lösung zur Überwindung potenzieller Einschränkungen sowie zur Verbesserung der Präzision und Zuverlässigkeit metagenomischer Analysen.
Plattform: Nanopore PromethION 48, Illumia und PacBio Revio
-

Bisulfit-Sequenzierung des gesamten Genoms (WGBS)
Whole Genome Bisulfit Sequencing (WGBS) gilt als Goldstandard-Methode für die eingehende Untersuchung der DNA-Methylierung, insbesondere der fünften Position in Cytosin (5-mC), einem zentralen Regulator der Genexpression und Zellaktivität. Das WGBS zugrunde liegende Prinzip umfasst die Behandlung mit Bisulfit, die die Umwandlung nicht methylierter Cytosine in Uracil (C zu U) induziert, während methylierte Cytosine unverändert bleiben. Diese Technik bietet eine Einzelbasenauflösung und ermöglicht es Forschern, das Methylom umfassend zu untersuchen und abnormale Methylierungsmuster aufzudecken, die mit verschiedenen Erkrankungen, insbesondere Krebs, verbunden sind. Durch den Einsatz von WGBS können Wissenschaftler beispiellose Einblicke in genomweite Methylierungslandschaften gewinnen und so ein differenziertes Verständnis der epigenetischen Mechanismen ermöglichen, die verschiedenen biologischen Prozessen und Krankheiten zugrunde liegen.
-

Assay für Transposase-zugängliches Chromatin mit Hochdurchsatzsequenzierung (ATAC-seq)
ATAC-seq ist eine Hochdurchsatz-Sequenzierungstechnik, die für die genomweite Analyse der Zugänglichkeit von Chromatin verwendet wird. Der Einsatz ermöglicht ein tieferes Verständnis der komplexen Mechanismen der globalen epigenetischen Kontrolle der Genexpression. Die Methode nutzt eine hyperaktive Tn5-Transposase, um gleichzeitig offene Chromatinregionen durch Einfügen von Sequenzierungsadaptern zu fragmentieren und zu markieren. Durch anschließende PCR-Amplifikation entsteht eine Sequenzierungsbibliothek, die eine umfassende Identifizierung offener Chromatinregionen unter spezifischen Raum-Zeit-Bedingungen ermöglicht. ATAC-seq bietet eine ganzheitliche Sicht auf zugängliche Chromatinlandschaften, im Gegensatz zu Methoden, die sich ausschließlich auf Transkriptionsfaktor-Bindungsstellen oder spezifische Histon-modifizierte Regionen konzentrieren. Durch die Sequenzierung dieser offenen Chromatinregionen deckt ATAC-seq Regionen mit höherer Wahrscheinlichkeit für aktive regulatorische Sequenzen und potenzielle Transkriptionsfaktor-Bindungsstellen auf und bietet wertvolle Einblicke in die dynamische Modulation der Genexpression im gesamten Genom.
-

16S/18S/ITS-Amplikonsequenzierung-PacBio
Die 16S- und 18S-rRNA-Gene dienen zusammen mit der ITS-Region (Internal Transcribed Spacer) aufgrund ihrer Kombination aus hochkonservierten und hypervariablen Regionen als zentrale molekulare Fingerabdruckmarker, was sie zu unschätzbaren Werkzeugen zur Charakterisierung prokaryotischer und eukaryotischer Organismen macht. Die Amplifikation und Sequenzierung dieser Regionen bietet einen isolierten Ansatz zur Untersuchung der mikrobiellen Zusammensetzung und Diversität in verschiedenen Ökosystemen. Während die Illumina-Sequenzierung typischerweise auf kurze hypervariable Regionen wie V3-V4 von 16S und ITS1 abzielt, wurde gezeigt, dass durch die Sequenzierung der gesamten Länge von 16S, 18S und ITS eine überlegene taxonomische Annotation erreichbar ist. Dieser umfassende Ansatz führt zu höheren Prozentsätzen genau klassifizierter Sequenzen und erreicht ein Auflösungsniveau, das bis zur Artenidentifizierung reicht. Die Single-Molecule Real-Time (SMRT)-Sequenzierungsplattform von PacBio zeichnet sich durch hochpräzise Long Reads (HiFi) aus, die die Amplifikate in voller Länge abdecken und mit der Präzision der Illumina-Sequenzierung mithalten können. Diese Fähigkeit ermöglicht Forschern einen unübertroffenen Vorteil – einen Panoramablick auf die genetische Landschaft. Die erweiterte Abdeckung erhöht die Auflösung bei der Annotation von Arten erheblich, insbesondere innerhalb von Bakterien- oder Pilzgemeinschaften, und ermöglicht so ein tieferes Verständnis der Feinheiten mikrobieller Populationen.
-

16S/18S/ITS-Amplikon-Sequenzierung-NGS
Die Amplikonsequenzierung mit Illumina-Technologie, die speziell auf die genetischen Marker 16S, 18S und ITS abzielt, ist eine leistungsstarke Methode zur Aufklärung der Phylogenie, Taxonomie und Artenhäufigkeit innerhalb mikrobieller Gemeinschaften. Dieser Ansatz beinhaltet die Sequenzierung der hypervariablen Regionen haushaltsgenetischer Marker. Ursprünglich als molekularer Fingerabdruck eingeführt vonWoeses et al1977 revolutionierte diese Technik die Mikrobiom-Profilierung, indem sie isolationsfreie Analysen ermöglichte. Durch die Sequenzierung von 16S (Bakterien), 18S (Pilze) und Internal Transscribed Spacer (ITS, Pilze) können Forscher nicht nur häufig vorkommende, sondern auch seltene und nicht identifizierte Arten identifizieren. Die Amplikonsequenzierung, die weithin als zentrales Werkzeug eingesetzt wird, hat sich als entscheidend für die Erkennung unterschiedlicher mikrobieller Zusammensetzungen in unterschiedlichen Umgebungen erwiesen, darunter im menschlichen Mund, im Darm, im Stuhl und darüber hinaus.
-

Neusequenzierung des gesamten Genoms von Bakterien und Pilzen
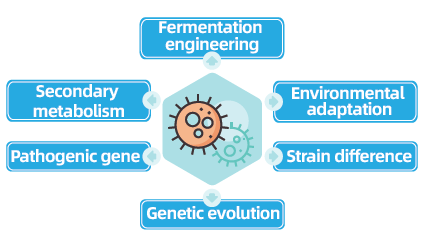
Projekte zur Neusequenzierung des gesamten Genoms von Bakterien und Pilzen sind von entscheidender Bedeutung für die Weiterentwicklung der mikrobiellen Genomik, da sie die Vervollständigung und den Vergleich mikrobieller Genome ermöglichen. Dies erleichtert die Fermentationstechnik, die Optimierung industrieller Prozesse und die Erforschung sekundärer Stoffwechselwege. Darüber hinaus ist die Neusequenzierung von Pilzen und Bakterien von entscheidender Bedeutung, um die Anpassung an die Umwelt zu verstehen, Stämme zu optimieren und die Dynamik der genetischen Evolution aufzudecken, mit weitreichenden Auswirkungen auf die Medizin, Landwirtschaft und Umweltwissenschaften.
-

PacBio – 16S/18S/ITS-Amplikonsequenzierung in voller Länge
Die Amplicon-Plattform (16S/18S/ITS) wurde mit jahrelanger Erfahrung in der Projektanalyse der mikrobiellen Diversität entwickelt und umfasst standardisierte Basisanalysen und personalisierte Analysen: Die Basisanalyse deckt den Mainstream-Analyseinhalt der aktuellen mikrobiellen Forschung ab, der Analyseinhalt ist reichhaltig und umfassend. und Analyseergebnisse werden in Form von Projektberichten präsentiert; Die Inhalte der personalisierten Analyse sind vielfältig. Je nach grundlegendem Analysebericht und Forschungszweck können Proben ausgewählt und Parameter flexibel eingestellt werden, um individuelle Anforderungen zu erfüllen. Windows-Betriebssystem, einfach und schnell.
-

PacBio-Transkriptom in voller Länge (keine Referenz)
Mithilfe der Isoform-Sequenzierungsdaten von Pacific Biosciences (PacBio) als Eingabe ist diese App in der Lage, Transkriptsequenzen in voller Länge (ohne Assemblierung) zu identifizieren. Durch die Kartierung von Sequenzen voller Länge gegen das Referenzgenom können Transkripte durch bekannte Gene, Transkripte, kodierende Regionen usw. optimiert werden. In diesem Fall kann eine genauere Identifizierung von mRNA-Strukturen, wie z. B. alternatives Spleißen usw., erreicht werden. Die gemeinsame Analyse mit NGS-Transkriptomsequenzierungsdaten ermöglicht eine umfassendere Annotation und eine genauere Quantifizierung der Expression auf Transkriptebene, was der nachgelagerten differenziellen Expression und Funktionsanalyse weitgehend zugute kommt.
-

Bisulfit-Sequenzierung mit reduzierter Repräsentation (RRBS)
Die reduzierte Bisulfit-Sequenzierung (RRBS) hat sich in der DNA-Methylierungsforschung als kostengünstige und effiziente Alternative zur Bisulfit-Sequenzierung des gesamten Genoms (WGBS) herausgestellt. Während WGBS durch die Untersuchung des gesamten Genoms mit Einzelbasenauflösung umfassende Erkenntnisse liefert, können die hohen Kosten ein limitierender Faktor sein. RRBS mildert diese Herausforderung strategisch durch die selektive Analyse eines repräsentativen Teils des Genoms. Diese Methode beruht auf der Anreicherung von CpG-Insel-reichen Regionen durch MspI-Spaltung und anschließender Größenauswahl von 200-500/600-bps-Fragmenten. Folglich werden nur Regionen in der Nähe von CpG-Inseln sequenziert, während diejenigen mit entfernten CpG-Inseln von der Analyse ausgeschlossen werden. Dieser Prozess ermöglicht in Kombination mit der Bisulfit-Sequenzierung einen hochauflösenden Nachweis der DNA-Methylierung, und der Sequenzierungsansatz PE150 konzentriert sich speziell auf die Enden der Inserts und nicht auf die Mitte, wodurch die Effizienz der Methylierungsprofilierung erhöht wird. Das RRBS ist ein unschätzbar wertvolles Werkzeug, das eine kostengünstige DNA-Methylierungsforschung ermöglicht und das Wissen über epigenetische Mechanismen erweitert.
-

Prokaryotische RNA-Sequenzierung
Die RNA-Sequenzierung ermöglicht die umfassende Profilierung aller RNA-Transkripte in Zellen unter bestimmten Bedingungen. Diese hochmoderne Technologie dient als wirksames Werkzeug und enthüllt komplexe Genexpressionsprofile, Genstrukturen und molekulare Mechanismen, die mit verschiedenen biologischen Prozessen verbunden sind. Die in der Grundlagenforschung, der klinischen Diagnostik und der Arzneimittelentwicklung weit verbreitete RNA-Sequenzierung bietet Einblicke in die Feinheiten der Zelldynamik und der genetischen Regulation. Unsere prokaryotische RNA-Probenverarbeitung ist auf prokaryotische Transkriptome zugeschnitten und umfasst die rRNA-Depletion und die gerichtete Bibliotheksvorbereitung.
Plattform: Illumina NovaSeq
-

Metatranskriptomsequenzierung
Der Metatranskriptom-Sequenzierungsdienst von BMKGENE nutzt die Sequenzierungstechnologie von Illumina und enthüllt die dynamische Genexpression einer Vielzahl von Mikroben, von Eukaryoten über Prokaryoten bis hin zu Viren, in natürlichen Umgebungen wie Boden, Wasser, Meer, Stuhl und Darm. Unser umfassender Service ermöglicht es Forschern, in die vollständigen Genexpressionsprofile komplexer mikrobieller Gemeinschaften einzutauchen. Über die taxonomische Analyse hinaus erleichtert unser Metatranskriptom-Sequenzierungsdienst die Erforschung der funktionellen Bereicherung und wirft Licht auf unterschiedlich exprimierte Gene und ihre Rollen. Entdecken Sie eine Fülle biologischer Erkenntnisse, während Sie sich durch die komplexen Landschaften der Genexpression, der taxonomischen Vielfalt und der Funktionsdynamik in diesen vielfältigen Umweltnischen bewegen.
-

De-novo-Pilzgenomassemblierung
BMKGENE bietet vielseitige Lösungen für Pilzgenome, die auf unterschiedliche Forschungsbedürfnisse und die gewünschte Vollständigkeit des Genoms eingehen. Allein die Verwendung der Short-Read-Illumina-Sequenzierung ermöglicht die Generierung eines Entwurfsgenoms. Short-Reads und Long-Read-Sequenzierung mit Nanopore oder Pacbio werden kombiniert, um ein verfeinertes Pilzgenom mit längeren Contigs zu erhalten. Darüber hinaus verbessert die Integration der Hi-C-Sequenzierung die Fähigkeiten weiter und ermöglicht die Erlangung eines vollständigen Genoms auf Chromosomenebene.