Nicht referenzbasierte mRNA-Sequenzierungs-NGs
Merkmale
● Erfassung von Poly -mRNA vor der Vorbereitung der Bibliothek
● unabhängig von jedem Referenzgenom: Basierend auf der De-novo-Zusammenstellung von Transkripten eine Liste von Unigenen, die mit mehreren Datenbanken kommentiert werden (NR, Schweizerisch, COG, KOG, Eierlikör, PFAM, GEG, KEGG)
● umfassende bioinformatische Analyse der Genexpression und Transkriptstruktur
Servicevorteile
●Umfangreiches Fachwissen: Mit einer Erfolgsbilanz bei der Verarbeitung von über 600.000 Proben bei BMKgene, die verschiedene Stichprobentypen wie Zellkulturen, Gewebe und Körperflüssigkeiten umfasst, bringt unser Team jedem Projekt eine Fülle von Erfahrung ein. Wir haben über 100.000 mRNA-Seq-Projekte in verschiedenen Forschungsbereichen erfolgreich abgeschlossen.
●Strenge Qualitätskontrolle: Wir implementieren Kernkontrollpunkte in allen Stufen, von der Proben- und Bibliotheksvorbereitung bis hin zur Sequenzierung und Bioinformatik. Diese akribische Überwachung sorgt für die Bereitstellung konsequent hochwertiger Ergebnisse.
● Umfassende Annotation: Wir verwenden mehrere Datenbanken, um die differentiell exprimierten Gene (DEGs) funktional zu kommentieren und die entsprechende Anreicherungsanalyse durchzuführen.
●Unterstützung nach dem Verkauf: Unser Engagement geht über den Abschluss des Projekts mit einem 3-monatigen Nachverkaufsdauer hinaus. Während dieser Zeit bieten wir Projekt-Follow-up, Fehlerbehebung und Q & A-Sitzungen an, um alle Abfragen im Zusammenhang mit den Ergebnissen zu beheben.
Probenanforderungen und Lieferung
| Bibliothek | Sequenzierungsstrategie | Daten empfohlen | Qualitätskontrolle |
| Poly A angereichert | Illumina PE150 DNBSEQ-T7 | 6-10 GB | Q30 ≥ 85% |
Beispielanforderungen:
Nukleotide:
| Conc. (Ng/μl) | Menge (μg) | Reinheit | Integrität |
| ≥ 10 | ≥ 0,2 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Begrenzte oder keine Protein- oder DNA -Kontamination auf Gel. | Für Pflanzen: Rin ≥ 4,0; Für Tiere: Rin ≥ 4,5; 5,0 ≥ 28s/18s ≥ 1,0; begrenzte oder keine Grundlinienerhebung |
● Pflanzen:
Wurzel, Stamm oder Blütenblatt: 450 mg
Blatt oder Samen: 300 mg
Frucht: 1,2 g
● Tier:
Herz oder Darm: 300 mg
Eingeweide oder Gehirn: 240 mg
Muskel: 450 mg
Knochen, Haare oder Haut: 1g
● Arthropoden:
Insekten: 6g
Crustacea: 300 mg
● Vollblut: 1 Röhre
● Zellen: 106 Zellen
Empfohlene Probenzustellung
Behälter: 2 ml Zentrifugenrohr (Zinnfolie wird nicht empfohlen)
Beispielkennzeichnung: Gruppe+Replikat zB A1, A2, A3; B1, B2, B3.
Lieferung:
1. Trockeneis: Proben müssen in Beutel gepackt und in Trockeneis begraben werden.
2. RNAsable -Röhrchen: RNA -Proben können in RNA -Stabilisierungsrohr (z. B. RNAsable®) getrocknet und in Raumtemperatur versendet werden.
Service Work Flow

Experimententwurf

Probenabgabe

RNA -Extraktion

Bibliothekskonstruktion

Sequenzierung

Datenanalyse

Nachverkaufsdienste
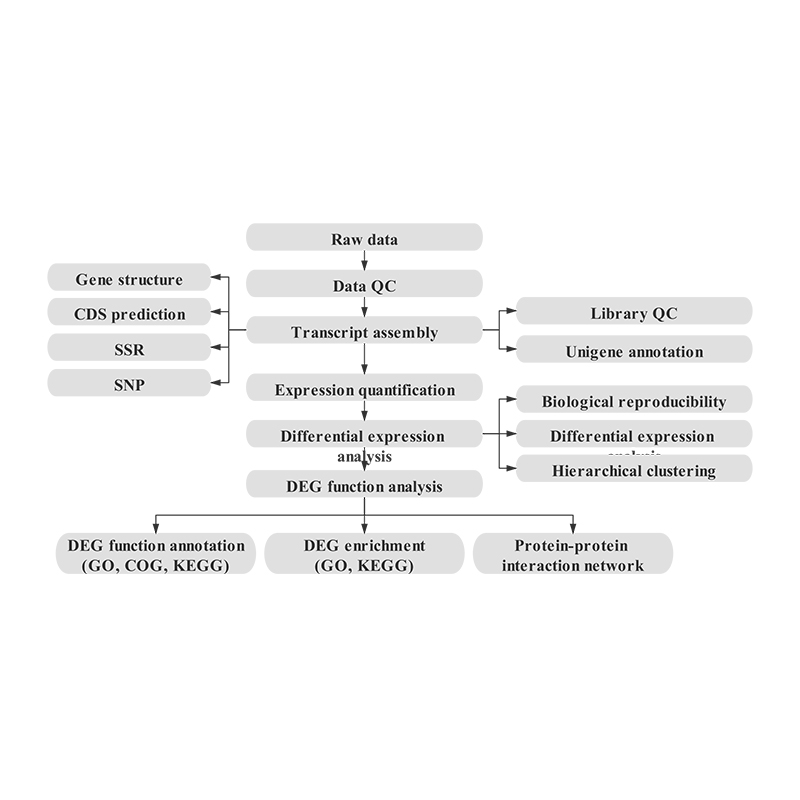
Bioinformatik
Transkriptomanordnung und Unigene -Auswahl
Unigen -Annotation
Probenkorrelation und Bewertung biologischer Replikate
Differentiell exprimierte Gene (DEGs)
Funktionale Annotation von DEGs
Funktionsanreicherung von DEGs
Untersuchen Sie die Fortschritte, die durch die eukaryotischen NGS -mRNA -Sequenzierungsdienste von BMKgene durch eine kuratierte Sammlung von Veröffentlichungen erleichtert wurden.
Shen, F. et al. (2020) 'De-novo Transkriptom-Assemblierung und geschlechtsspezifische Genexpression in den Gonaden von Amur-Wels (Silurus asotus), Genomics, 112 (3), S. 2603–2614. doi: 10.1016/j.ygeno.2020.01.026.
Zhang, C. et al. (2016) 'Transkriptomanalyse des Saccharose -Stoffwechsels während der Gliebensschwellung und -entwicklung in Zwiebeln (Allium cepa L.)', Grenzen in Plant Science, 7 (September), p. 212763. Doi: 10.3389/fpls.2016.01425/bibtex.
Zhu, C. et al. (2017) 'De -novo -Versammlung, Charakterisierung und Annotation für das Transkriptom von Sarcocheilthys sinensis', PLOS ONE, 12 (2). doi: 10.1371/journal.pone.0171966.
Zou, L. et al. (2021) 'De -novo -Transkriptomanalyse liefert Einblicke in die Salztoleranz von Podocarpus macrophyllus unter Salzgehaltstress', BMC Plant Biology, 21 (1), S. 1–17. doi: 10.1186/s12870-021-03274-1/figures/9.