Genomentwicklung, Pangenom
Was ist Pan-Genom?
Angesammelte Beweise zeigen, dass die Varianz zwischen verschiedenen Stämmen einer Art riesig sein kann. Ein einziges Genom ist weit ausreichend, um das gesamte Bild genetischer Informationen einer einzelnen Spezies zu erhalten. Der Zweck der Pan-Genom-Studie besteht darin, eine umfassendere genomische Graphe einer Spezies zu erhalten und die Beziehungen zwischen Merkmalen und genetischen Codes zu dekodieren, indem Genom de novo-Assemblierung zahlreicher Stämme durchgeführt wird, was einen tieferen und breiteren Abbau der Variationen ermöglicht.
Trends der Pan-Genom-Studie
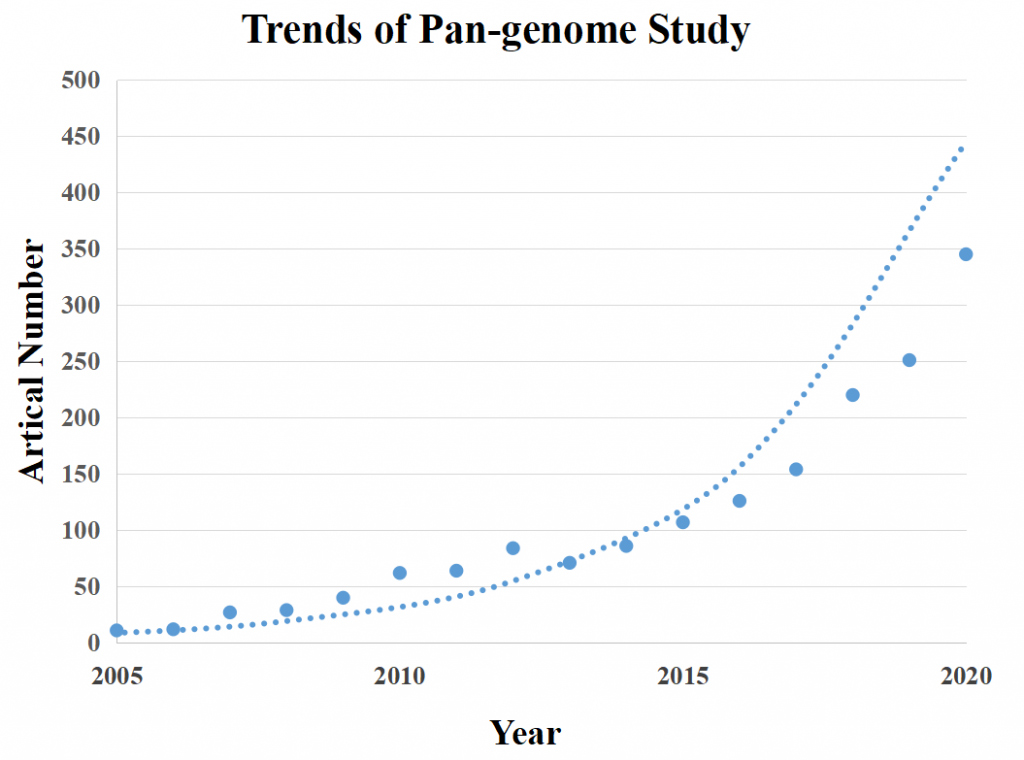
Abbildung 1 Die Trends der veröffentlichten Forschungsarbeiten von Pan-Genom.
HINWEIS: Die Abbildung zeigt das Ergebnis der Einnahme von „Pan-Genom“ als Schlüsselwort zur Suche nach den Titeln von Artikeln, die in Nature, Cell and Science Series Journals veröffentlicht wurden
A. Lesevorgänge aus mehreren Proben sind auf eine Referenz ausgerichtet und nicht ausgerichtete, werden in neuartigen Contigs zusammengesetzt. Durch Hinzufügen dieser neuartigen Contigs zur ursprünglichen Referenzsequenz kann eine Pangenomreferenz konstruiert werden. Dispensable -Regionen werden basierend auf der Zuordnung aller Lesevorgänge zum Pangenom ermittelt.
B. Die De -novo -Assemblierung der Genome mehrerer Akzessionen ermöglicht die Identifizierung von entbehrlichen genomischen Regionen.
C. Ein Pan-Genom-Diagramm kann aus Alignments des gesamten Genoms oder der De-novo-Diagramm-Assemblierung konstruiert werden, die die varianten Informationen von entbehrlichen Regionen effizient als eindeutige Pfade durch die Grafik speichert.
Wie konstruiere ich ein Pan-Genom?
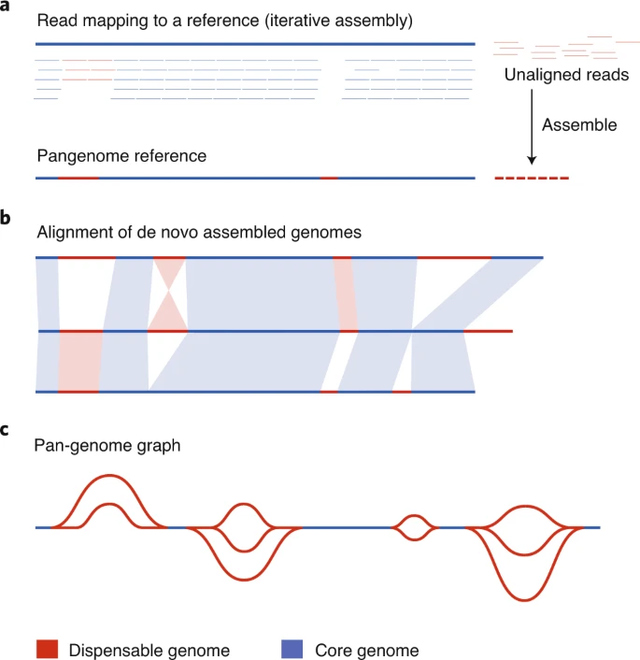
Abbildung 2 Vergleich von Pan-Genom-Ansätzen1
A. Lesevorgänge aus mehreren Proben sind auf eine Referenz ausgerichtet und nicht ausgerichtete, werden in neuartigen Contigs zusammengesetzt. Durch Hinzufügen dieser neuartigen Contigs zur ursprünglichen Referenzsequenz kann eine Pangenomreferenz konstruiert werden. Dispensable -Regionen werden basierend auf der Zuordnung aller Lesevorgänge zum Pangenom ermittelt.
B. Die De -novo -Assemblierung der Genome mehrerer Akzessionen ermöglicht die Identifizierung von entbehrlichen genomischen Regionen.
C. Ein Pan-Genom-Diagramm kann aus Alignments des gesamten Genoms oder der De-novo-Diagramm-Assemblierung konstruiert werden, die die varianten Informationen von entbehrlichen Regionen effizient als eindeutige Pfade durch die Grafik speichert.
Kürzlich veröffentlichte Pan-Genome
● Vergewaltigungs-Pan-Genome2

● Tomaten-Pan-Genome 3

● Reispfanne -Genome4

● Sonnenblumen-Pan-Genom5

● Sojabohnen-Pan-Genom 6

● Reis-Pan-Genom7

● Gerste-Pan-Genom8

● Weizenpfannengenom9

● Sorghum Pan-Genom10

● Phytoplankton-Pan-Genom11

Referenz
1. Bayer PE, Golicz AA, Scheben A., Batley J., Edwards D. Pflanzen-Pan-Genome sind die neue Referenz. Nat -Pflanzen. 2020; 6 (8): 914-920. doi: 10.1038/s41477-020-0733-0
2. Song JM, Guan Z, Hu J. et al. Acht hochwertige Genome zeigen Pan-Genom-Architektur und Ökotypdifferenzierung von Brassica Napus. Nat -Pflanzen. 2020; 6 (1): 34-45. doi: 10.1038/s41477-019-0577-7
3. Gao L, Gonda I, Sun H, et al. Das Tomaten-Pan-Genom entdeckt neue Gene und ein seltenes Allel, das Obstgeschmack reguliert. Nat Genet. 2019; 51 (6): 1044-1051. doi: 10.1038/s41588-019-0410-2
4. Zhao Q, Feng Q, Lu H, et al. Die Pan-Genom-Analyse unterstreicht das Ausmaß der genomischen Variation von kultiviertem und wildem Reis [veröffentlichte Korrektur erscheint im NAT-Genet. 2018 Aug; 50 (8): 1196]. Nat Genet. 2018; 50 (2): 278-284. doi: 10.1038/s41588-018-0041-z
5. Hübner S, Bercovich N, Todesco M, et al. Sonnenblumen-Pan-Genom-Analyse zeigt, dass die Hybridisierung den Gengehalt und die Krankheitsresistenz verändert hat. Nat -Pflanzen. 2019; 5 (1): 54-62. doi: 10.1038/s41477-018-0329-0
6. Liu Y, Du H, Li P, et al. Pan-Genom aus wilden und kultivierten Sojabohnen. Zelle. 2020; 182 (1): 162-176.E13. doi: 10.1016/j.cell.2020.05.023
7. Qin P, Lu H, Du H, et al. Die Pan-Genom-Analyse von 33 genetisch vielfältigen Reisakzitionen zeigt versteckte genomische Variationen [online vor dem Druck, 2021, 25. Mai]. Zelle. 2021; S0092-8674 (21) 00581-x. doi: 10.1016/j.cell.2021.04.046
8. Jayakodi M, Padmarasu S., Haberer G. et al. Das Gerste-Pan-Genom zeigt das verborgene Erbe der Mutationszucht. Natur. 2020; 588 (7837): 284-289. doi: 10.1038/s41586-020-2947-8
9. Walkowiak S., Gao L., Monat C. et al. Mehrere Weizengenome zeigen globale Variationen in der modernen Zucht. Natur. 2020; 588 (7837): 277-283. doi: 10.1038/s41586-020-2961-x
10. Tao Y, Luo H, Xu J, et al. Umfangreiche Variationen im Pan-Genom von kultiviertem und wildem Sorghum [online vor Druck, 2021 20. Mai]. Nat -Pflanzen. 2021; 10.1038 / s41477-021-00925-x. doi: 10.1038/s41477-021-00925-x
11. Fan X, Qiu H., Han W. et al. Das Phytoplankton -Pangenom zeigt einen umfangreichen prokaryotischen horizontalen Gentransfer verschiedener Funktionen. SCI adv. 2020; 6 (18): EABA0111. Veröffentlicht 2020 Apr 29. doi: 10.1126/sciadv.aba0111
Postzeit: Jan.-04-2022


