Sequenzierung des gesamten menschlichen Exoms
Servicefunktionen
● Zwei Exom-Panels verfügbar, basierend auf der Zielanreicherung mit Sonden: Sure Select Human All Exon v6 (Agilent) und xGen Exome Hybridization Panel v2 (IDT).
● Sequenzierung auf Illumina NovaSeq.
● Bioinformatische Pipeline, die auf die Analyse von Krankheiten oder Tumoren ausgerichtet ist.
Servicevorteile
●Zielt auf die proteinkodierende Region ab: Durch die Erfassung und Sequenzierung proteinkodierender Regionen wird hWES verwendet, um Varianten im Zusammenhang mit der Proteinstruktur aufzudecken.
●Kostengünstig:hWES liefert etwa 85 % der mit menschlichen Krankheiten verbundenen Mutationen aus 1 % des menschlichen Genoms.
●Hohe Genauigkeit: Mit der hohen Sequenzierungstiefe erleichtert hWES die Erkennung sowohl häufiger Varianten als auch seltener Varianten mit Häufigkeiten von weniger als 1 %.
●Strenge Qualitätskontrolle: Wir implementieren fünf zentrale Kontrollpunkte in allen Phasen, von der Proben- und Bibliotheksvorbereitung bis hin zur Sequenzierung und Bioinformatik. Diese sorgfältige Überwachung gewährleistet die Lieferung gleichbleibend hochwertiger Ergebnisse.
●Umfassende bioinformatische Analyse: Unsere Pipeline geht über die Identifizierung von Variationen des Referenzgenoms hinaus, da sie fortschrittliche Analysen umfasst, die speziell auf Forschungsfragen im Zusammenhang mit genetischen Aspekten von Krankheiten oder der Tumoranalyse ausgerichtet sind.
●Post-Sales-Support:Unser Engagement geht über den Projektabschluss hinaus und umfasst einen dreimonatigen Kundendienstzeitraum. Während dieser Zeit bieten wir Projektnachverfolgung, Unterstützung bei der Fehlerbehebung und Frage-und-Antwort-Sitzungen an, um alle Fragen zu den Ergebnissen zu beantworten.
Beispielspezifikationen
| Exon-Capture-Strategie | Sequenzierungsstrategie | Empfohlene Datenausgabe |
| Sure Select Human All Exon v6 (Agilent) oder xGen Exome Hybridization Panel v2 (IDT)
| Illumina NovaSeq PE150 | 5–10 GB Bei Mendelschen Störungen/seltenen Krankheiten: > 50x Für Tumorproben: ≥ 100x |
Beispielanforderungen
| Probentyp
| Menge(Qubit®)
| Konzentration | Volumen
| Reinheit (NanoDrop™) |
|
Genomische DNA
| ≥ 50 ng | ≥ 6 ng/μL | ≥ 15 μL | OD260/280=1,8-2,0 Keine Verschlechterung, keine Kontamination
|
Bioinformatik

Die bioinformatische Analyse von hWES-Erkrankungsproben umfasst:
● Sequenzierungsdaten-QC
● Referenzgenomausrichtung
● Identifizierung von SNPs und InDels
● Funktionale Annotation von SNPs und InDels

Die bioinformatische Analyse von Tumorproben umfasst:
● Sequenzierungsdaten-QC
● Referenzgenomausrichtung
● Identifizierung von SNPs, InDels und somatischen Variationen
● Identifizierung von Keimbahnvarianten
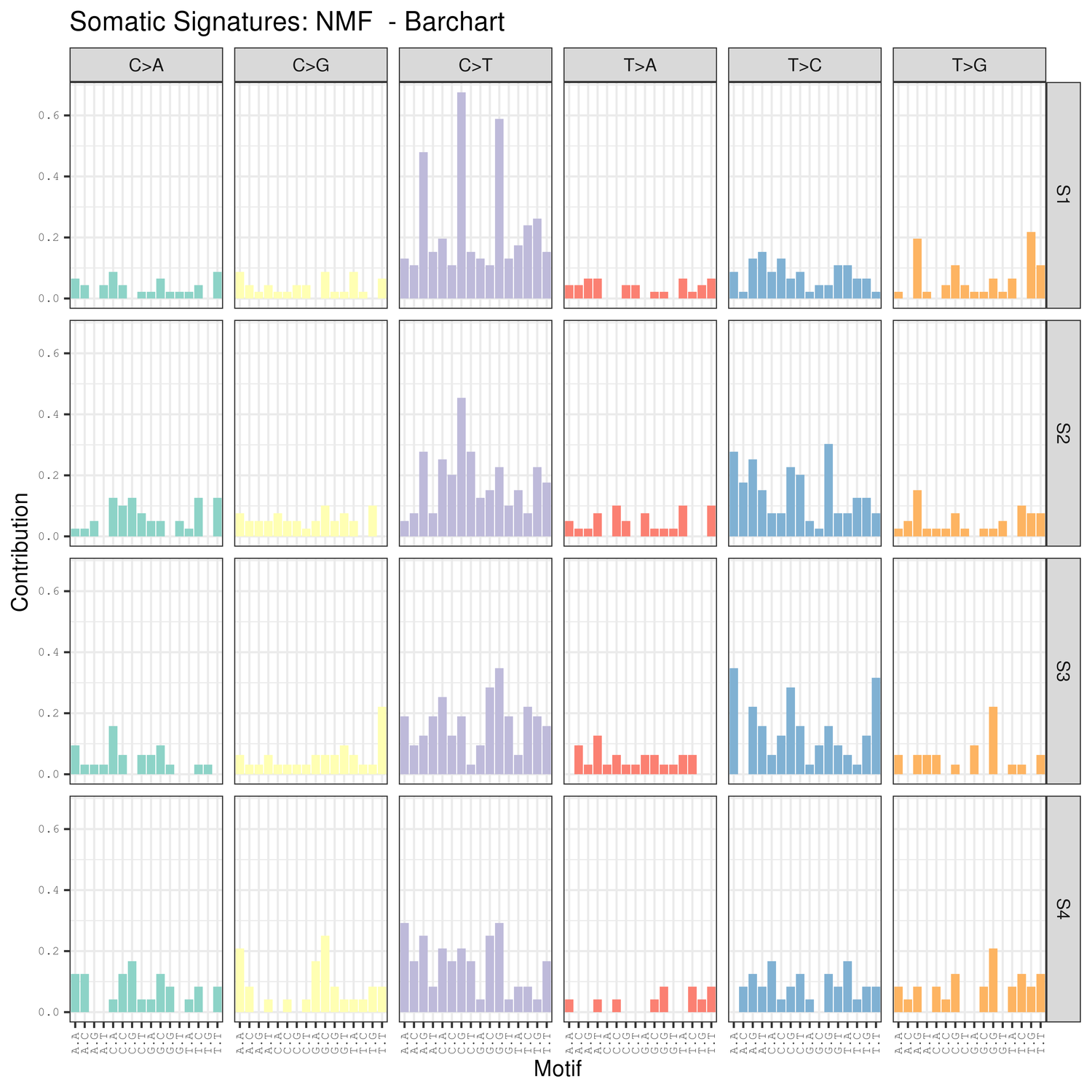
● Analyse von Mutationssignaturen
● Identifizierung von Antriebsgenen anhand von Gain-of-Function-Mutationen
● Mutationsanmerkung auf der Ebene der Arzneimittelanfälligkeit
● Heterogenitätsanalyse – Berechnung von Reinheit und Ploidie
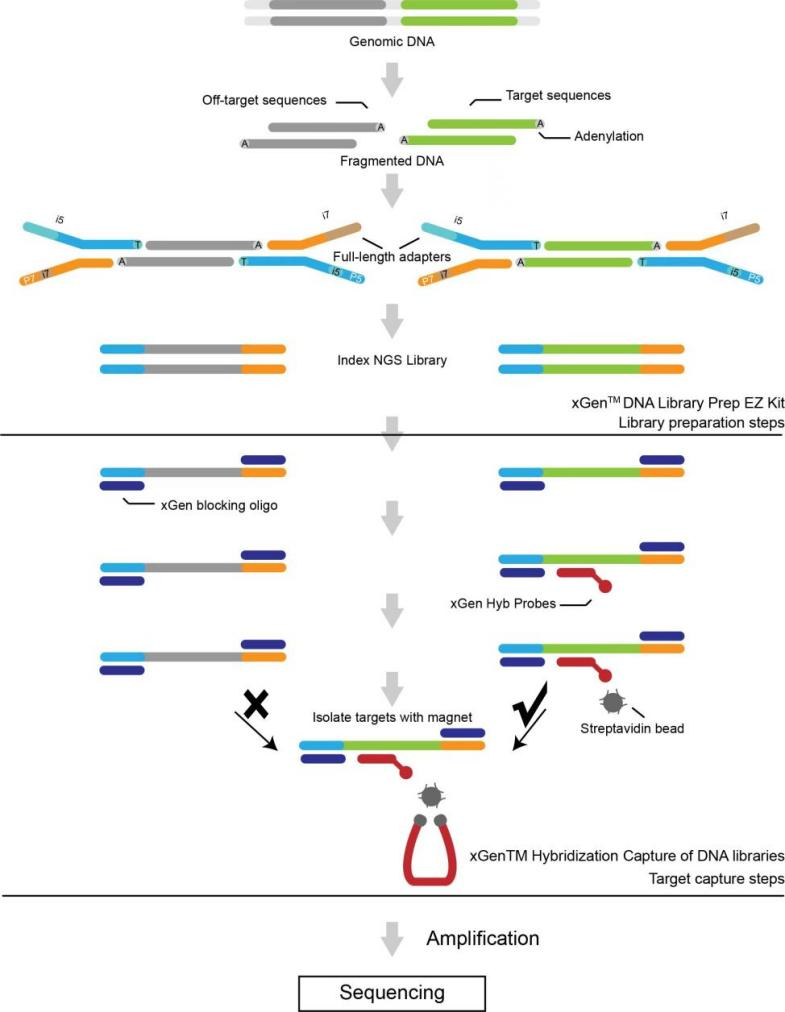
Service-Workflow

Musterlieferung

DNA-Extraktion

Bibliotheksbau

Sequenzierung

Datenanalyse

Datenlieferung

Kundendienst
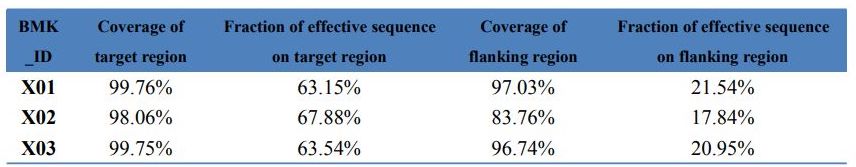
Daten-QC – Statistik der Exom-Erfassung
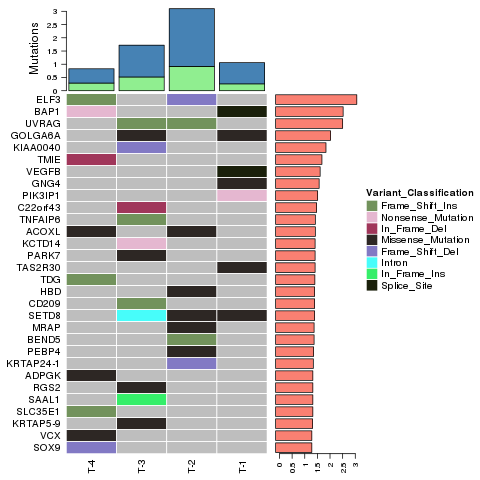
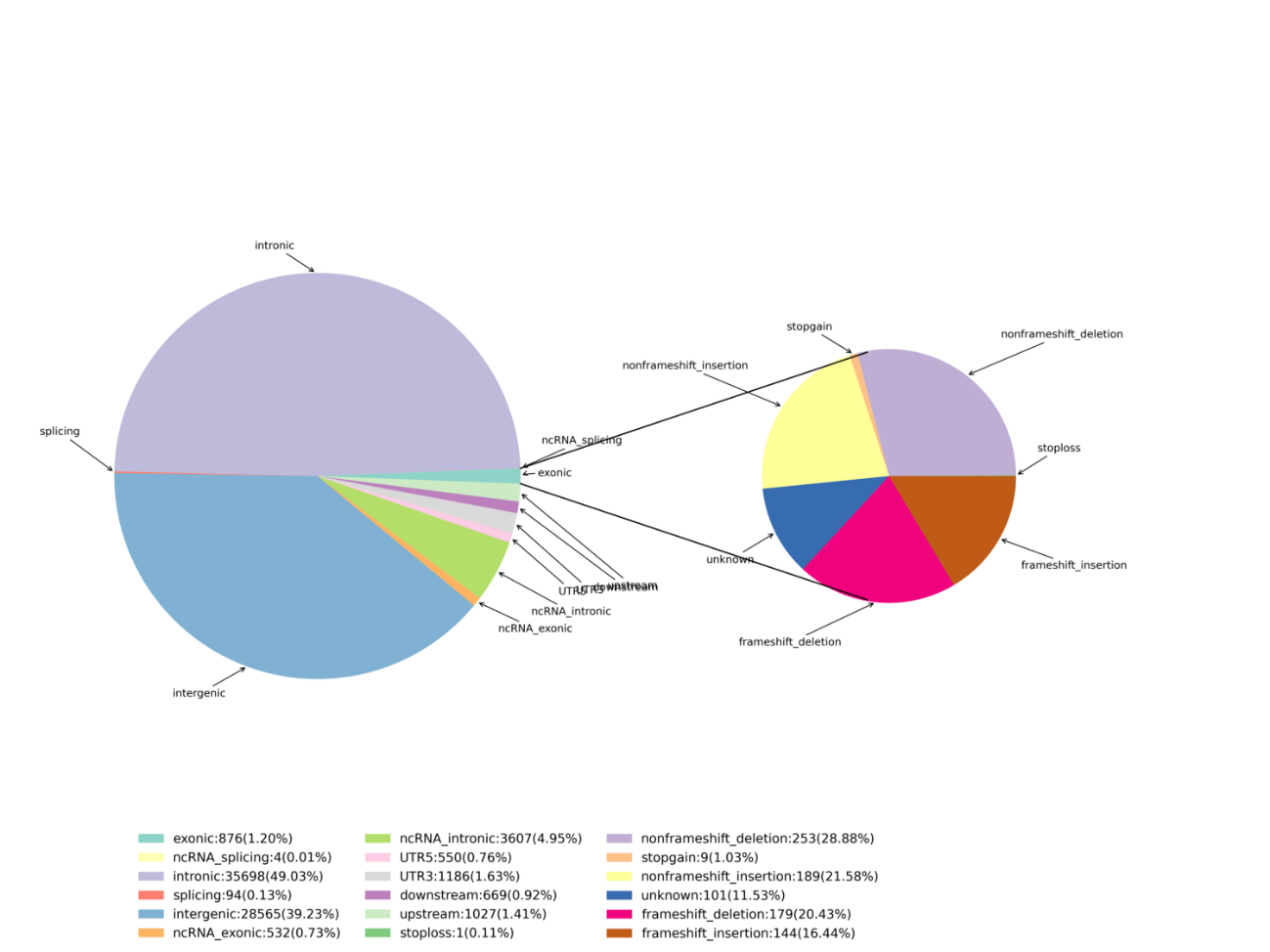
Variantenidentifikation – InDels
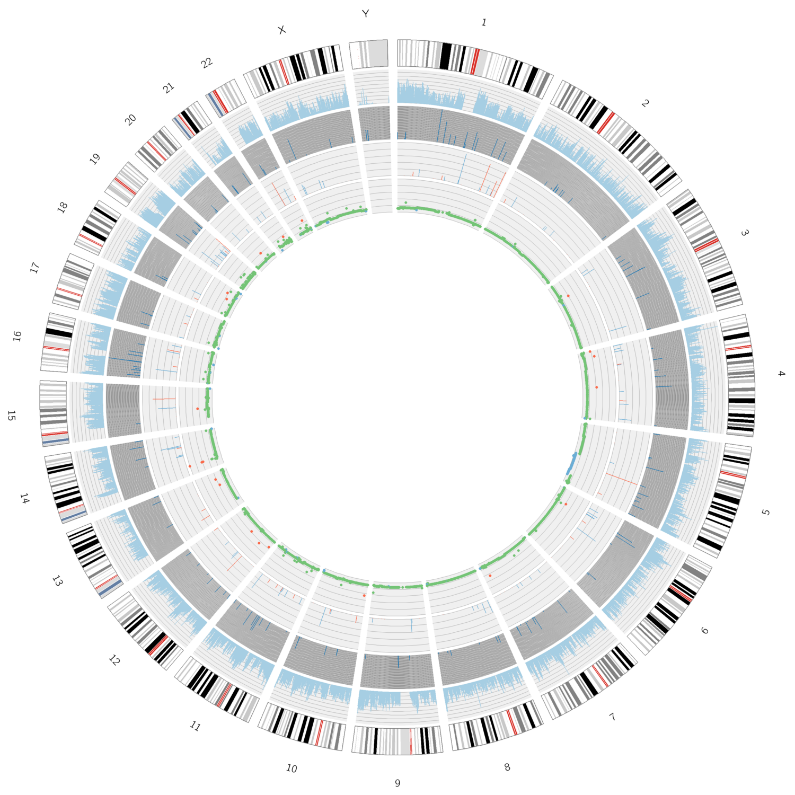
Erweiterte Analyse: Identifizierung und Verteilung schädlicher SNPs/InDels – Circos-Plot
Tumoranalyse: Identifizierung und Verteilung somatischer Mutationen – Circos-Plot
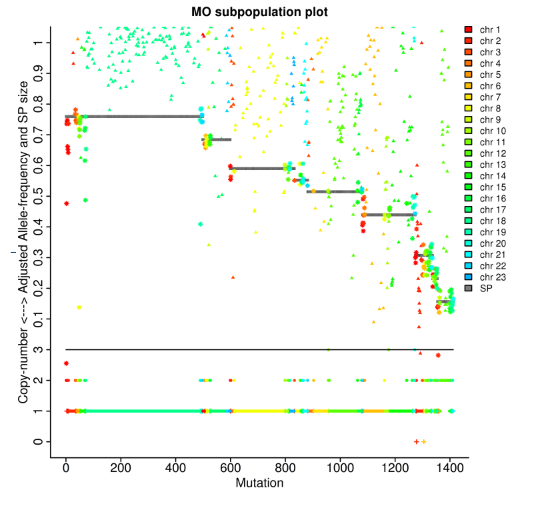
Tumoranalyse: klonale Abstammungslinien