Hi-C-basierte Genomanordnung
Servicefunktionen
● Sequenzierung auf Illumina Novaseq mit PE150.
● Der Service erfordert Gewebeproben anstelle von extrahierten Nukleinsäuren, um mit Formaldehyd zu vernetzen und die DNA-Protein-Wechselwirkungen zu sparen.
● Das Hi-C-Experiment beinhaltet eine Einschränkung und die Endreparatur der klebrigen Enden mit Biotin, gefolgt von einer Kreislauf der resultierenden stumpfen Enden, während die Wechselwirkungen erhalten bleiben. Die DNA wird dann mit Streptavidin -Perlen heruntergezogen und für die nachfolgende Bibliotheksvorbereitung gereinigt.
Servicevorteile

Überblick über Hi-C
(Lieberman-Aidgen E et al.,,Wissenschaft, 2009)
●Beseitigung der Notwendigkeit genetischer Populationsdaten:HI-C ersetzt die wesentlichen Informationen, die für die Verankerung von Contig erforderlich sind.
●Hohe Markerdichte:was zu einem hohen Verankerungsverhältnis über 90%führt.
●Umfangreiche Expertise und Veröffentlichungsunterlagen:BMKgene verfügt über umfangreiche Erfahrung mit mehr als 2000 Fällen von Hi-C-Genom-Assemblierung aus 1000 verschiedenen Arten und verschiedenen Patenten. Über 200 veröffentlichte Fälle haben einen akkumulativen Einflussfaktor von über 2000.
●Hochqualifiziertes Bioinformatik -Team:Mit internen Patenten und Software-Urheberrechten für Hi-C-Experimente und Datenanalyse ermöglicht die selbst entwickelte Visualisierungsdatensoftware manuelles Blockbewegung, Umkehr, Widerruf und Wiederholung.
●Unterstützung nach dem Verkauf:Unser Engagement geht über den Abschluss des Projekts hinaus mit einer 3-monatigen Nachverkaufsdauer hinaus. Während dieser Zeit bieten wir Projekt-Follow-up, Fehlerbehebung und Q & A-Sitzungen an, um alle Abfragen im Zusammenhang mit den Ergebnissen zu beheben.
●Umfassende Annotation: Wir verwenden mehrere Datenbanken, um die Gene funktional mit identifizierten Variationen zu kommentieren und die entsprechende Anreicherungsanalyse durchzuführen.
Servicespezifikationen
| Bibliotheksvorbereitung | Sequenzierungsstrategie | Empfohlene Datenausgabe | Qualitätskontrolle |
| Hi-C-Bibliothek | Illumina Novaseq PE150 | 100x | Q30 ≥ 85% |
Stichprobenanforderungen
| Gewebe | Erforderliche Menge |
| Tierviscera | ≥ 2 g |
| Tiermuskel | |
| Blutes Säugetier | ≥ 2 ml |
| Geflügel/Fischblut | |
| Pflanzlich- frisches Blatt | ≥ 3 g |
| Kultivierte Zellen | ≥ 1x107 |
| Insekt | ≥ 2 g |
Service Work Flow

Experimententwurf

Probenabgabe

Bibliothekskonstruktion

Sequenzierung

Datenanalyse

Nachverkaufsdienste
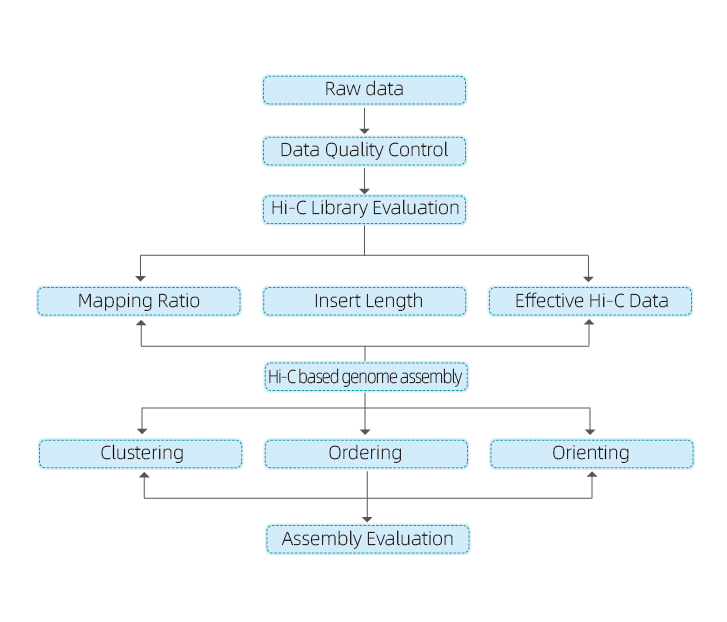
1) Rohdaten QC
2) HI-C-Bibliothek QC: Schätzung gültiger Hi-C-Interaktionen
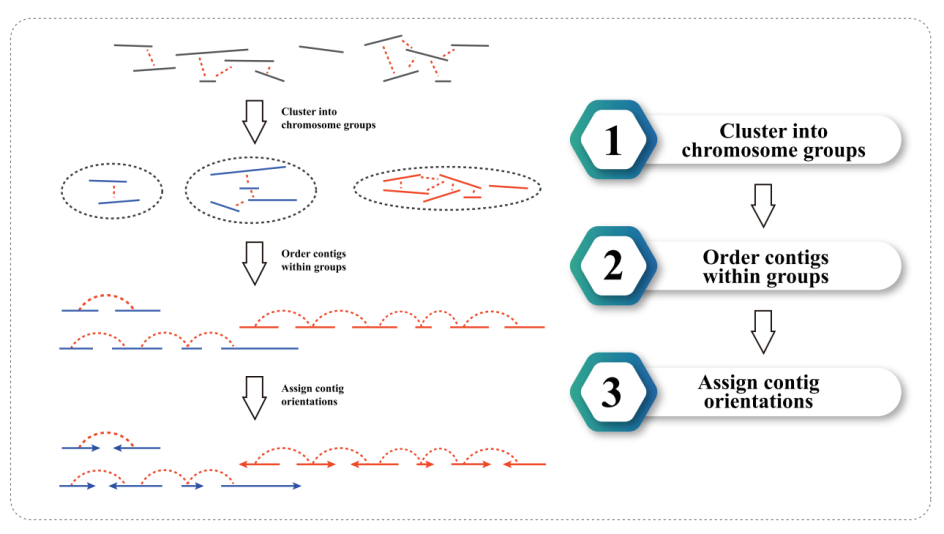
3) HI-C-Assemblierung: Clustering von Contigs in Gruppen, gefolgt von Contig-Order in jeder Gruppe und zugewiesener Contig-Orientierung
4) HI-C-Bewertung
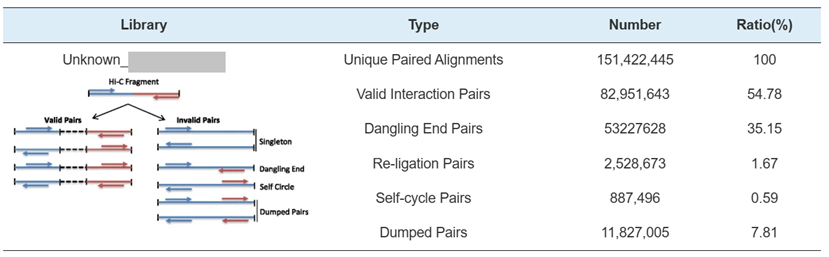
HI-C-Bibliothek QC-Schätzung von Hi-C-gültigen Interaktionspaaren
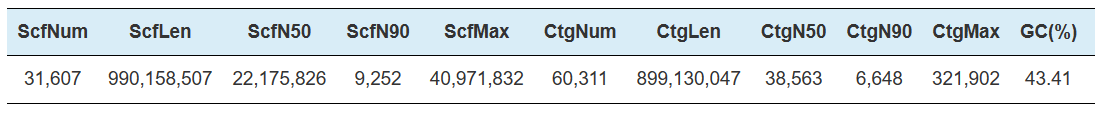
Hi-C-Assemblierung-Statistik
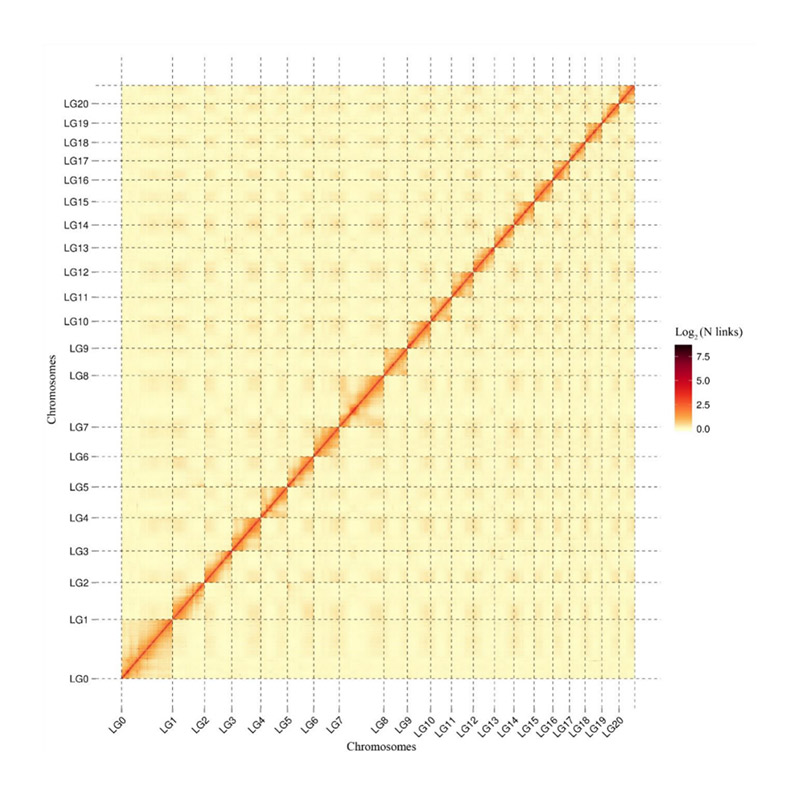
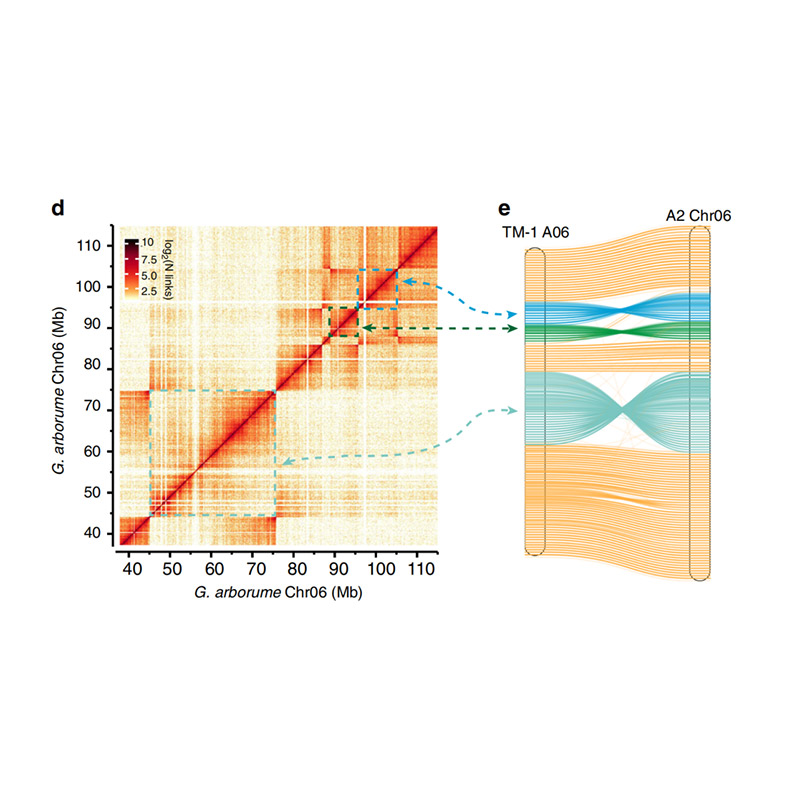
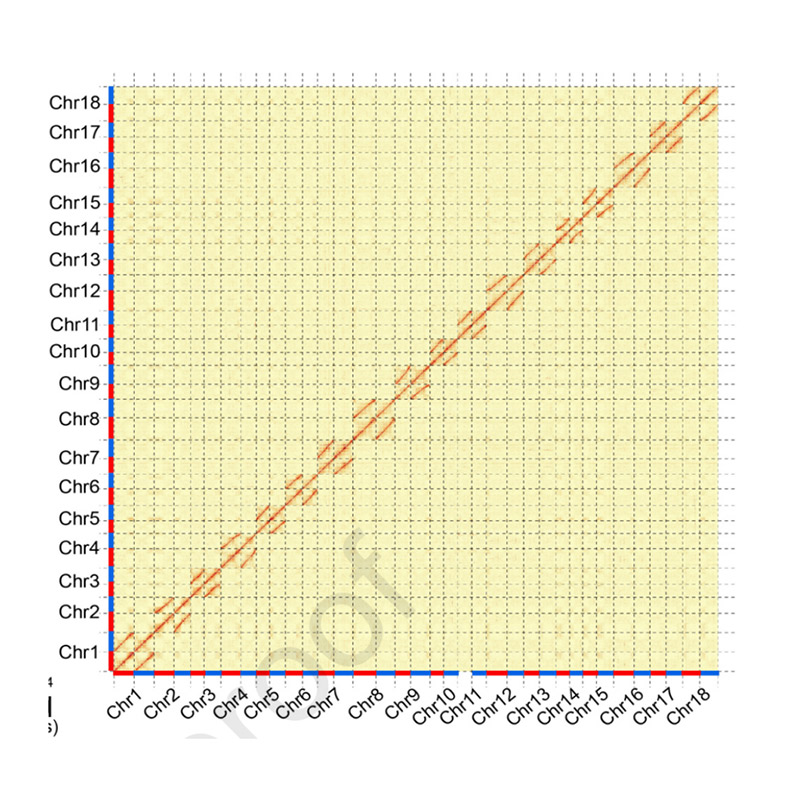
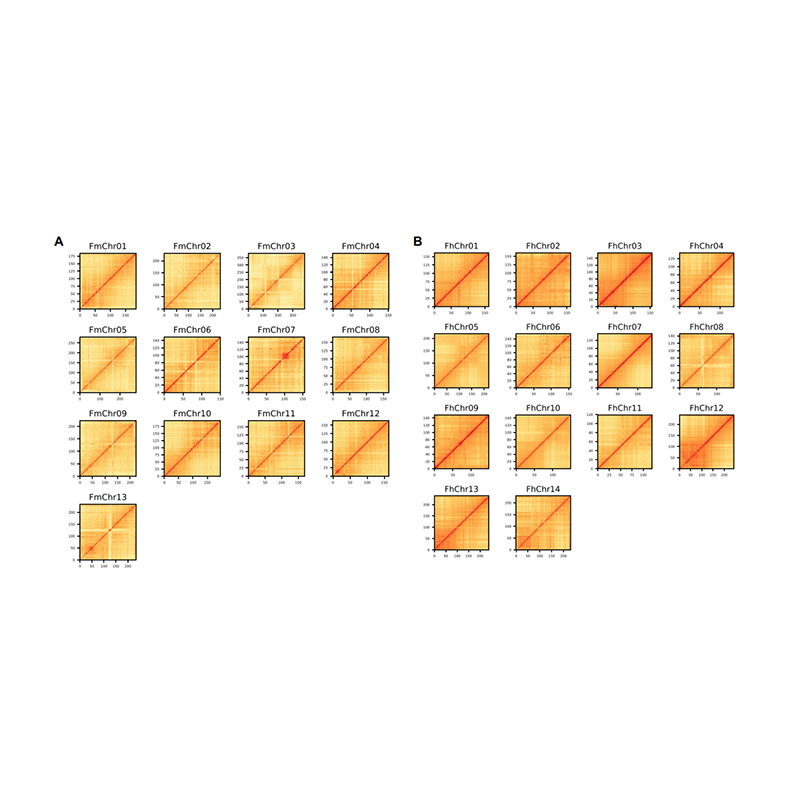
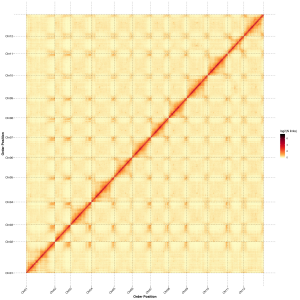
Bewertung nach der Assembly-Heatmap der Signalintensität zwischen Behältern
Untersuchen Sie die Fortschritte, die durch die HI-C-Versammlungsdienste von BMKgene durch eine kuratierte Sammlung von Veröffentlichungen erleichtert wurden.
Tian, T. et al. (2023) "Genomansammlung und genetische Dissektion eines prominenten dürreresistenten Maiskeimplasmas", Naturgenetik 2023 55: 3, 55 (3), S. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'Eine Versammlung von Chromosomen im Maßstab des asiatischen Honeybee-Apis-Cerana-Genoms', Grenzen in Genetik, 11, p. 524140. Doi: 10.3389/fgene.2020.00279/bibtex.
F. et al. (2023) „Aufschluss über die Evolution der Tropane -Alkaloid -Biosynthese durch Analyse von zwei Genomen in der Solanaceae -Familie“, Nature Communications 2023 14: 1, 14 (1), S. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) 'Genome des Banyan-Baum- und Bestäuber-Wespen liefern Einblicke in die Fig-Wasp-Coevolution', Cell, 183 (4), S. 875-889.e17. doi: 10.1016/j.cell.2020.09.043