HI-C-basierte Chromatin-Interaktion auf der Basis
Servicefunktionen
● Sequenzierung auf Illumina Novaseq mit PE150.
● Der Service erfordert Gewebeproben anstelle von extrahierten Nukleinsäuren, um mit Formaldehyd zu vernetzen und die DNA-Protein-Wechselwirkungen zu sparen.
● Das Hi-C-Experiment beinhaltet eine Einschränkung und die Endreparatur der klebrigen Enden mit Biotin, gefolgt von einer Kreislauf der resultierenden stumpfen Enden, während die Wechselwirkungen erhalten bleiben. Die DNA wird dann mit Streptavidin -Perlen heruntergezogen und für die nachfolgende Bibliotheksvorbereitung gereinigt.
Servicevorteile
●Optimale Beschränkungsenzymdesign: um eine hohe HI-C-Effizienz bei verschiedenen Arten mit bis zu 93% gültigen Interaktionspaaren zu gewährleisten.
●Umfangreiche Expertise und Veröffentlichungsunterlagen:BMKgene verfügt über umfangreiche Erfahrung mit> 2000 Hi-C-Sequenzierungsprojekten aus 800 verschiedenen Arten und verschiedenen Patenten. Über 100 veröffentlichte Fälle mit einem akkumulativen Einflussfaktor von über 900.
●Hochqualifiziertes Bioinformatik-Team:Mit internen Patenten und Software-Urheberrechten für HI-C-Experimente und Datenanalyse sowie einer selbst entwickelten Visualisierungsdatensoftware.
●Unterstützung nach dem Verkauf:Unser Engagement geht über den Abschluss des Projekts hinaus mit einer 3-monatigen Nachverkaufsdauer hinaus. Während dieser Zeit bieten wir Projekt-Follow-up, Fehlerbehebung und Q & A-Sitzungen an, um alle Abfragen im Zusammenhang mit den Ergebnissen zu beheben.
●Umfassende Annotation: Wir verwenden mehrere Datenbanken, um die Gene funktional mit identifizierten Variationen zu kommentieren und die entsprechende Anreicherungsanalyse durchzuführen.
Servicespezifikationen
| Bibliothek | Sequenzierungsstrategie | Empfohlene Datenausgabe | Hi-C-Signalauflösung |
| Hi-C-Bibliothek | Illumina PE150 | Chromatinschleife: 150x Tad: 50x | Chromatinschleife: 10 kb TAD: 40 KB |
Serviceanforderungen
| Probentyp | Erforderliche Menge |
| Tiergewebe | ≥2g |
| Vollblut | ≥2ml |
| Pilze | ≥1g |
| Pflanztes junges Gewebe | 1G/Aliquot, 2-4 Aliquoten empfohlen |
| Kultivierte Zellen | ≥1x107 |
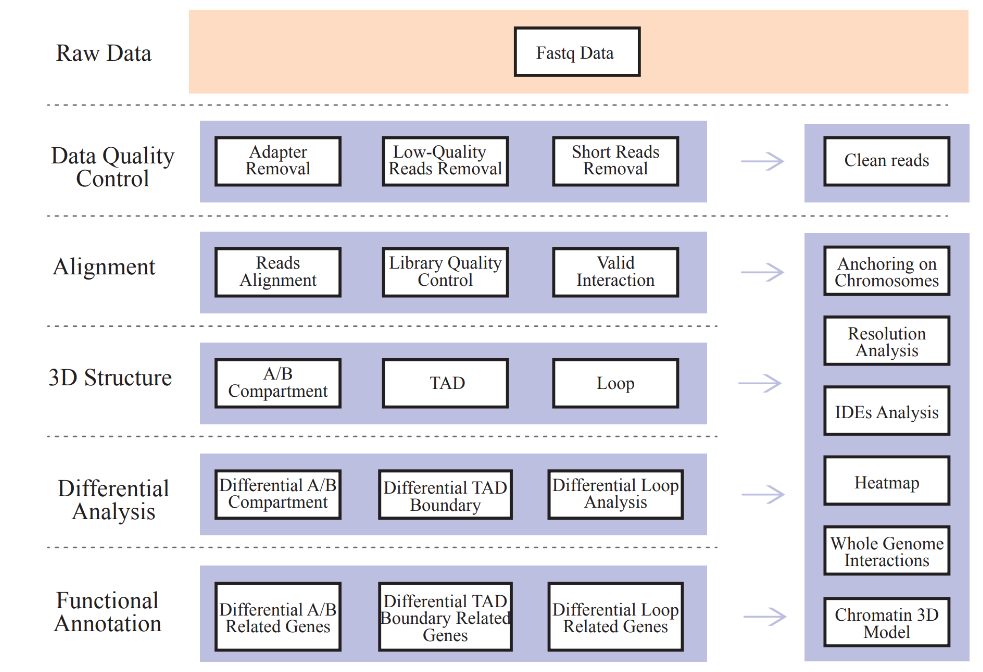
Enthält die folgende Analyse:
● Rohdaten QC;
● Mapping- und Hi-C-Bibliothek QC: Gültige Interaktionspaare und Interaktionsabfall-Exponenten (IDEs);
● Genomweite Interaktionsprofilerstellung: CIS/Trans-Analyse und HI-C-Interaktionskarte;
● Analyse der A/B -Kompartimentverteilung;
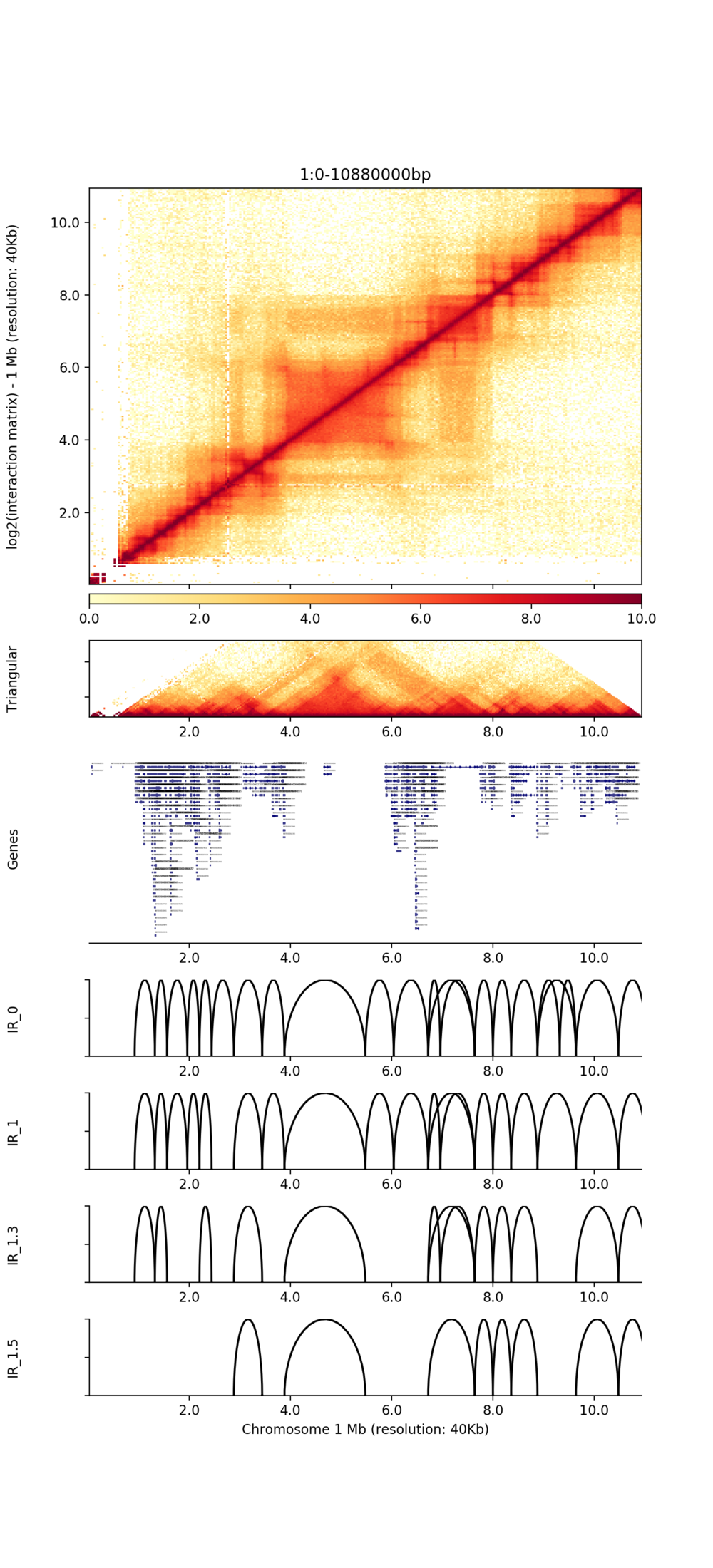
● Identifizierung von Tads und Chromatinschleifen;
● Differentialanalyse zu Elementen der 3D -Chromatinstruktur zwischen Proben und entsprechende funktionelle Annotation assoziierter Gene.
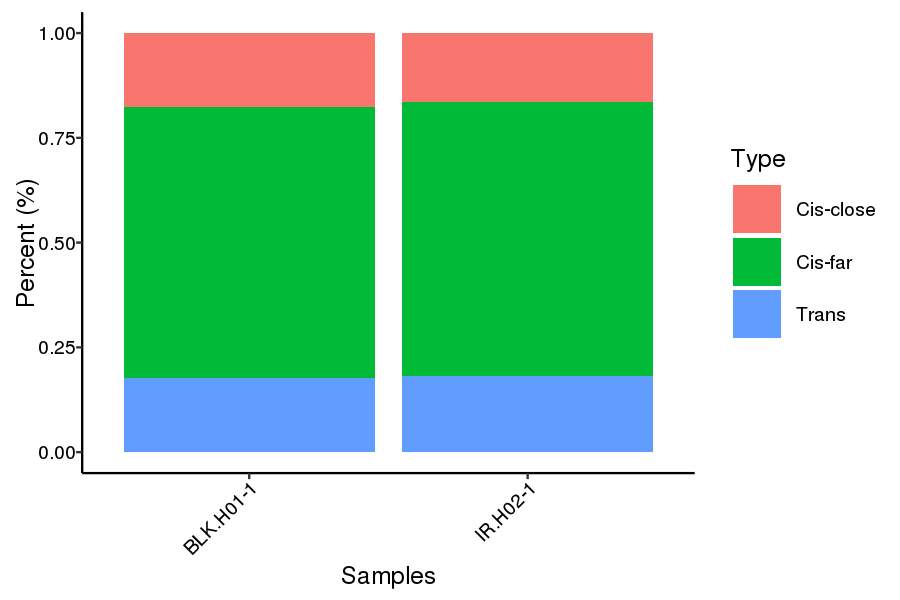
CIS- und Trans -Proportional -Verteilung
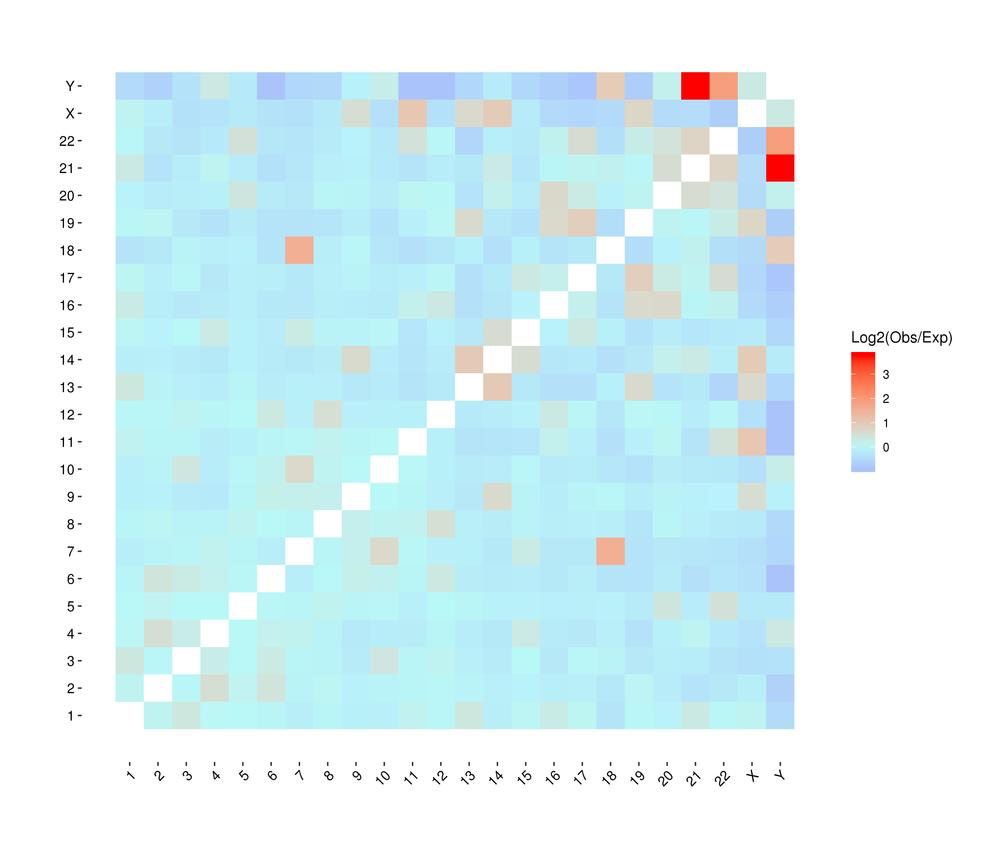
Wärmemap von chromosomalen Wechselwirkungen zwischen Proben
Genomweite Verteilung von A/B-Kompartimenten
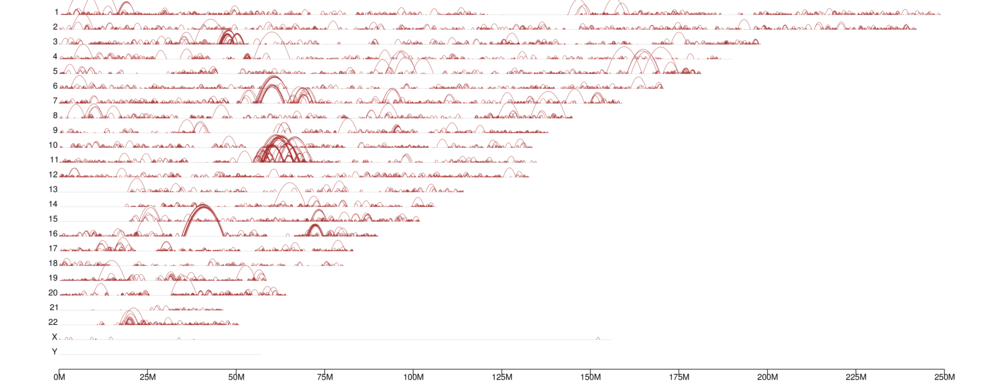
Genomweite Verteilung von Chromatinschleifen
Visualisierung von Tads
Untersuchen Sie die Forschungsergebnisse, die durch die HI-C-Sequenzierungsdienste von BMKgene durch eine kuratierte Sammlung von Veröffentlichungen erleichtert wurden.
Meng, T. et al. (2021) 'Eine vergleichende integrierte Multi-OMICS-Analyse identifiziert CA2 als neuartiges Ziel für das Chordom.',Neuroonkologie23 (10), S. 1709–1722. doi: 10.1093/neuonc/noab156.
Xu, L. et al. (2021) '3D-Desorganisation und Umlagerung von Genom liefern Einblicke in die Pathogenese der NAFLD durch integrierte HI-C-, Nanopore- und RNA-Sequenzierung',Acta Pharmaceutica Sinica B, 11 (10), S. 3150–3164. doi: 10.1016/j.apsb.2021.03.022.