Genomweite Assoziationsanalyse
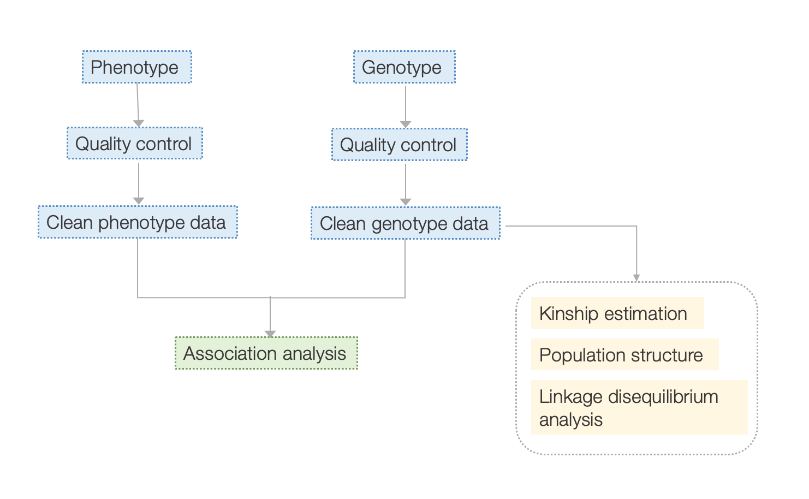
Arbeitsablauf

Servicevorteile
●Umfangreiche Expertise und Publikationsaufzeichnungen: Mit der gesammelten Erfahrung in GWAS hat BMKGene Hunderte von Artenprojekten in der Populations-GWAS-Forschung abgeschlossen, Forscher bei der Veröffentlichung von mehr als 100 Artikeln unterstützt und der kumulative Impact-Faktor erreichte 500.
● Umfassende bioinformatische Analyse: Der Arbeitsablauf umfasst die SNP-Merkmals-Assoziationsanalyse, die Bereitstellung einer Reihe von Kandidatengenen und ihrer entsprechenden funktionalen Annotation.
●Hochqualifiziertes Bioinformatik-Team und kurze Analysezyklen: Mit großer Erfahrung in der fortgeschrittenen Genomanalyse liefert das Team von BMKGene umfassende Analysen mit kurzer Bearbeitungszeit.
●Post-Sales-Support:Unser Engagement geht über den Projektabschluss hinaus und umfasst einen dreimonatigen Kundendienstzeitraum. Während dieser Zeit bieten wir Projektnachverfolgung, Unterstützung bei der Fehlerbehebung und Frage-und-Antwort-Sitzungen an, um alle Fragen zu den Ergebnissen zu beantworten.
Leistungsbeschreibung und Anforderungen
| Art der Sequenzierung | Empfohlene Bevölkerungsskala | Sequenzierungsstrategie | Nukleotidanforderungen |
| Sequenzierung des gesamten Genoms | 200 Proben | 10x | Konzentration: ≥ 1 ng/µL Gesamtmenge ≥ 30 ng Begrenzte oder keine Verschlechterung oder Kontamination |
| Specific-Locus Amplified Fragment (SLAF) | Tag-Tiefe: 10x Anzahl der Tags: < 400 MB: WGS wird empfohlen < 1 GB: 100.000 Tags 1 GB > 2 GB: 300.000 Tags Maximal 500.000 Tags | Konzentration ≥ 5 ng/µL Gesamtmenge ≥ 80 ng Nanodrop OD260/280=1,6-2,5 Agarosegel: kein oder begrenzter Abbau oder Kontamination
|
Materialauswahl



Verschiedene Sorten, Unterarten, Landrassen/Genbanken/gemischte Familien/Wildressourcen
Verschiedene Sorten, Unterarten, Landrassen
Halbgeschwisterfamilie/Vollgeschwisterfamilie/wilde Ressourcen
Service-Workflow

Experimentdesign

Musterlieferung

RNA-Extraktion

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
Beinhaltet die folgende Analyse:
- Genomweite Assoziationsanalyse: LM-, LMM-, EMMAX-, FASTLMM-Modell
- Funktionelle Annotation von Kandidatengenen
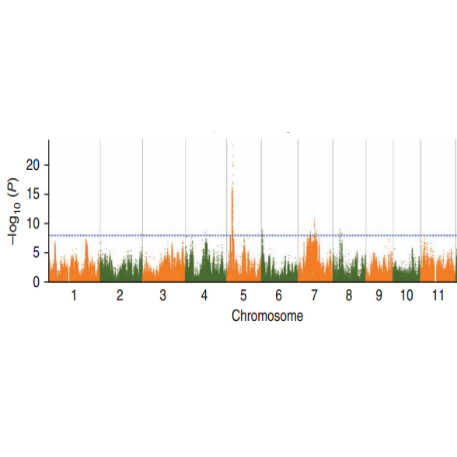
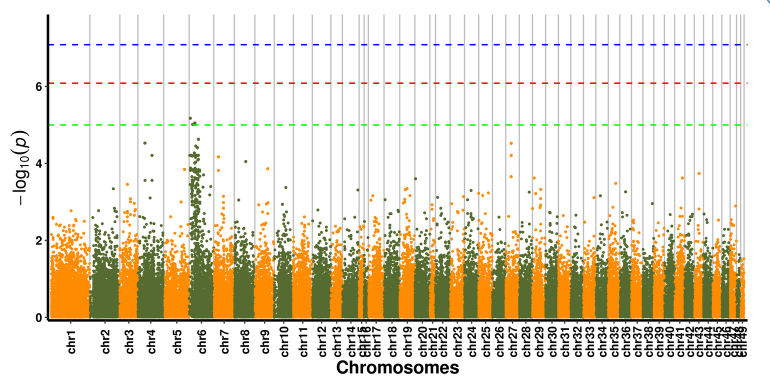
SNP-Merkmals-Assoziationsanalyse – Manhattan-Diagramm
SNP-Merkmals-Assoziationsanalyse – QQ-Diagramm
Entdecken Sie die Fortschritte, die durch die de GWAS-Dienste von BMKGene ermöglicht werden, anhand einer kuratierten Sammlung von Veröffentlichungen:
Lv, L. et al. (2023) „Einblick in die genetischen Grundlagen der Ammoniaktoleranz bei der Schwertmuschel Sinonovacula constricta durch genomweite Assoziationsstudie“,Aquakultur, 569, S. 739351. doi: 10.1016/J.AQUACULTURE.2023.739351.
Li, X. et al. (2022) „Multi-Omics-Analysen von 398 Fuchsschwanzhirse-Akzessionen zeigen Genomregionen, die mit Domestizierung, Metabolitenmerkmalen und entzündungshemmenden Wirkungen verbunden sind“,Molekulare Pflanze, 15(8), S. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) „Genome-Wide Association Mapping of Hulless Barely Phenotypes in Dürreumgebung“,Grenzen der Pflanzenwissenschaft, 13, S. 924892. doi: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) „GmST1, das eine Sulfotransferase kodiert, verleiht Resistenz gegen die Sojabohnenmosaikvirusstämme G2 und G3“,Pflanze, Zelle und Umwelt, 44(8), S. 2777–2792. doi: 10.1111/PCE.14066.