De -novo -Pilzgenomanordnung
Servicefunktionen
Mit drei möglichen Optionen zur Auswahl abhängig vom gewünschten Grad der Genomfotografie:
● Entwurf der Genomoption: Kurzread-Sequenzierung mit Illumina Novaseq PE150.
● Option für Pilzfeingenom:
Genome Survey: Illumina Novaseq PE150.
Genomanordnung: Pacbio Revio (HiFi liest) oder Nanopore -Promethion 48.
● Pilzgenom auf Chromosomenebene:
Genome Survey: Illumina Novaseq PE150.
Genomanordnung: Pacbio Revio (HiFi liest) oder Nanopore -Promethion 48.
Anker mit Hi-C-Baugruppe.
Servicevorteile
●Mehrere Sequenzierungsstrategien verfügbar: Für unterschiedliche Forschungsziele und Anforderungen der Genomfotografie
●Komplette Bioinformatik -Workflow:Dies umfasst die Genomanordnung und die Vorhersage mehrerer genomischer Elemente, der Annotation des funktionellen Gens und der Verankerung von Contig.
●Umfangreiches Fachwissen: Mit über 12.000 mikrobiellen Genomen bringen wir mehr als ein Jahrzehnt Erfahrung, ein hochqualifiziertes Analyseteam, umfassende Inhalte und hervorragende Unterstützung nach dem Verkauf.
●Unterstützung nach dem Verkauf:Unser Engagement geht über den Abschluss des Projekts hinaus mit einer 3-monatigen Nachverkaufsdauer hinaus. Während dieser Zeit bieten wir Projekt-Follow-up, Fehlerbehebung und Q & A-Sitzungen an, um alle Abfragen im Zusammenhang mit den Ergebnissen zu beheben.
Servicespezifikationen
| Service | Sequenzierungsstrategie | Qualitätskontrolle |
| Entwurfsgenom | Illumina PE150 100x | Q30 ≥ 85% |
| Feines Genom | Genomumfrage: Illumina PE150 50 x Baugruppe: Pacbio Hifi 30x oder Nanopore 100x | Contig N50 ≥ 1MB (Pacbio Unceellular) Contig n50 ≥ 2 MB (ontt einzellulär) Contig N50 ≥500 kb (Andere) |
| Genom auf Chromosomenebene | Genomumfrage: Illumina PE150 50 x Baugruppe: Pacbio Hifi 30x oder Nanopore 100x Hi-C-Baugruppe 100x | Contig -Verankerungsverhältnis> 90%
|
Serviceanforderungen
| Konzentration (ng/µl) | Gesamtmenge (µg) | Volumen (ul) | OD260/280 | OD260/230 | |
| Pacbio | ≥20 | ≥2 | ≥20 | 1.7-2.2 | ≥ 1,6 |
| Nanopore | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1.0-3.0 |
| Illumina | ≥1 | ≥0,06 | ≥20 | - | - |
Einzelliger Pilz: ≥3,5 x 1010 Zellen
Makropilz: ≥ 10 g
Service Work Flow

Probenabgabe

Bibliothekskonstruktion

Sequenzierung

Datenanalyse

Nachverkaufsdienste
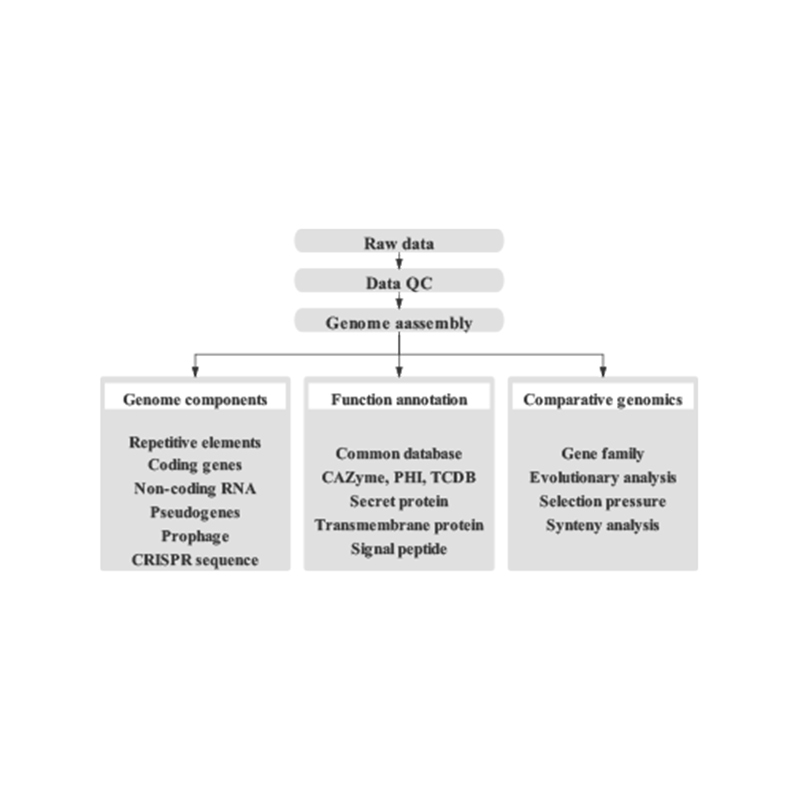
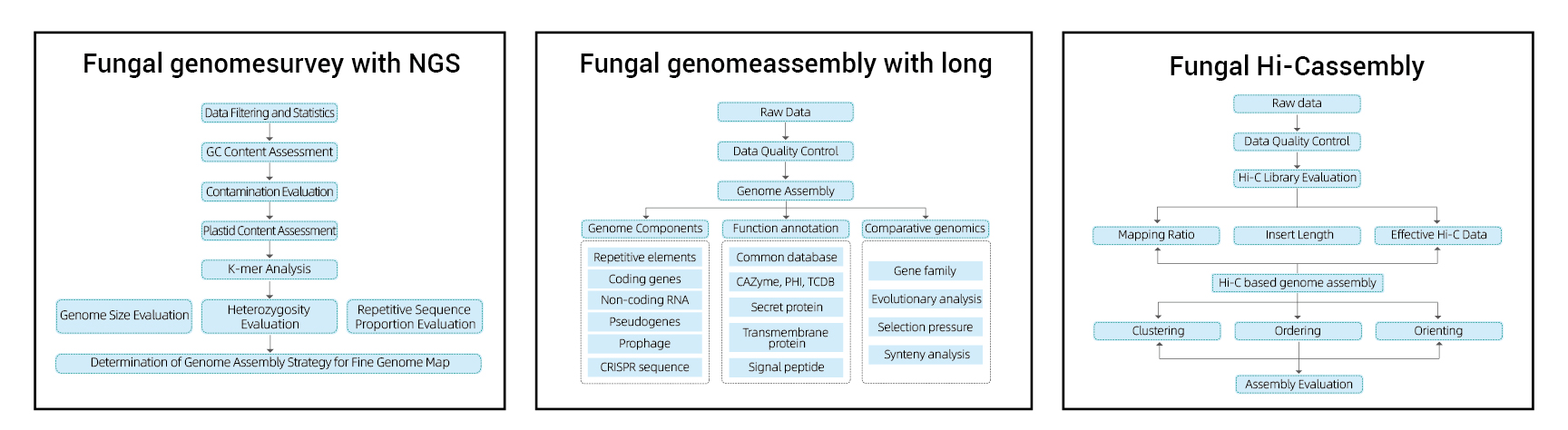
Enthält die folgende Analyse:
Genomumfrage:
- Datenqualitätsregelung der Datenqualität
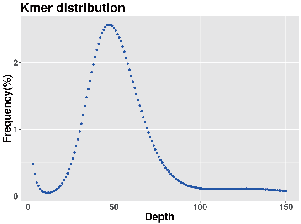
- Genomschätzung: Größe, Heterozygotie, sich wiederholende Elemente
Fine Genom Assembly:
- Datenqualitätsregelung der Datenqualität
- De novoMontage
- Analyse der Genomkomponenten: Vorhersage von CDs und mehreren genomischen Elementen
- Funktionale Annotation mit mehreren allgemeinen Datenbanken (Go, Kegg usw.) und erweiterten Datenbanken (Karte, VFDB usw.)
Hi-C-Baugruppe:
- HI-C-Bibliotheksbewertung.
- Contigs verankern das Clustering, die Bestellung und Orientierung
- Bewertung der HI-C-Assemblierung: Basierend auf Referenzgenom und Wärmekarte
Genomumfrage: K-MER-Verteilung
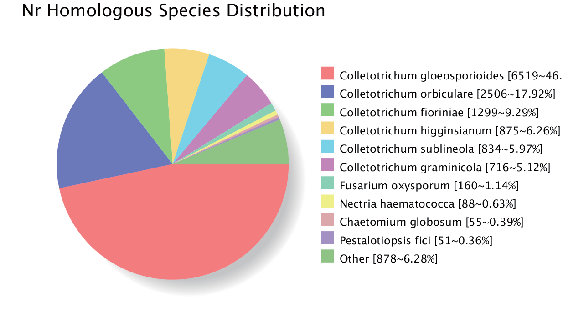
Genomassemblierung: Gen -Homologe Annotation (NR -Datenbank)
Genomanordnung: Funktionsgen Annotation (GO)
Untersuchen Sie die Fortschritte, die durch die Versammlungsdienste von BMKgene durch eine kuratierte Sammlung von Veröffentlichungen erleichtert wurden.
Hao, J. et al. (2023) 'Integrierte Omic -Profilerstellung des medizinischen Pilzpilzes inonotus obliquus unter untergetauchten Bedingungen',BMC -Genomik, 24 (1), S. 1–12. doi: 10.1186/s12864-023-09656-Z/Abbildungen/3.
Lu, L. et al. (2023) 'Genomsequenzierung zeigt die Evolution und die pathogenen Mechanismen des Weizenscharfen -Augenpot -Pathogen -Rhizoctonia cerealis',Das Erntejournal, 11 (2), S. 405–416. doi: 10.1016/j.cj.2022.07.024.
Zhang, H. et al. (2023) 'Genomressourcen für vier Clarireedia -Arten, die einen Dollarspot für verschiedene Rasengräse verursachen',Pflanzenerkrankung, 107 (3), S. 929–934. doi: 10.1094/pdis-08-22-1921-a
Zhang, SS et al. (2023) "genetischer und molekularer Hinweis auf ein tetrapolares Paarungssystem in der essbaren Pilz -Grifola -Frondosa",Journal of Fungi, 9 (10), p. 959. doi: 10.3390/jof9100959/s1.