Eukaryotische mRNA-Sequenzierung-NGS
Merkmale
● Einfangen von Poly-mRNA vor der Bibliotheksvorbereitung
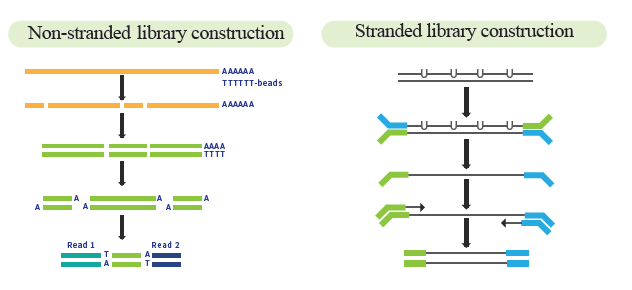
● Optionale direktionale mRNA-Bibliotheksvorbereitung, um den Erhalt strangspezifischer Sequenzierungsdaten zu ermöglichen
● Bioinformatische Analyse der Genexpression und Transkriptstruktur
Vorteile
●Umfangreiches Fachwissen: Unser Team hat bei BMKgene über 600.000 Proben verarbeitet, die verschiedene Probentypen wie Zellkulturen, Gewebe und Körperflüssigkeiten umfassen. Wir bringen einen großen Erfahrungsschatz in jedes Projekt ein und haben über 100.000 mRNA-Seq-Projekte in verschiedenen Forschungsbereichen erfolgreich abgeschlossen.
●Strenge Qualitätskontrolle: Wir implementieren zentrale Kontrollpunkte in allen Phasen, von der Proben- und Bibliotheksvorbereitung bis hin zur Sequenzierung und Bioinformatik. Diese sorgfältige Überwachung gewährleistet die Lieferung konstant hochwertiger Ergebnisse und gibt Ihnen die Gewissheit, dass Ihr Projekt in zuverlässigen Händen ist.
●Umfassende Anmerkung: Wir verwenden mehrere Datenbanken, um die differentiell exprimierten Gene (DEGs) funktional zu annotieren und entsprechende Anreicherungsanalysen durchzuführen. Dieser umfassende Ansatz bietet Einblicke in die zellulären und molekularen Prozesse, die der Transkriptomantwort zugrunde liegen, und stellt sicher, dass Sie umfassend über die Ergebnisse Ihres Projekts informiert sind.
●Post-Sales-Support: Unser Engagement geht über den Projektabschluss hinaus und umfasst einen 3-monatigen Kundendienstzeitraum. Während dieser Zeit bieten wir Projektnachverfolgung, Unterstützung bei der Fehlerbehebung und Frage-und-Antwort-Sitzungen an, um alle Fragen zu den Ergebnissen zu beantworten.
Musteranforderungen und Lieferung
| Bibliothek | Sequenzierungsstrategie | Daten empfohlen | Qualitätskontrolle |
| Poly A angereichert | Illumina PE150 DNBSEQ-T7 | 6–10 GB | Q30≥85 % |
Probenanforderungen:
Nukleotide:
| Konz. (ng/μl) | Menge (μg) | Reinheit | Integrität | |
| Standardbibliothek | ≥ 10 | ≥ 0,2 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Auf dem Gel wird nur eine begrenzte oder keine Protein- oder DNA-Kontamination angezeigt. | Für Pflanzen: RIN≥4,0; Für Tiere: RIN≥4,5; 5,0≥28S/18S≥1,0; begrenzte oder keine Grundlinienhöhe |
| Richtungsbibliothek | ≥ 10 | ≥ 0,2 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Auf dem Gel wird nur eine begrenzte oder keine Protein- oder DNA-Kontamination angezeigt. | Für Pflanzen: RIN≥4,0; Für Tiere: RIN≥4,5; 5,0≥28S/18S≥1,0; begrenzte oder keine Grundlinienhöhe |
● Pflanzen:
Wurzel, Stamm oder Blütenblatt: 450 mg
Blatt oder Samen: 300 mg
Frucht: 1,2 g
●Tier:
Herz oder Darm: 300 mg
Eingeweide oder Gehirn: 240 mg
Muskel: 450 mg
Knochen, Haare oder Haut: 1g
● Arthropoden:
Insekten: 6g
Krebstiere: 300 mg
● Vollblut: 1 Tube
● Zellen: 106 Zellen
Empfohlene Musterlieferung
Behälter: 2 ml Zentrifugenröhrchen (Alufolie wird nicht empfohlen)
Probenbeschriftung: Gruppe+Replikation, z. B. A1, A2, A3; B1, B2, B3... ...
Lieferung:
1. Trockeneis: Proben müssen in Beutel verpackt und in Trockeneis vergraben werden.
2. RNA-Tischröhrchen: RNA-Proben können in RNA-Stabilisierungsröhrchen (z. B. RNAstable®) getrocknet und bei Raumtemperatur versendet werden.
Service-Workflow

Experimentdesign

Musterlieferung

RNA-Extraktion

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
Bioinformatik
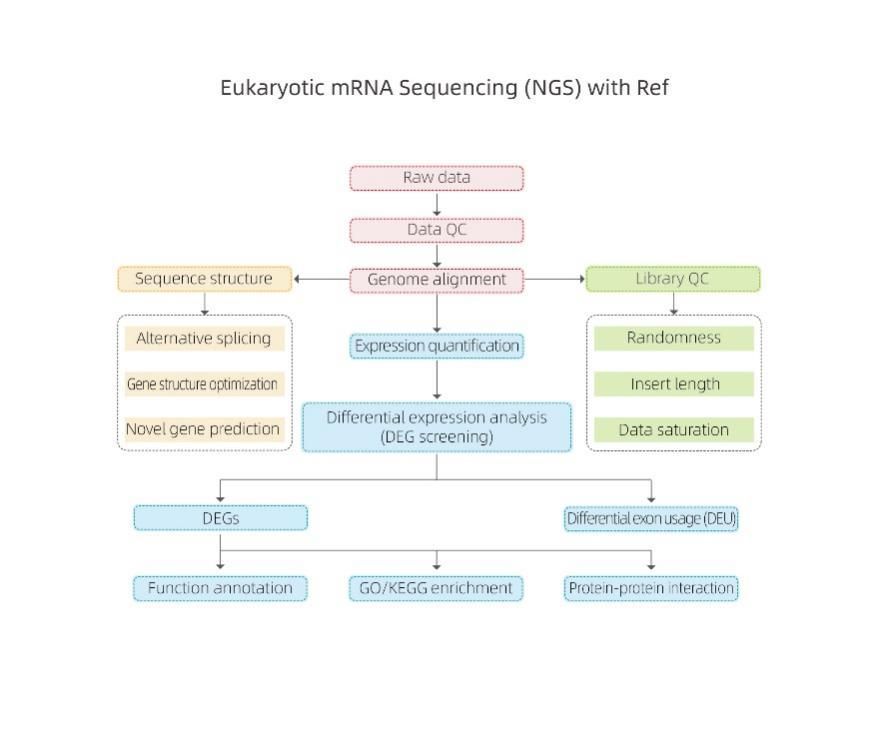
Eukaryotisch Arbeitsablauf zur mRNA-Sequenzierungsanalyse
Bioinformatik
ØQualitätskontrolle der Rohdaten
ØReferenzgenom-Alignment
ØAnalyse der Transkriptstruktur
ØExpressionsquantifizierung
ØDifferentialausdrucksanalyse
ØFunktionsannotation und -anreicherung
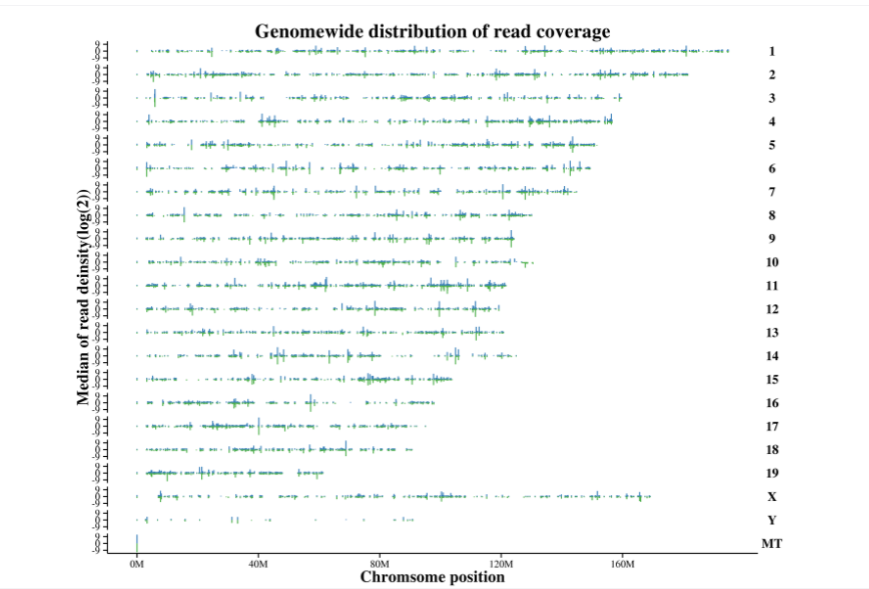
Referenz-Genomausrichtung
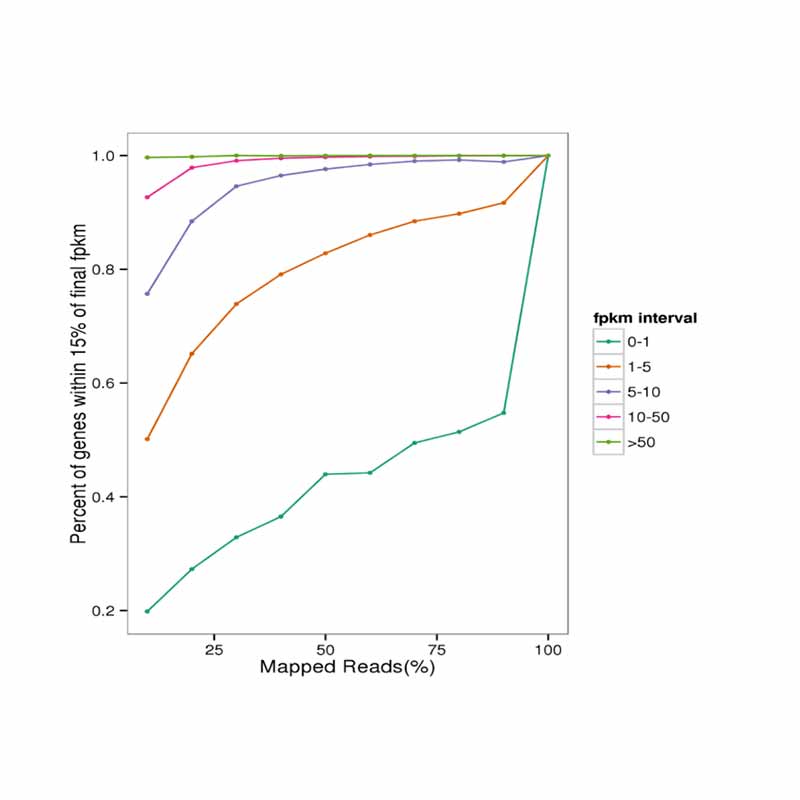
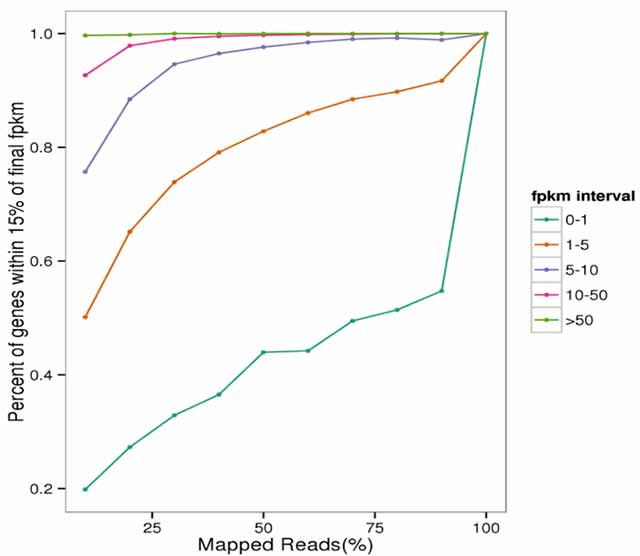
Datensättigung
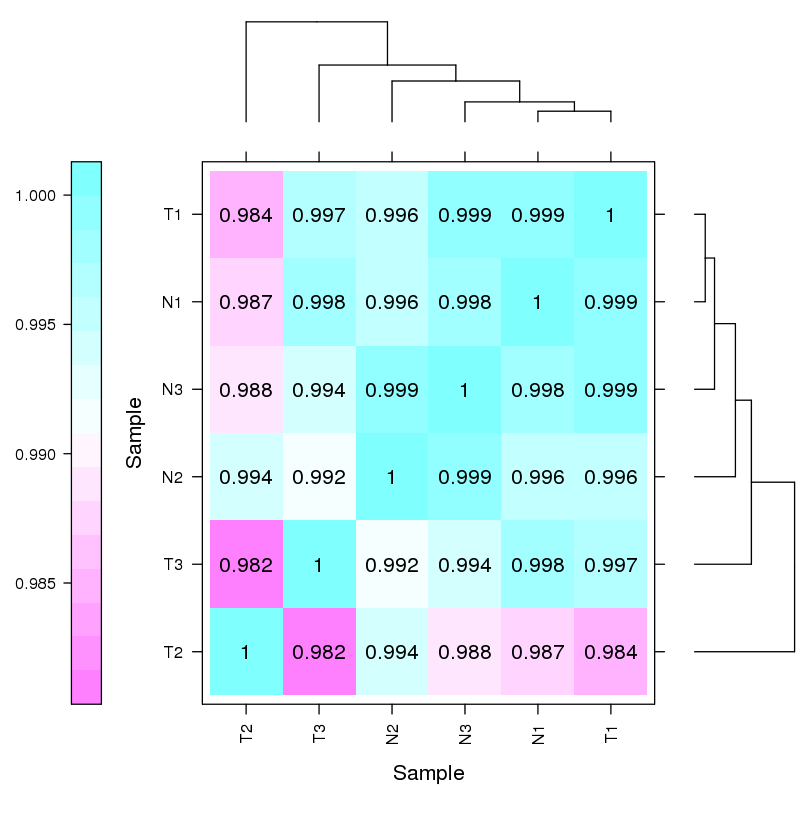
Probenkorrelation und Bewertung biologischer Replikate
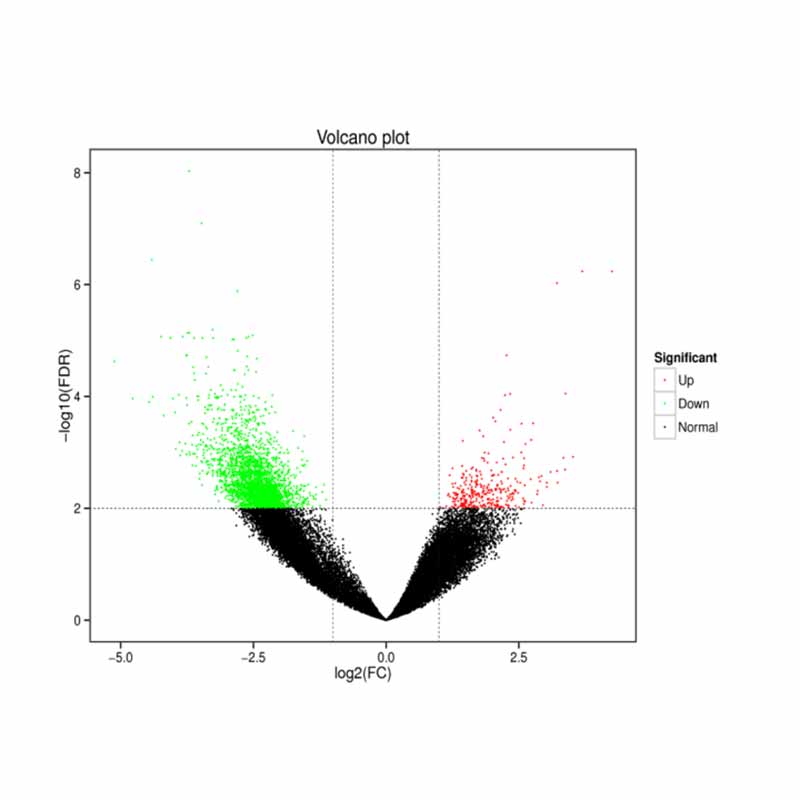
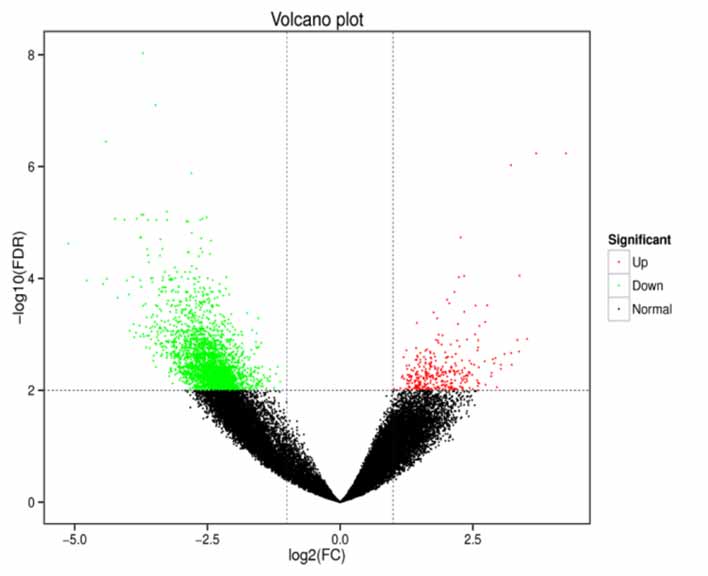
Differentiell exprimierte Gene (DEGs)
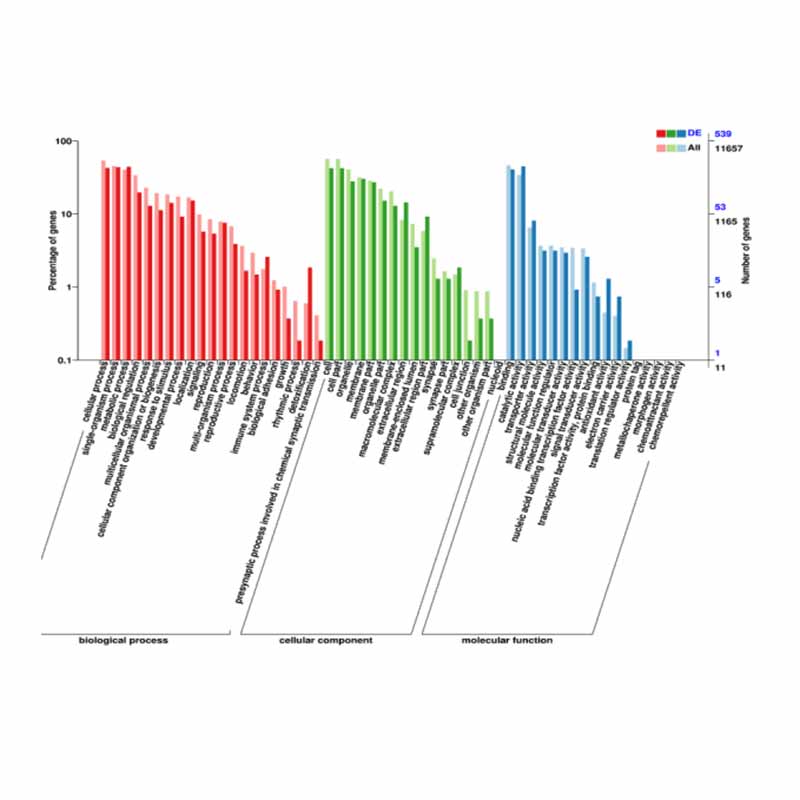
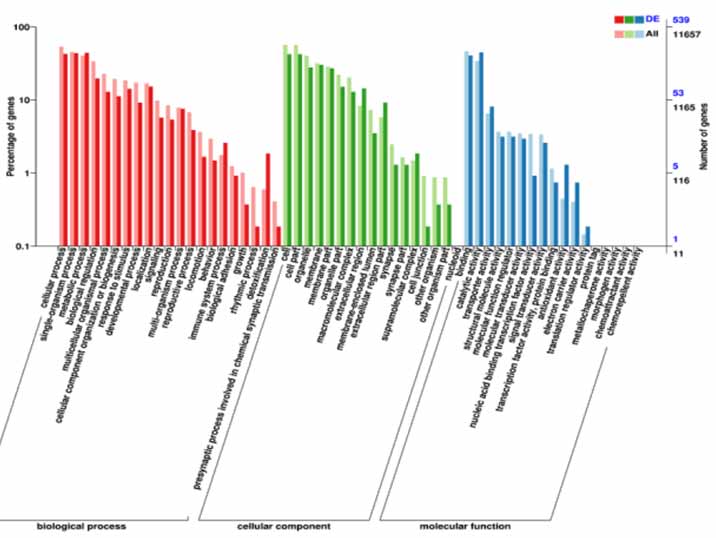
Funktionale Annotation von DEGs
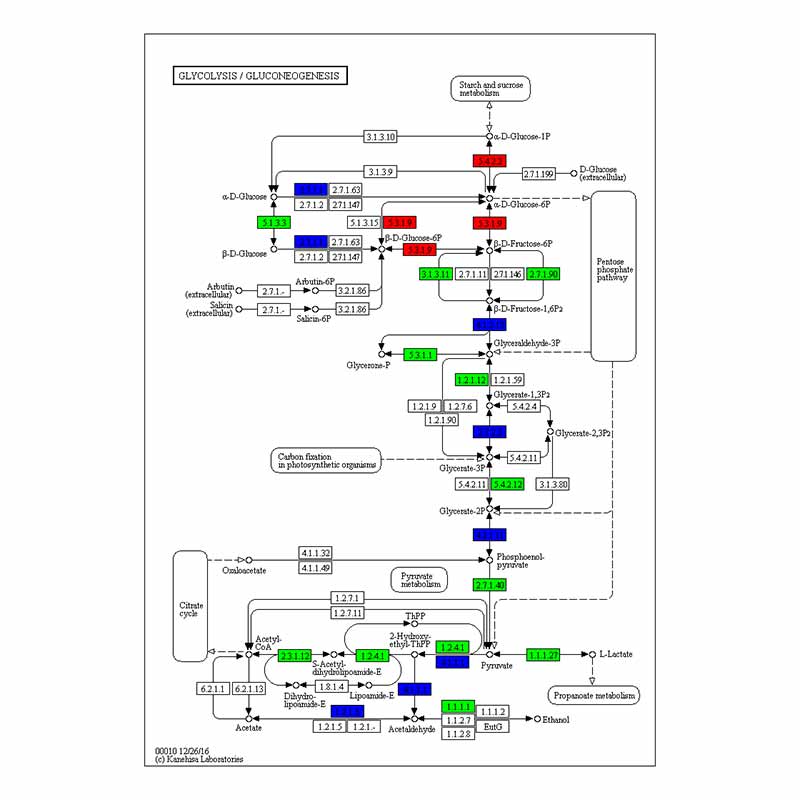
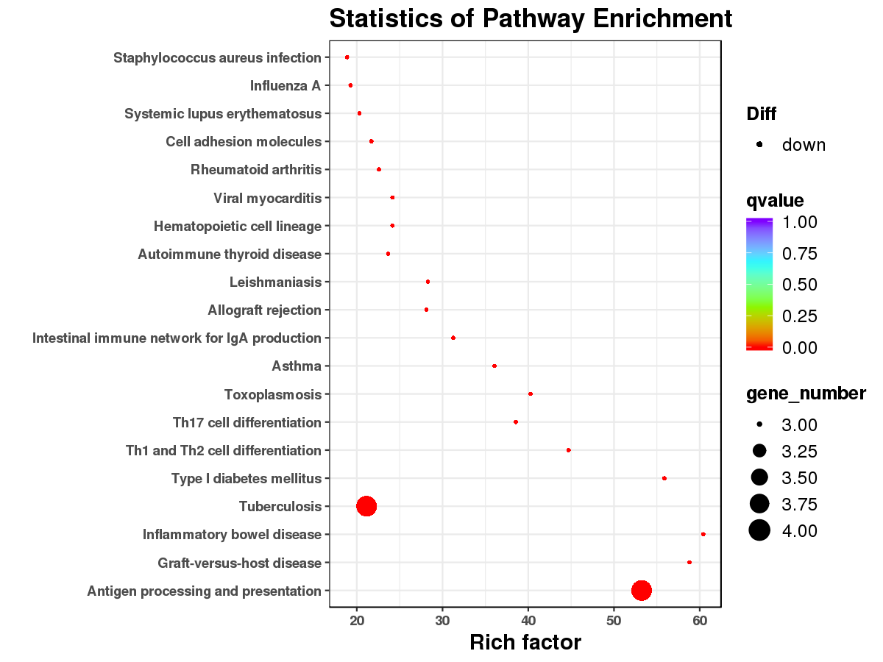
Funktionelle Anreicherung von DEGs
Entdecken Sie die Fortschritte, die durch die eukaryotischen NGS-mRNA-Sequenzierungsdienste von BMKGene ermöglicht werden, anhand einer kuratierten Sammlung von Veröffentlichungen.
Huang, L. et al. (2023) „Triclosan und Triclocarban schwächen die Geruchskapazität von Goldfischen, indem sie die Geruchsstofferkennung einschränken, die olfaktorische Signalübertragung stören und die olfaktorische Informationsverarbeitung stören“, Water Research, 233, S. 119736. doi: 10.1016/J.WATRES.2023.119736.
Jia, LJ et al. (2023) „Aspergillus fumigatus kapert menschliches p11, um pilzhaltige Phagosomen auf den nicht abbaubaren Weg umzuleiten“, Cell Host & Microbe, 31(3), S. 373-388.e10. doi: 10.1016/J.CHOM.2023.02.002.
Jin, K. et al. (2022) „TCP-Transkriptionsfaktoren, die an der Triebentwicklung von Ma-Bambus (Dendrocalamus latiflorus Munro) beteiligt sind“, Frontiers in Plant Science, 13, S. 884443. doi: 10.3389/FPLS.2022.884443/BIBTEX.
Wen, X. et al. (2022) „Das Chrysanthemum lavandulifolium-Genom und der molekulare Mechanismus, der verschiedenen Kapitulumtypen zugrunde liegt“, Horticulture Research, 9. doi: 10.1093/HR/UHAB022.
Zhang, Yujie et al. (2023) „Ein Kaskaden-Nanoreaktor zur Verbesserung der sonodynamischen Therapie bei Darmkrebs durch synergistische ROS-Verstärkung und Autophagieblockade“, Nano Today, 49, S. 101798. doi: 10.1016/J.NANTOD.2023.101798.