DNBSEQ Vorgefertigte Bibliotheken
Merkmale
●Plattform:MGI-DNBSEQ-T7
●Sequenzierungsmodi:PE150
●Übertragung von Illumina -Bibliotheken zuMgi:Aktivierung der Sequenzierung hoher Datenvolumina zu niedrigen Kosten.
●Qualitätskontrolle von Bibliotheken vor der Sequenzierung.
●Sequenzierungsdaten QC und Lieferung:Lieferung von QC -Bericht und Rohdaten im Fastq -Format nach Demultiplexing und Filterung von Q30.
Vorteile
●Vielseitigkeit der Sequenzierungsdienste:Der Kunde kann sich für die Sequenz nach Spur oder Datenmenge entscheiden.
●Hohe Datenausgabe:1500 GB/Spur
●Lieferung des Sequenzierungs -QC -Berichts:mit Qualitätsmetriken, Datengenauigkeit und Gesamtleistung des Sequenzierungsprojekts.
●Reifer Sequenzierungsprozess:mit kurzer Turn-Around-Zeit.
●Strenge Qualitätskontrolle: Wir implementieren strenge QC-Anforderungen, um die Bereitstellung konsequent hochwertiger Ergebnisse zu gewährleisten.
Stichprobenanforderungen
| Datenmenge (x) | Konzentration (qPCR/nm) | Volumen | |
| Teilspur
| X ≤ 10 GB | ≥ 1nm | ≥ 25 μl |
| 10 GB <x ≤ 50 GB | ≥ 2 nm | ≥ 25 μl | |
| 50 GB <x ≤ 100 GB | ≥ 3 nm | ≥ 25 μl | |
| X> 100 GB | ≥ 4 nm | ||
| einspurig | Pro Spur | ≥ 1,5 nm / Bibliothekspool | ≥ 25 μl / Bibliothekspool |
Zusätzlich zu Konzentration und Gesamtmenge ist auch ein geeignetes Spitzenmuster erforderlich.
HINWEIS: Die Fahrspursequenzierung von Bibliotheken mit niedriger Diversität erfordert Phix Spike-In, um eine robuste Basisanrufe zu gewährleisten.
Wir empfehlen, vorpoolte Bibliotheken als Beispiele einzureichen. Wenn Sie BMKgene benötigen, um Bibliothekspooling durchzuführen, lesen Sie bitte die
Bibliotheksanforderungen für eine teilweise Spursequenzierung.
Bibliotheksgröße (Peak Map)
Der Hauptpeak sollte innerhalb von 300-450 bp liegen.
Bibliotheken sollten einen einzelnen Hauptpeak, keine Adapterkontamination und keine Primer -Dimere haben.
Service Workflow




Bibliothek QC -Bericht
Ein Bericht über die Qualität der Bibliothek wird vor der Abfolge, der Bewertung der Bibliotheksmenge und der Fragmentierung bereitgestellt.
Sequenzierung QC -Bericht
Tabelle 1. Statistik zu Sequenzierungsdaten.
| Beispiel -ID | Bmkid | Rohe liest | Rohdaten (BP) | Clean Reads (%) | Q20 (%) | Q30 (%) | GC (%) |
| C_01 | BMK_01 | 22.870,120 | 6.861.036.000 | 96,48 | 99.14 | 94.85 | 36.67 |
| C_02 | BMK_02 | 14.717.867 | 4,415.360.100 | 96.00 | 98.95 | 93.89 | 37.08 |
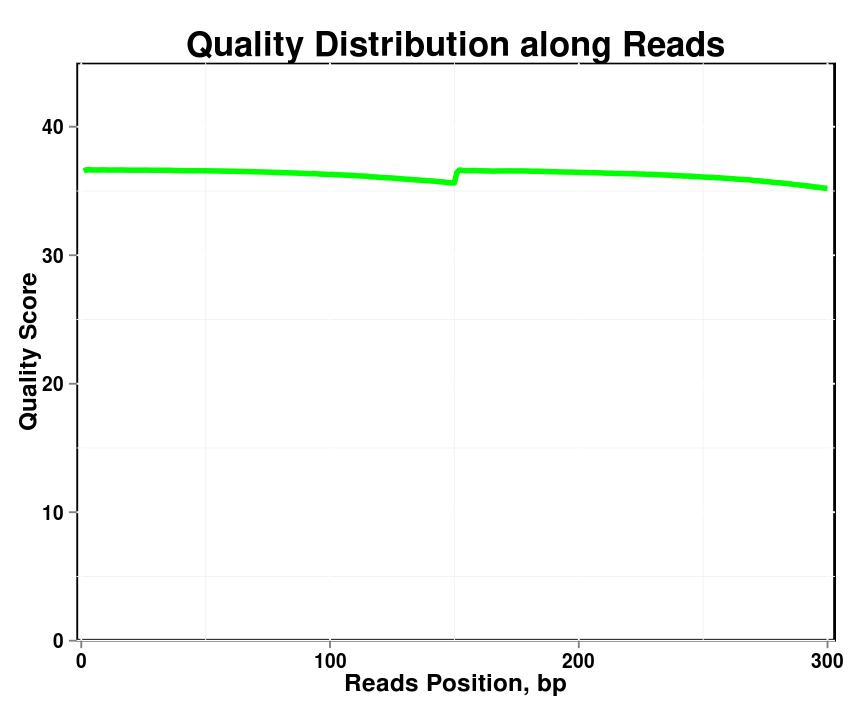
Abbildung 1. Qualitätsverteilung entlang der Lesevorgänge in jeder Probe
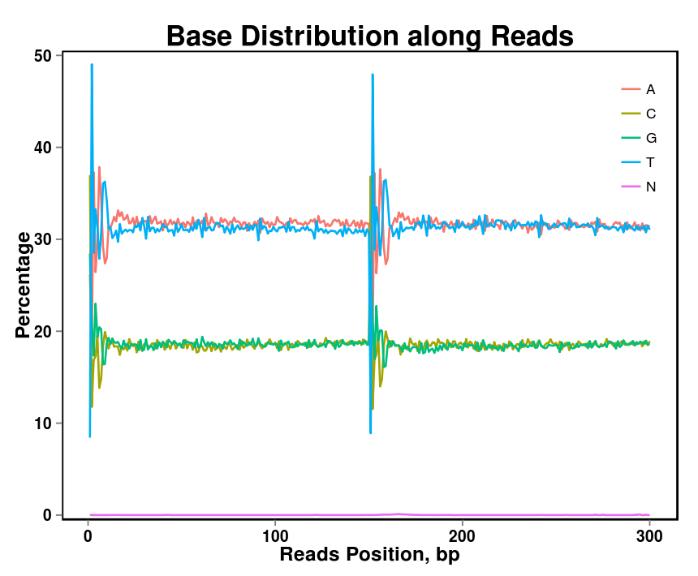
Abbildung 2. Basisinhaltsverteilung
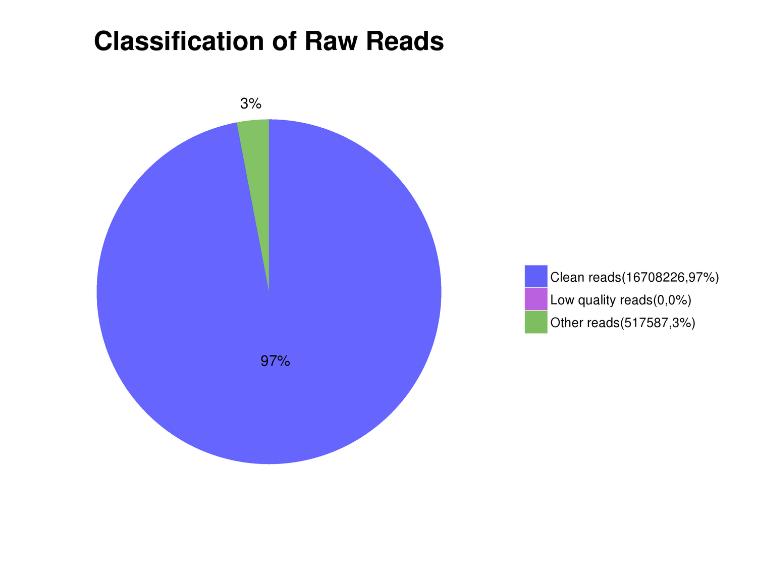
Abbildung 3.. Verteilung von Leseinhalten in Sequenzierungsdaten