16S/18S/ITS-Amplikonsequenzierung-PacBio
Servicefunktionen
● Sequenzierungsplattform: PacBio Revio
● Sequenzierungsmodus: CCS (HiFi liest)
● Amplifikation der Zielregion, gefolgt von einer Tandemverknüpfung der Amplifikate vor der Vorbereitung der HiFi-SMRT-Glockenbibliothek
Servicevorteile
●Höhere taxonomische Auflösung: THan Short-Amplicon-Sequenzierung,Dies ermöglicht höhere OTU-Klassifizierungsraten auf Artenebene.
●Hochpräzises Base-Calling: Sequenzierung im PacBio CCS-Modus (HiFi-Lesevorgänge).
●Isolationsfrei: Schnelle Identifizierung der mikrobiellen Zusammensetzung in Umweltproben.
●Weit verbreitet: Diverse mikrobielle Gemeinschaftsstudien.
●Umfassende bioinformatische Analyse: Das neueste QIIME2-Paket (quantitative Einblicke in die mikrobielle Ökologie) mit vielfältigen Analysen in Bezug auf Datenbank, Annotation, OTU/ASV.
●Umfangreiches Fachwissen: Mit Tausenden von jährlich durchgeführten Amplikon-Sequenzierungsprojekten bringt BMKGENE über ein Jahrzehnt Erfahrung, ein hochqualifiziertes Analyseteam, umfassende Inhalte und exzellenten Post-Sales-Support mit.
Leistungsbeschreibung
| Bibliothek | Sequenzierungsstrategie | Daten empfohlen |
| Amplikon | PacBio Revio | 10/30/50 K-Tags (CCS) |
Beispielanforderungen
| Konzentration (ng/µL) | Gesamtmenge (µg) | Volumen (µL) |
| ≥5 | ≥0,3 | ≥20 |
Empfohlene Musterlieferung
Frieren Sie die Proben 3-4 Stunden lang in flüssigem Stickstoff ein und lagern Sie sie zur Langzeitkonservierung in flüssigem Stickstoff oder bei -80 Grad. Der Probenversand mit Trockeneis ist erforderlich.
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
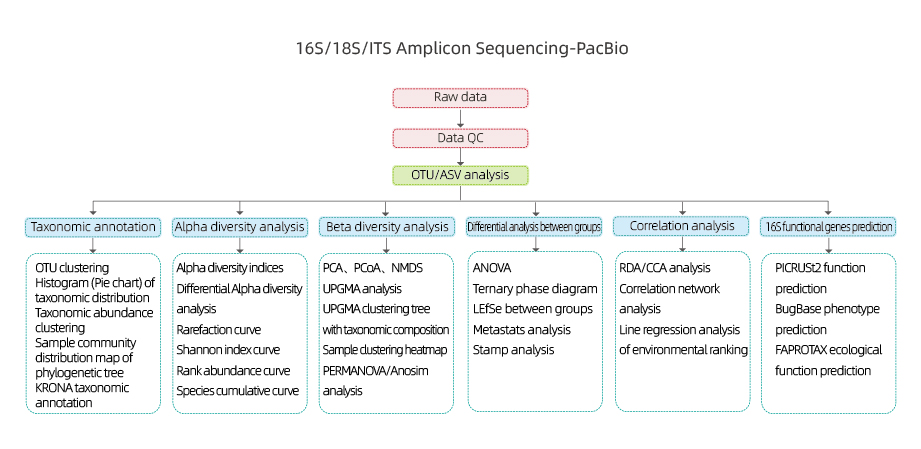
Beinhaltet die folgende Analyse:
●Qualitätskontrolle der Rohdaten
●OTU-Clustering/De-Noise (ASV)
●OTU-Anmerkung
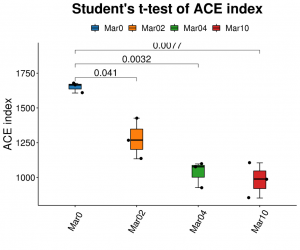
●Alpha-Diversitätsanalyse: mehrere Indizes, einschließlich Shannon, Simpson und ACE.
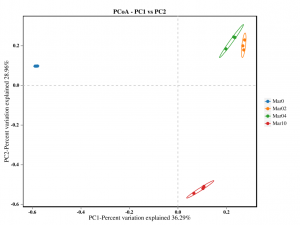
●Beta-Diversity-Analyse
●Gruppenübergreifende Analyse
●Korrelationsanalyse: zwischen Umweltfaktoren und OUT-Zusammensetzung und -Vielfalt
●Vorhersage funktioneller 16S-Gene
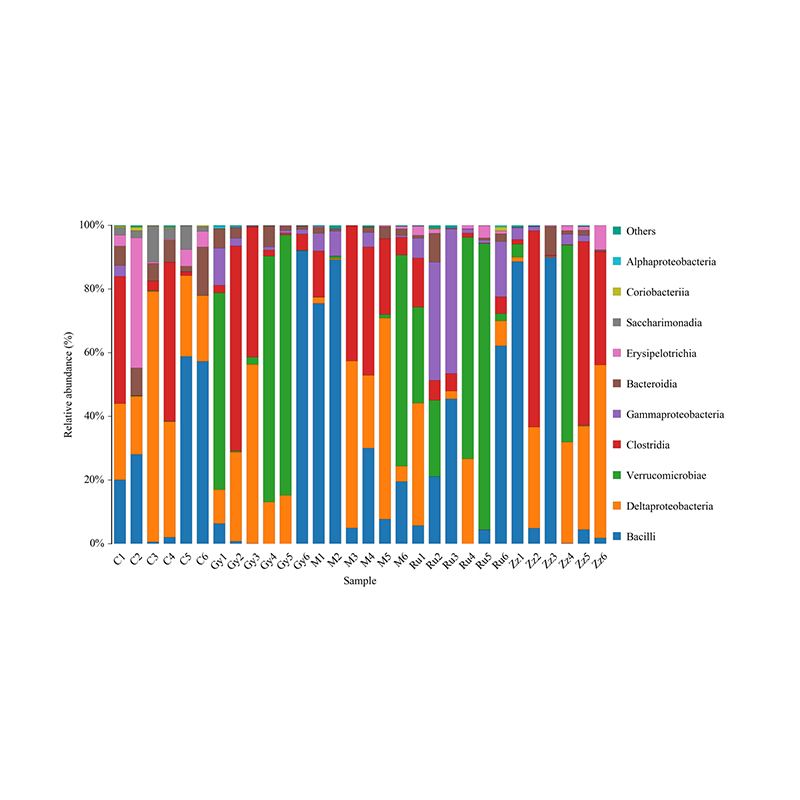
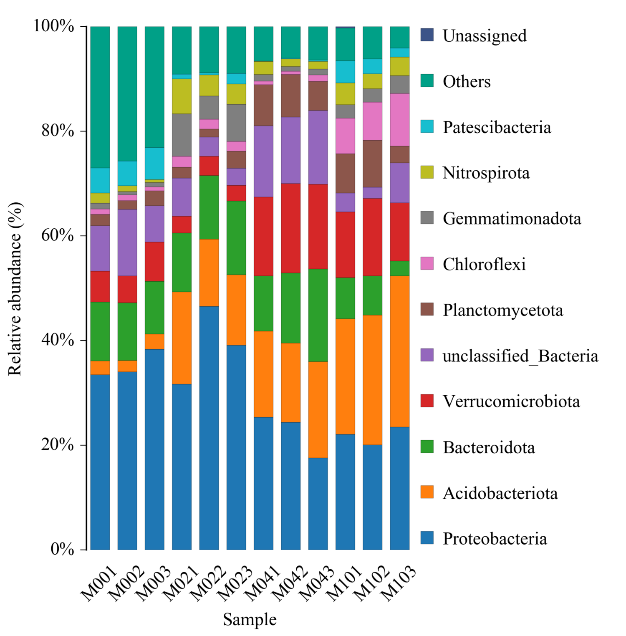
Histogramm der taxonomischen Verteilung
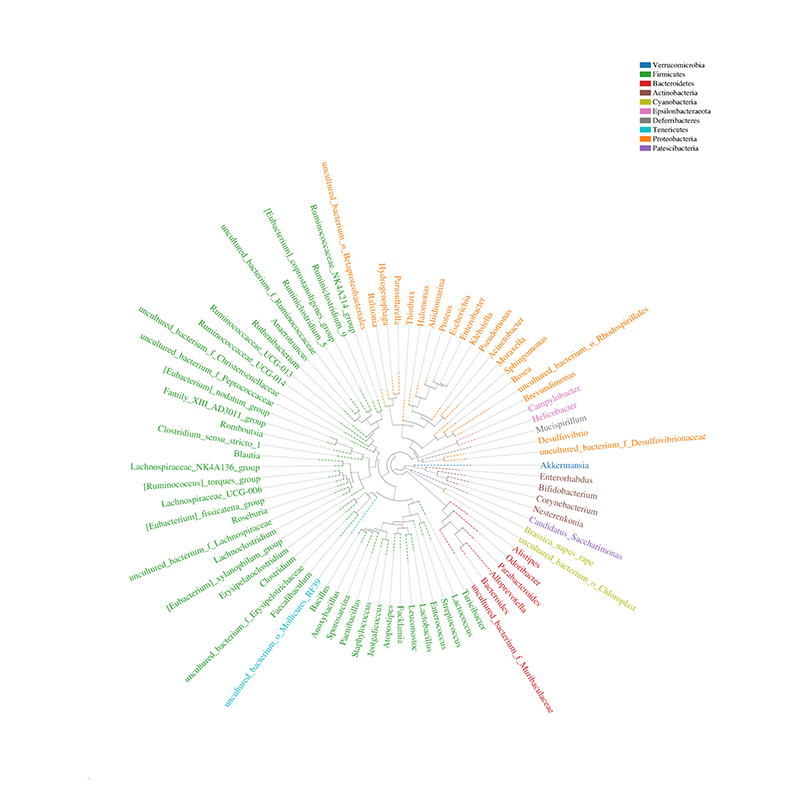
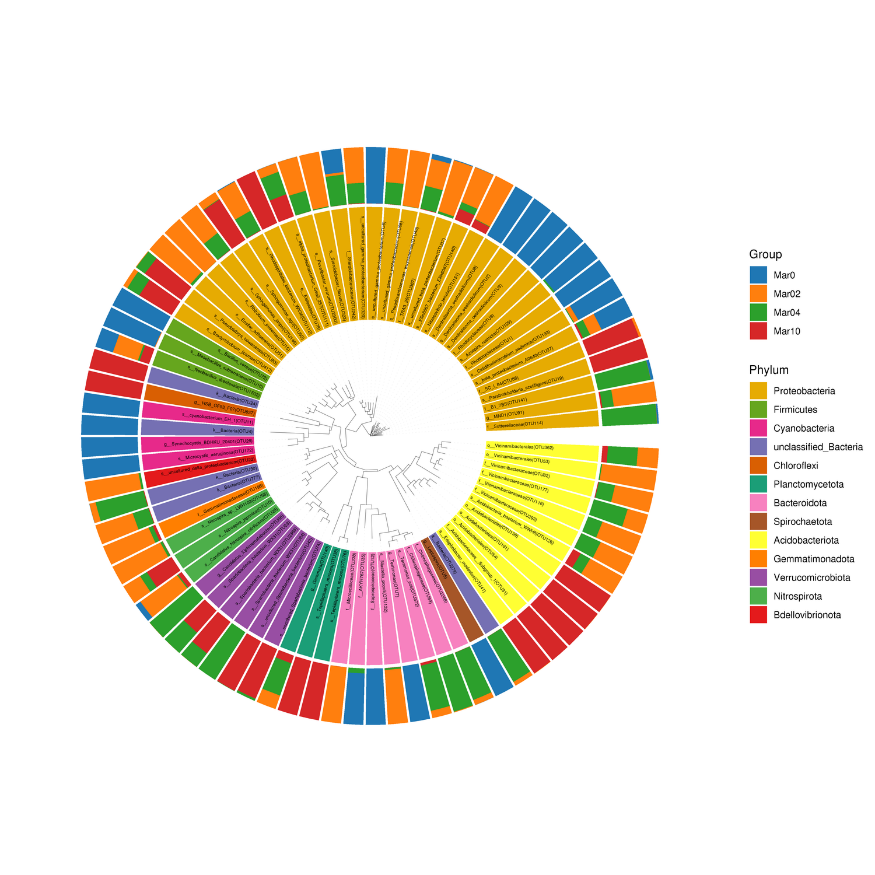
Stammbaum der Gemeinschaftsverbreitung
Alpha-Diversitätsanalyse: ACE
Beta-Diversitätsanalyse: PCoA
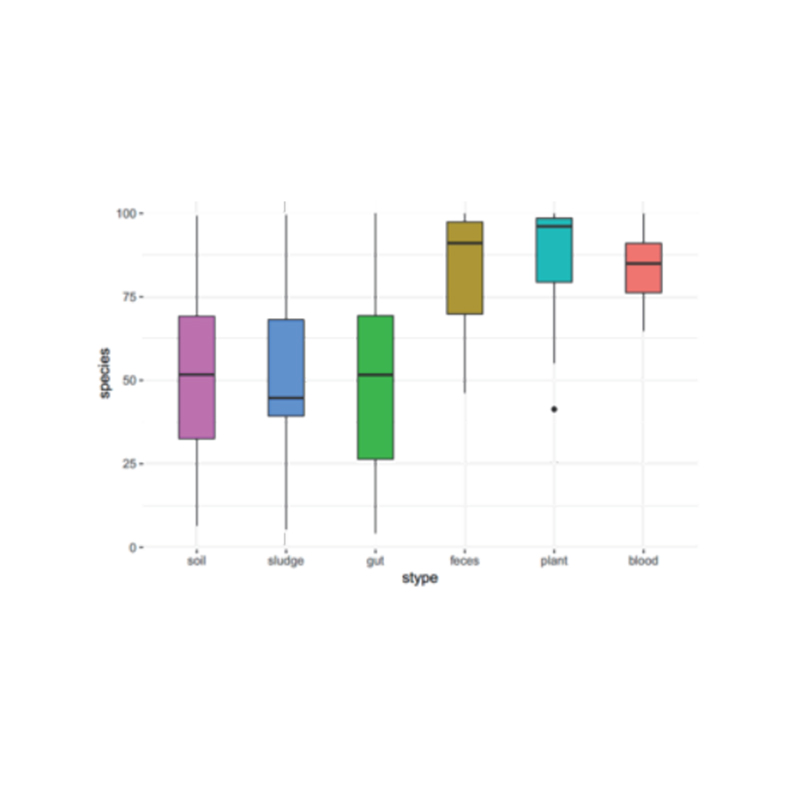
Intergruppenanalyse: ANOVA
Entdecken Sie die Fortschritte, die durch die Amplikon-Sequenzierungsdienste von BMKGene mit PacBio ermöglicht werden, anhand einer kuratierten Sammlung von Veröffentlichungen.
Gao, 1), S. 16. doi: 10.3390/FERMENTATION9010016/S1.
Li, S. et al. (2023) „Erfassung der mikrobiellen dunklen Materie in Wüstenböden mithilfe kulturomischer Metagenomik und hochauflösender Analyse“, npj Biofilms and Microbiomes 2023 9:1, 9(1), S. 1–14. doi: 10.1038/s41522-023-00439-8.
Mu, L. et al. (2022) „Auswirkungen von Fettsäuresalzen auf Fermentationseigenschaften, Bakterienvielfalt und aerobe Stabilität von Mischsilage aus Luzerne, Reisstroh und Weizenkleie“, Journal of the Science of Food and Agriculture, 102(4), S. 1475– 1487. doi: 10.1002/JSFA.11482.
Yang, J. et al. (2023) „Die Wechselwirkung zwischen Biomarkern für oxidativen Stress und Darmmikrobiota bei der antioxidativen Wirkung von Extrakten aus Sonchus brachyotus DC.“ in Oxazolone-Induced Intestinal Oxidative Stress in Adult Zebrafish“, Antioxidants 2023, Bd. 12, Seite 192, 12(1), S. 192. doi: 10.3390/ANTIOX12010192.