10x Genomics Visium räumliches Transkriptom
Technisches Programm

Merkmale
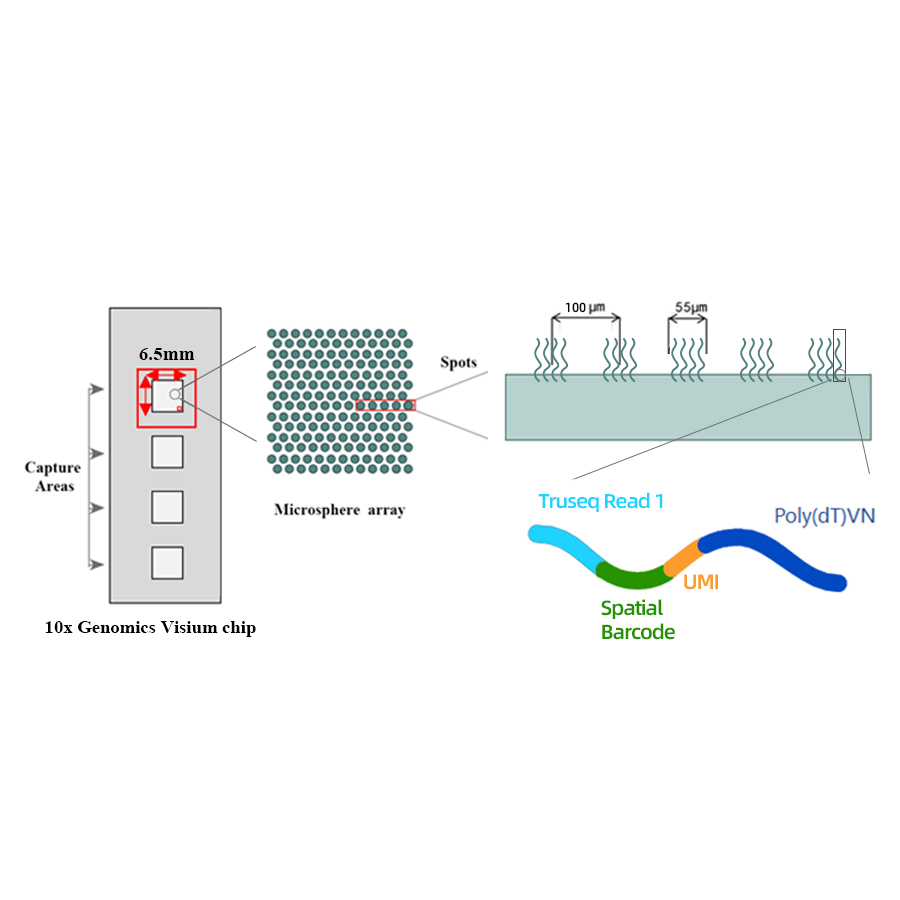
● Auflösung: 100 µm
● Spotdurchmesser: 55 µm
● Anzahl der Stellen: 4992
● Erfassungsbereich: 6,5 x 6,5 mm
● Jeder barcodierte Punkt ist mit Primern beladen, die aus 4 Abschnitten bestehen:
- Poly (DT) -Sschwanz für mRNA -Priming und cDNA -Synthese
- Einzigartige molekulare Kennung (UMI), um die Verstärkungsverzerrung zu korrigieren
- Räumlicher Barcode
- Bindungssequenz des partiellen Les 1 -Sequenzierungsprimers
● H & E -Färbung von Abschnitten
Vorteile
●One-Stop-Service: Integriert alle Erfahrungen und Fähigkeiten basierende Schritte, einschließlich Kryoabschnitt, Färbung, Gewebeoptimierung, räumlicher Barcodierung, Bibliotheksvorbereitung, Sequenzierung und Bioinformatik.
● Hochqualifiziertes technisches Team: Mit Erfahrung in über 250 Gewebetypen und mehr als 100 Arten, einschließlich Mensch, Maus, Säugetier, Fisch und Pflanzen.
●Echtzeit-Update zum gesamten Projekt: mit vollständige Kontrolle des experimentellen Fortschritts.
●Umfassende Standard -Bioinformatik:Das Paket umfasst 29 Analysen und mehr als 100 hochwertige Zahlen.
●Customisierte Datenanalyse und Visualisierung: verfügbar für verschiedene Forschungsanfragen.
●Optionale gemeinsame Analyse mit einer Einzelzell-mRNA-Sequenzierung
Spezifikationen
| Stichprobenanforderungen | Bibliothek | Sequenzierungsstrategie | Daten empfohlen | Qualitätskontrolle |
| OCT-eingebettete Kryo-Proben (Optimaler Durchmesser: ca. 6x6x6 mm³) 2 Blöcke pro Probe | 10x Visium -cDNA -Bibliothek | Illumina PE150 | 50k PE liest pro Spot (60 GB) | Rin> 7 |
Weitere Informationen zu den Anleitungen und dem Service -Workflow für Beispiele zur VorbereitungBmkgene Experte
Service Workflow
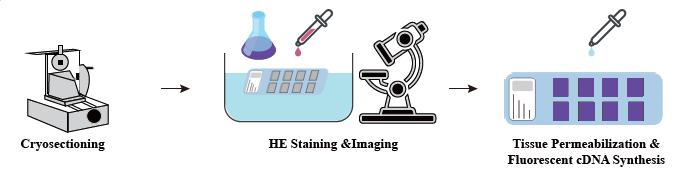
In der Probenvorbereitungsphase wird eine anfängliche Bulk-RNA-Extraktionsstudie durchgeführt, um sicherzustellen, dass eine hochwertige RNA erhalten werden kann. In der Stufe der Gewebeoptimierung werden die Abschnitte gefärbt und visualisiert und die Permeabilisierungsbedingungen für die mRNA -Freisetzung aus Gewebe optimiert. Das optimierte Protokoll wird dann während der Bibliothekskonstruktion angewendet, gefolgt von Sequenzierung und Datenanalyse.
Der vollständige Service-Workflow umfasst Echtzeit-Updates und Client-Bestätigungen, um eine reaktionsschnelle Rückkopplungsschleife aufrechtzuerhalten, um eine reibungslose Projektausführung zu gewährleisten.

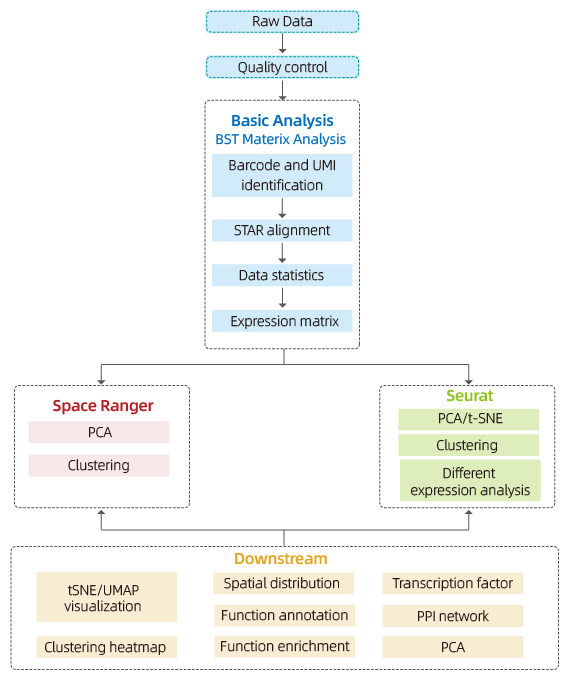
Enthält die folgende Analyse:
Datenqualitätskontrolle:
o Datenausgangs- und Qualitätsbewertungsverteilung
o Generkennung pro Punkt
o Gewebeabdeckung
Analyse der inneren Stichprobe:
o Genreichtum
o Spot -Clustering, einschließlich reduzierter Dimensionsanalyse
o Differentiale Expressionsanalyse zwischen Clustern: Identifizierung von Marker -Genen
o Funktionelle Annotation und Anreicherung von Markergenen
Analyse zwischen Gruppen
o Neuvernetzung von Flecken aus beiden Proben (z. B. krank und kontrolliert) und neu Cluster
o Identifizierung von Markergenen für jeden Cluster
o Funktionelle Annotation und Anreicherung von Markergenen
o Differenzielle Expression desselben Clusters zwischen den Gruppen
Analyse der inneren Stichprobe
Spot Clustering

Identifizierung von Markergenen und räumliche Verteilung


Analyse zwischen Gruppen
Datenkombination aus beiden Gruppen und Wiedererlebnis
Markergene neuer Cluster
Erforschen Sie die Fortschritte, die durch den räumlichen Transkriptomik -Service von BMKgene durch 10 -fach Visium in diesen vorgestellten Veröffentlichungen erleichtert wurden:
Chen, D. et al. (2023) 'Mthl1, ein potenzieller Drosophila -Homolog von GPCRs von Säugetieren, ist an Antitumorreaktionen auf injizierte onkogene Zellen in Fliegen beteiligt.Verfahren der Nationalen Akademie der Wissenschaften der Vereinigten Staaten von Amerika, 120 (30), p. E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'Stahl ermöglicht eine hochauflösende Abgrenzung von räumlich-zeitlichen transkriptomischen Daten',Briefings in Bioinformatik, 24 (2), S. 1–10. doi: 10.1093/bib/bbad068.
Liu, C. et al. (2022) 'ein räumlich -zeitlicher Atlas der Organogenese bei der Entwicklung von Orchideenblüten',Nukleinsäurenforschung, 50 (17), S. 9724–9737. doi: 10.1093/nar/gkac773.
Wang, J. et al. (2023) 'Integration der räumlichen Transkriptomik und RNA-Sequenzierung der Einzelnukleus zeigt die potenziellen therapeutischen Strategien für das Uterus-Leiomyom “,Internationales Journal of Biological Sciences, 19 (8), S. 2515–2530. doi: 10.7150/ijbs.83510.