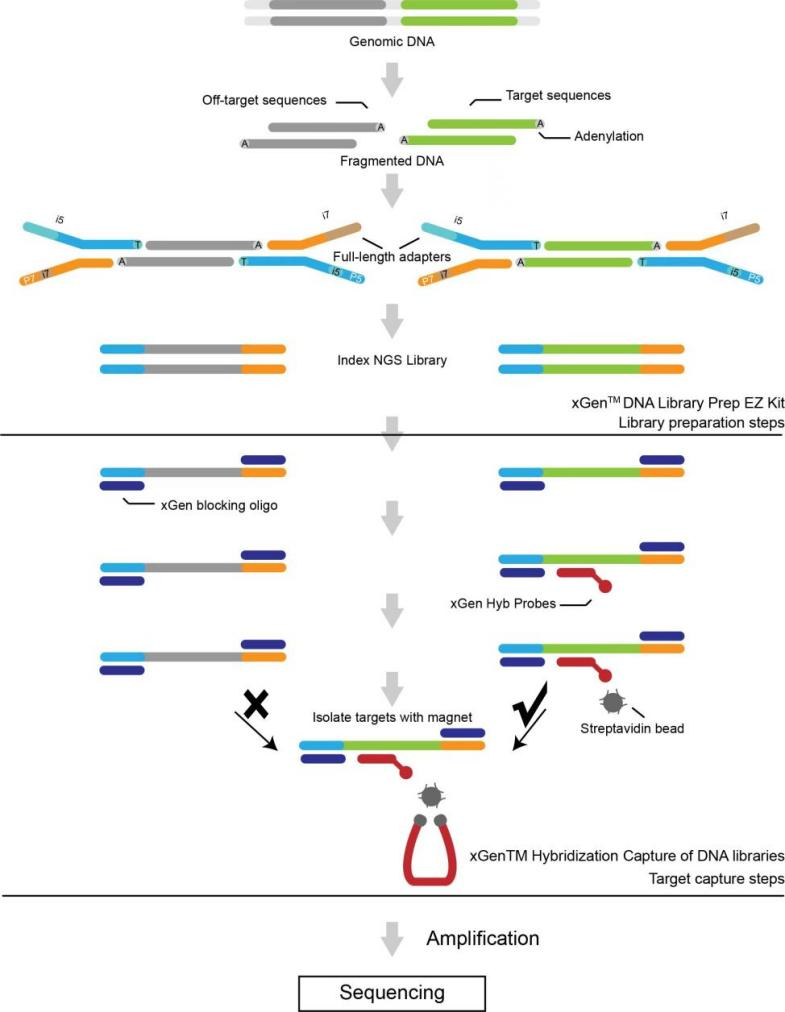
Human hel exome sekventering
Servicefunktioner
● To exome paneler tilgængelige baseret på målberigelse med sonder: Vælg Human All Exon V6 (Agilent) og XGen Exome Hybridization Panel V2 (IDT).
● Sekventering på Illumina Novaseq.
● Bioinformatisk rørledning rettet mod sygdomsanalyse eller tumoranalyse.
Servicefordele
●Mål Proteinkodningsregion: Ved at fange og sekventering af proteinkodningsregioner bruges HWE'er til at afsløre varianter relateret til proteinstruktur.
●Omkostningseffektiv:HWE'er giver ca. 85% af den humane sygdomsassocierede mutationer fra 1% af det humane genom.
●Høj nøjagtighed: Med høj sekventeringsdybde letter HWES påvisning af både almindelige varianter og sjældne varianter med frekvenser lavere end 1%.
●Streng kvalitetskontrol: Vi implementerer fem kernekontrolpunkter på tværs af alle faser, fra prøve- og biblioteksforberedelse til sekventering og bioinformatik. Denne omhyggelige overvågning sikrer levering af konsekvent resultater af høj kvalitet.
●Omfattende bioinformatikanalyse: Vores rørledning går ud over at identificere variationer i referencegenomet, da det inkorporerer avanceret analyse designet til specifikt at tackle forskningsspørgsmål relateret til genetiske aspekter af sygdomme eller tumoranalyse.
●Support efter salg:Vores forpligtelse strækker sig ud over projektets afslutning med en 3-måneders serviceperiode efter salg. I løbet af denne tid tilbyder vi projektopfølgning, fejlfinding af hjælp og Q & A-sessioner til at tackle eventuelle forespørgsler relateret til resultaterne.
Prøvespecifikationer
| Exon Capture Strategy | Sekventeringsstrategi | Anbefalet dataoutput |
| Sørg for at vælge Human All Exon V6 (Agilent) eller XGen Exome Hybridization Panel V2 (IDT)
| Illumina Novaseq PE150 | 5 -10 GB For Mendelian -lidelser/sjældne sygdomme:> 50x Til tumorprøver: ≥ 100x |
Prøvebehov
| Prøvetype
| Beløb(Qubit®)
| Koncentration | Bind
| Purity (Nanodrop ™) |
|
Genomisk DNA
| ≥ 50 ng | ≥ 6 ng/μl | ≥ 15 μl | OD260/280 = 1,8-2,0 Ingen nedbrydning, ingen forurening
|
Bioinformatik

Bioinformatisk analyse af HWES-sygdomsprøver inkluderer:
● Sekventeringsdata QC
● Referencegenomjustering
● Identifikation af SNP'er og indeller
● Funktionel annotation af SNP'er og indeller

Bioinformatisk analyse af tumorprøver inkluderer:
● Sekventeringsdata QC
● Referencegenomjustering
● Identifikation af SNP'er, indeller og somatiske variationer
● Identifikation af kimlinievarianter
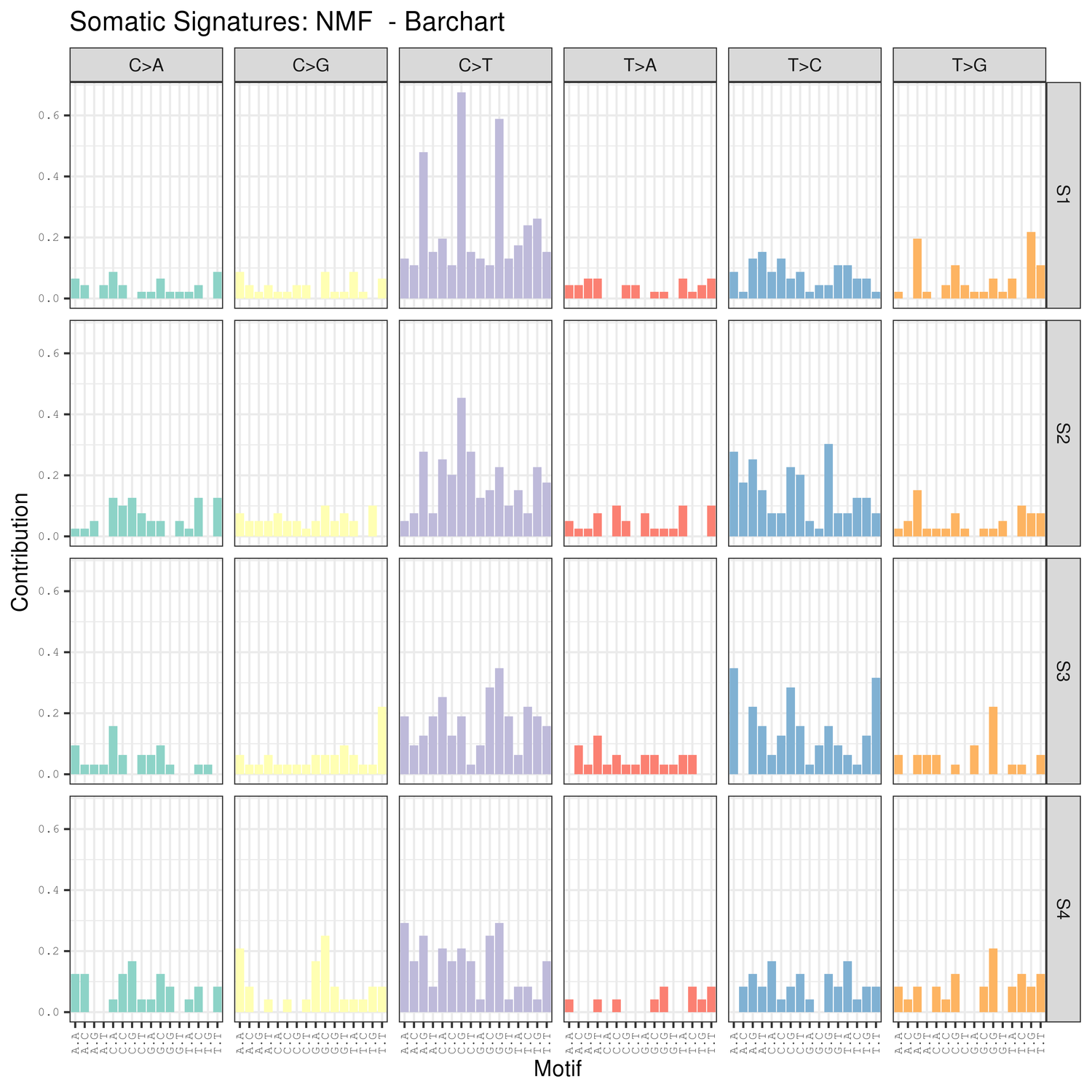
● Analyse af mutationssignaturer
● Identifikation af drevgener baseret på gain-of-funktion-mutationer
● Mutation Anotation på niveauet for lægemiddelfølsomhed
● Heterogenitetsanalyse - Beregning af renhed og ploidy
Service Work Flow

Eksempel på levering

DNA -ekstraktion

Bibliotekskonstruktion

Sekventering

Dataanalyse

Data levering

Eftersalg tjenester
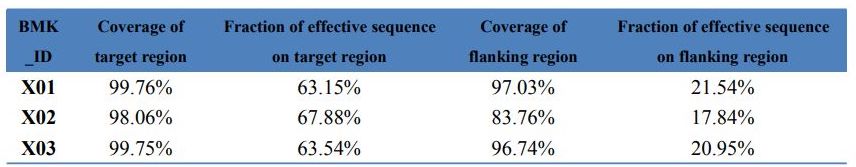
Data QC - Statistik over Exome Capture
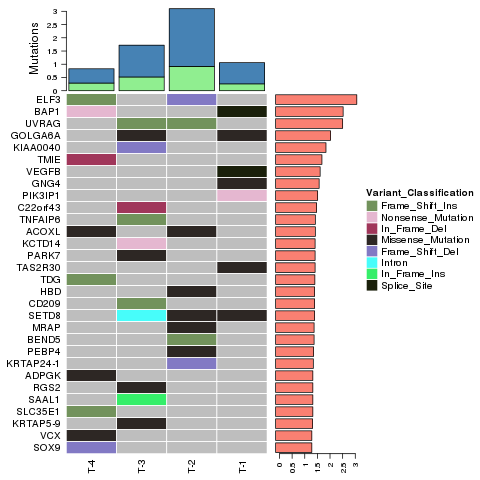
Variantidentifikation - Indeller
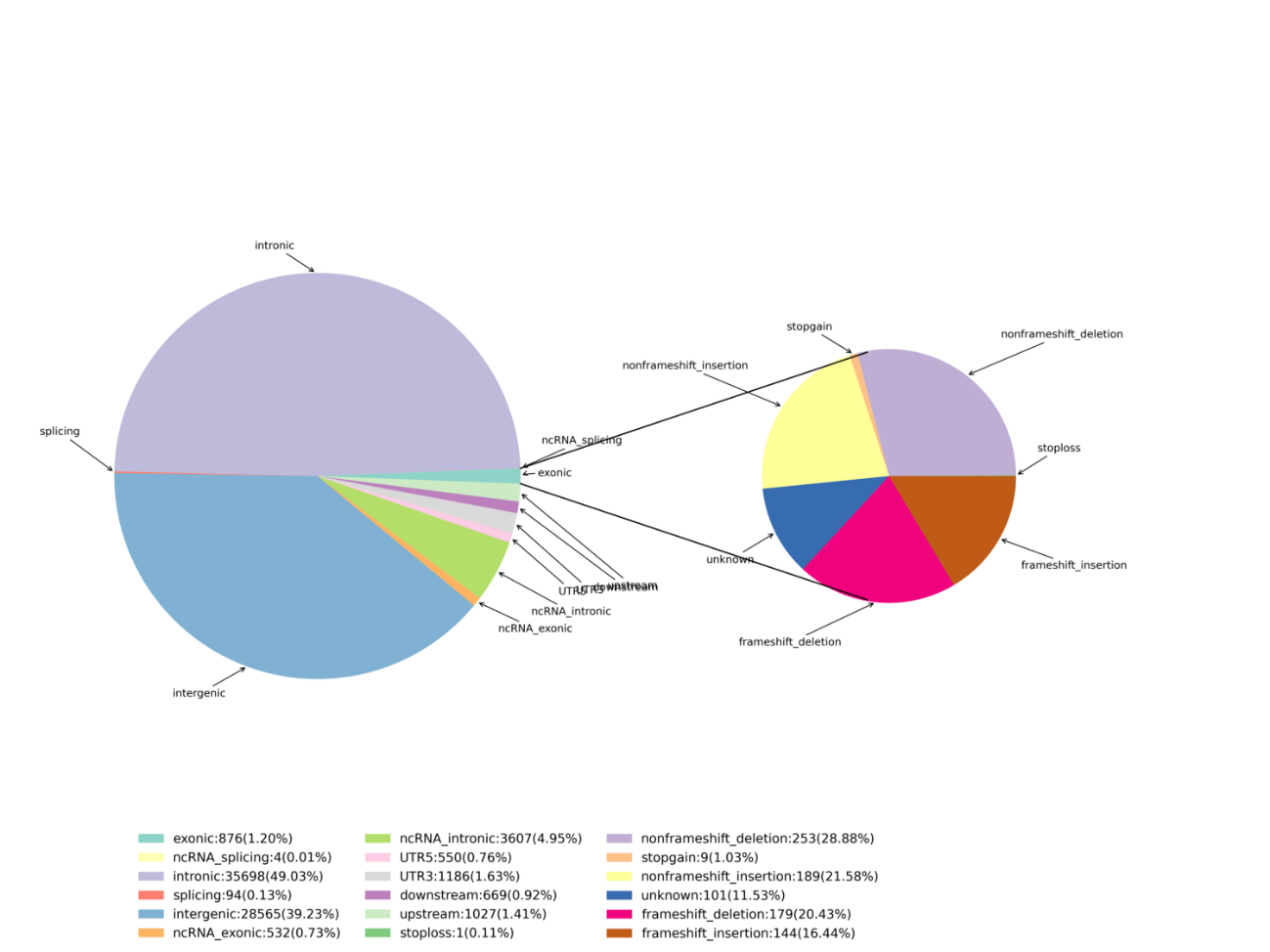
Avanceret analyse: Identifikation og distribution af skadelige SNP'er/Indeller - Circos Plot
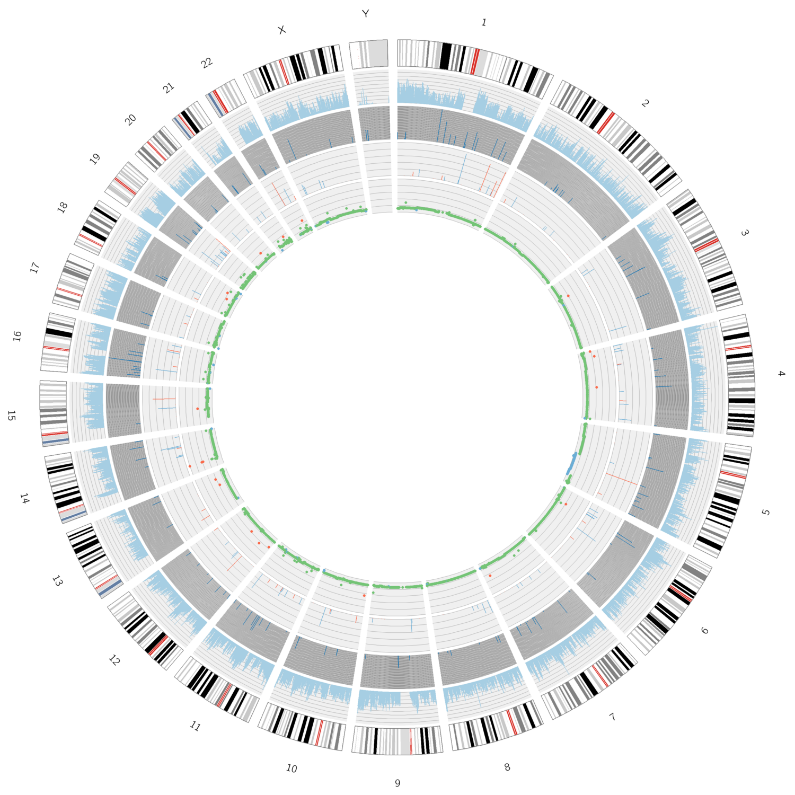
Tumoranalyse: Identifikation og distribution af somatiske mutationer - Circos Plot
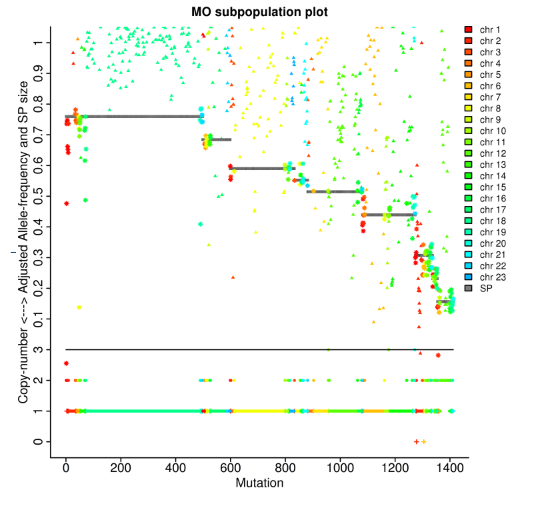
Tumoranalyse: Klonale linjer