Hi-C-baseret genomforsamling
Servicefunktioner
● Sekventering på Illumina Novaseq med PE150.
● Service kræver vævsprøver, i stedet for ekstraherede nukleinsyrer, til tværbinding med formaldehyd og bevarer DNA-proteininteraktionerne.
● Hi-C-eksperimentet involverer begrænsning og slutreparation af de klæbrige ender med biotin, efterfulgt af cirkularisering af de resulterende stumpe ender, mens interaktioner bevares. DNA'et trækkes derefter ned med streptavidinperler og oprenses til efterfølgende biblioteksforberedelse.
Servicefordele

Oversigt over Hi-C
(Lieberman-aiden E et al.,Videnskab, 2009)
●Fjernelse af behovet for genetiske populationsdata:HI-C erstatter de væsentlige oplysninger, der kræves til overordnet forankring.
●Høj markørtæthed:Fører til et højt forankringsforhold over 90%.
●Omfattende ekspertise og publikationsregistre:BMKGENE har enorm erfaring med mere end 2000 tilfælde af HI-C-genomsamling fra 1000 forskellige arter og forskellige patenter. Over 200 offentliggjorte sager har en akkumulerende påvirkningsfaktor på over 2000.
●Højt kvalificeret bioinformatik team:Med interne patenter og softwareophavsret til HI-C-eksperimenter og dataanalyse muliggør den selvudviklede visualiseringsdata-software manuel blokering, reversering, tilbagekaldelse og gentagelse.
●Support efter salg:Vores forpligtelse strækker sig ud over projektets afslutning med en 3-måneders serviceperiode efter salg. I løbet af denne tid tilbyder vi projektopfølgning, fejlfinding af hjælp og Q & A-sessioner til at tackle eventuelle forespørgsler relateret til resultaterne.
●Omfattende annotation: Vi bruger flere databaser til funktionelt at kommentere generne med identificerede variationer og udføre den tilsvarende berigelsesanalyse, hvilket giver indsigt i flere forskningsprojekter.
Servicespecifikationer
| Biblioteksforberedelse | Sekventeringsstrategi | Anbefalet dataoutput | Kvalitetskontrol |
| Hi-C-bibliotek | Illumina Novaseq PE150 | 100x | Q30 ≥ 85% |
Prøvebehov
| Væv | Krævet beløb |
| Dyresiskancera | ≥ 2 g |
| Dyremuskel | |
| Pattedyrblod | ≥ 2 ml |
| Fjerkræ/fiskeblod | |
| Plante- frisk blad | ≥ 3 g |
| Dyrkede celler | ≥ 1x107 |
| Insekt | ≥ 2 g |
Service Work Flow

Eksperimentdesign

Eksempel på levering

Bibliotekskonstruktion

Sekventering

Dataanalyse

Eftersalg tjenester
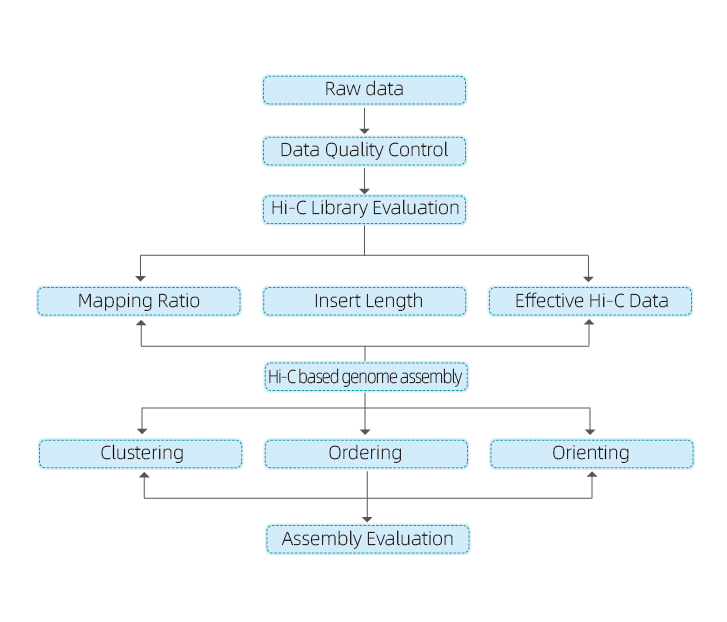
1) Rå data QC
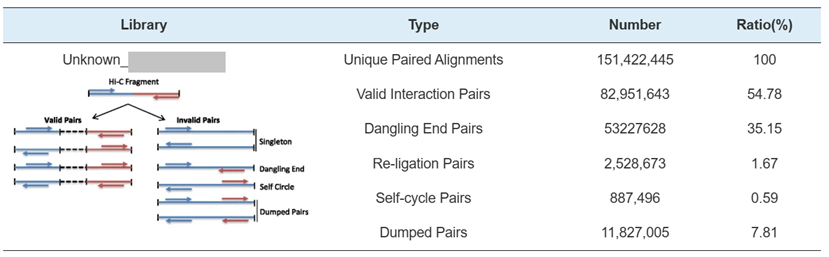
2) Hi-C-bibliotek QC: Estimering af gyldige HI-C-interaktioner
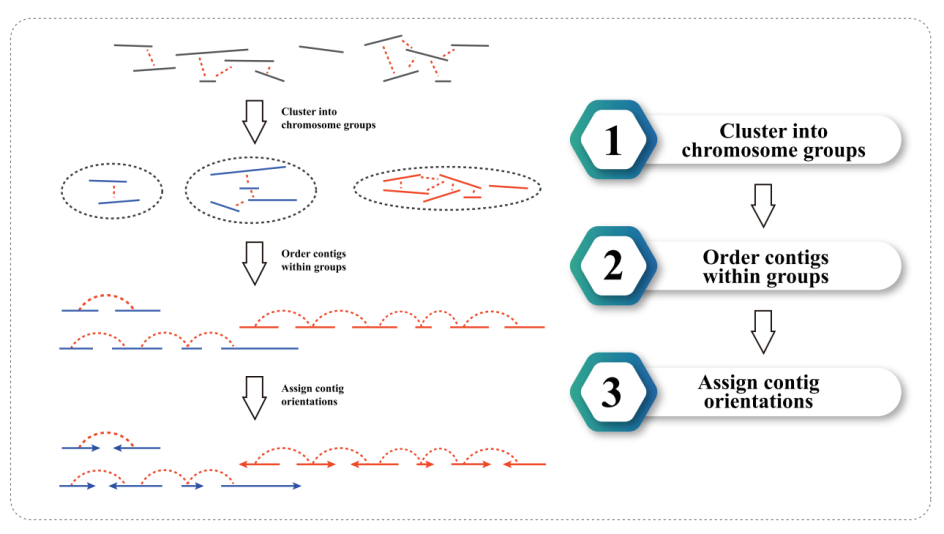
3) Hi-C-samling: Clustering of Contigs i grupper, efterfulgt af Contig-bestilling inden for hver gruppe og tildeling af Contig Orientation
4) Hi-C-evaluering
Hi-C Bibliotek QC-Estimering af Hi-C Gyldige interaktionspar
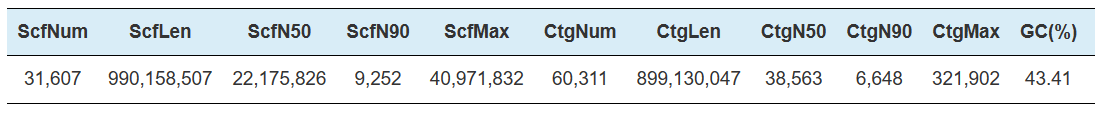
Hi-C-samling-Statistik
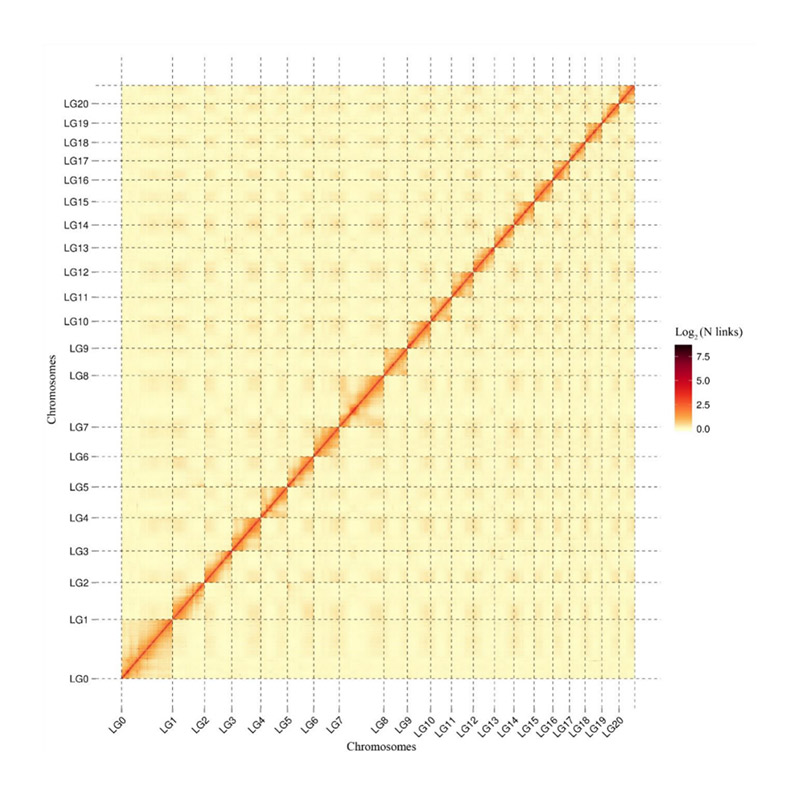
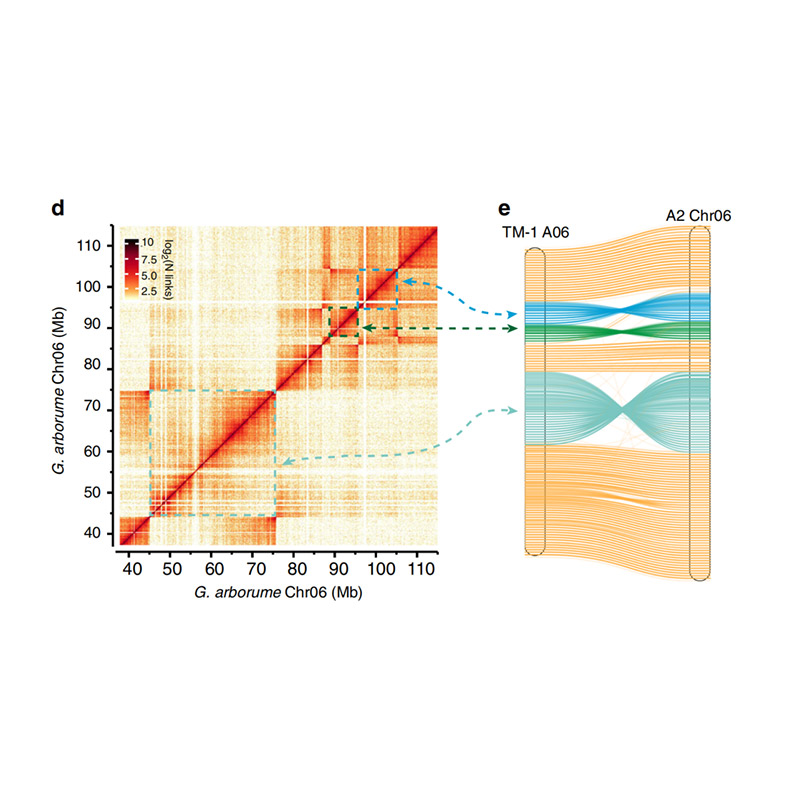
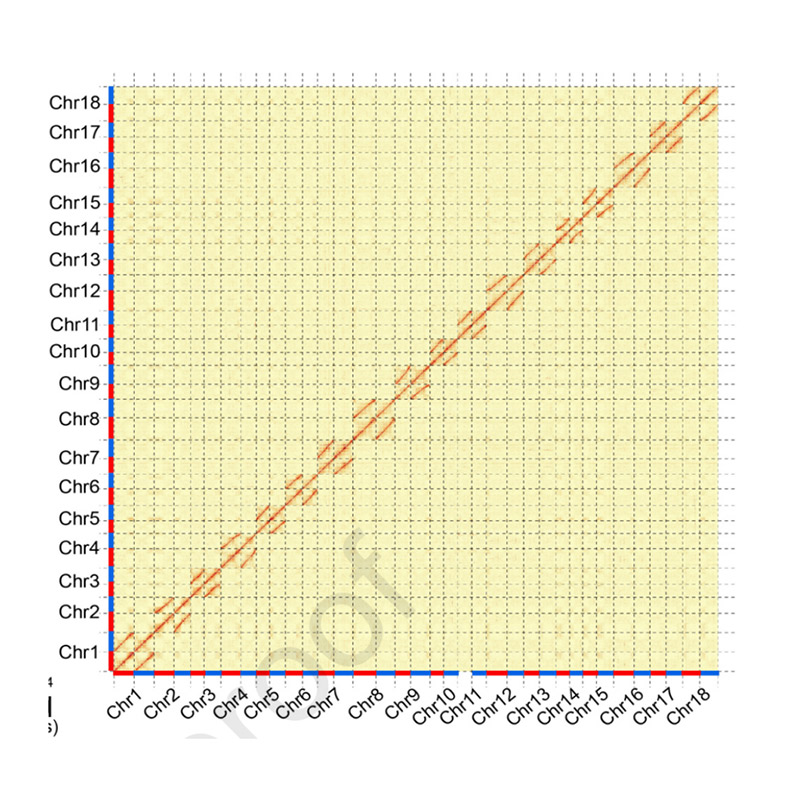
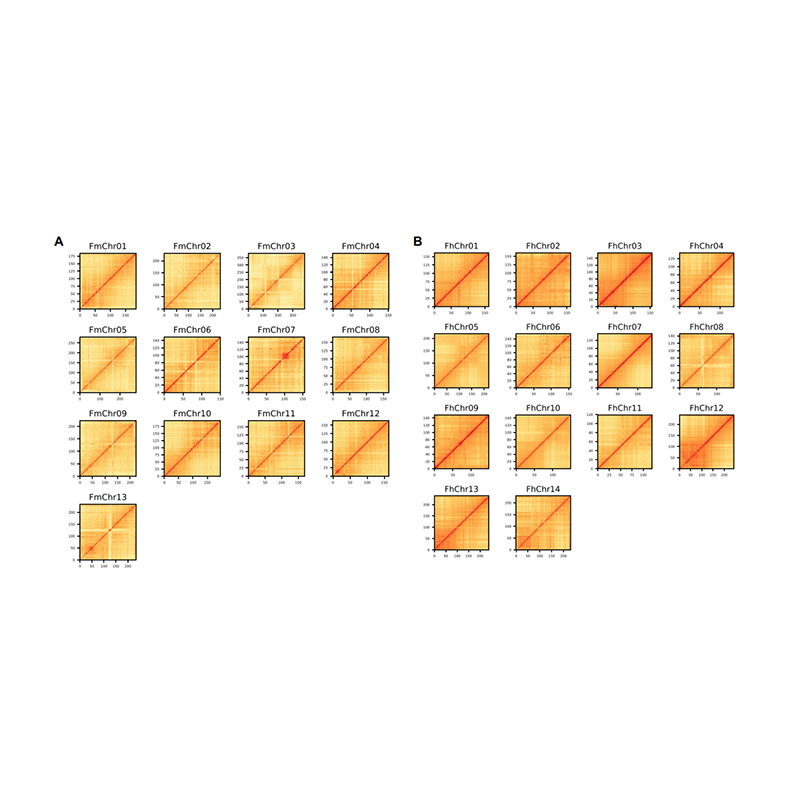
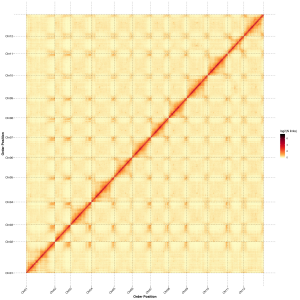
Evaluering efter forsamling-varmekort af signalintensitet mellem skraldespande
Udforsk de fremskridt, der er lettet af BMKGENEs HI-C Assembly Services gennem en kurateret samling af publikationer.
Tian, T. et al. (2023) 'Genomforsamling og genetisk dissektion af en fremtrædende tørkebestandig majs-kimplasme', Nature Genetics 2023 55: 3, 55 (3), s. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'En kromosomskala-samling af den asiatiske honningbi-apis cerana genom', Frontiers in Genetics, 11, p. 524140. Doi: 10.3389/fgene.2020.00279/Bibtex.
Zhang, F. et al. (2023) 'afslørende udvikling af tropanalkaloidbiosyntesen ved at analysere to genomer i Solanaceae -familien', Nature Communications 2023 14: 1, 14 (1), s. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) 'Genomer fra Banyan Tree and Pollinator Wasp giver indsigt i fig-wasp coevolution', Cell, 183 (4), s. 875-889.E17. doi: 10.1016/j.cell.2020.09.043