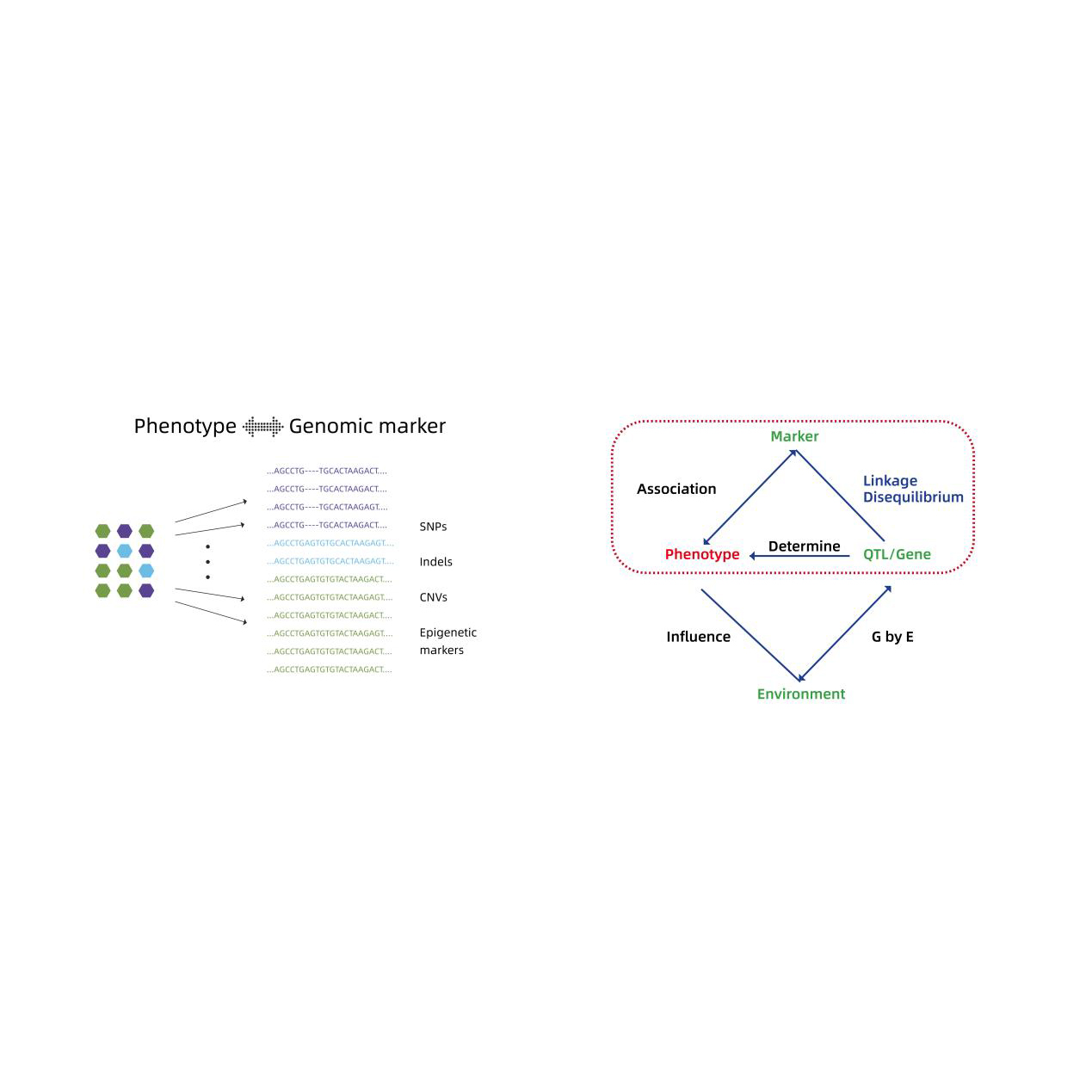
Genom-dækkende associationsanalyse
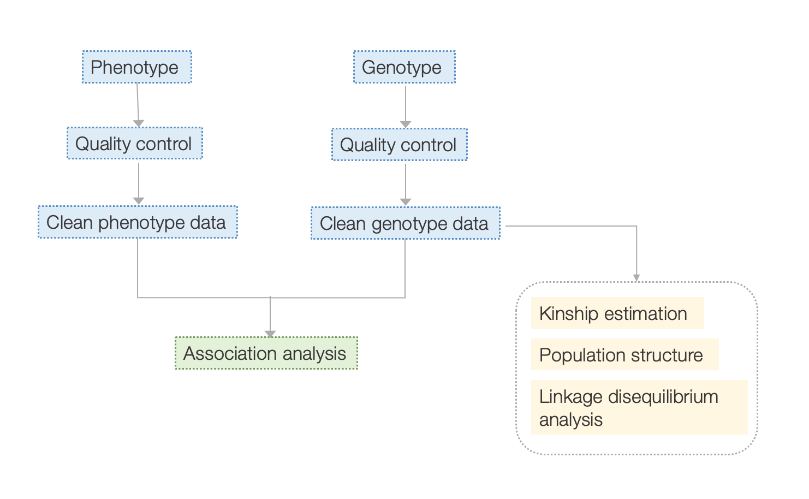
Arbejdsgang

Service fordele
●Omfattende ekspertise og publikationsoptegnelser: med akkumuleret erfaring i GWAS har BMKGene gennemført hundredvis af artsprojekter i populations-GWAS-forskning, hjulpet forskere med at publicere mere end 100 artikler, og den kumulative effektfaktor nåede 500.
● Omfattende bioinformatikanalyse: Workflow inkluderer SNP-trækassocieringsanalyse, der leverer et sæt kandidatgener og deres tilsvarende funktionelle annotering.
●Højt kvalificeret bioinformatikteam og kort analysecyklus: Med stor erfaring i avanceret genomisk analyse leverer BMKGenes team omfattende analyser med en hurtig ekspeditionstid.
●Support efter salg:Vores engagement strækker sig ud over projektafslutning med en 3-måneders eftersalgsserviceperiode. I løbet af denne tid tilbyder vi projektopfølgning, fejlfindingshjælp og Q&A-sessioner for at løse eventuelle spørgsmål relateret til resultaterne.
Servicespecifikationer og krav
| Type af sekventering | Anbefalet befolkningsskala | Sekvenseringsstrategi | Nukleotidkrav |
| Helgenom-sekventering | 200 prøver | 10x | Koncentration: ≥ 1 ng/µL Samlet mængde ≥ 30ng Begrænset eller ingen nedbrydning eller forurening |
| Specific-Locus Amplified Fragment (SLAF) | Tag dybde: 10x Antal tags: < 400 Mb: WGS anbefales < 1 Gb: 100K tags 1 Gb > 2Gb: 300K tags Max 500.000 tags | Koncentration ≥ 5 ng/µL Samlet mængde ≥ 80 ng Nanodrop OD260/280=1,6-2,5 Agarosegel: ingen eller begrænset nedbrydning eller forurening
|
Materialevalg



Forskellige sorter, underarter, landracer/genbanker/blandede familier/vilde ressourcer
Forskellige sorter, underarter, landracer
Halvsøskendefamilie/helsøskendefamilie/vilde ressourcer
Service Work Flow

Eksperimentdesign

Prøve levering

RNA-ekstraktion

Biblioteksbyggeri

Sekvensering

Dataanalyse

Eftersalgsservice
Indeholder følgende analyse:
- Genom-dækkende associationsanalyse: LM, LMM, EMMAX, FASTLMM model
- Funktionel annotering af kandidatgener
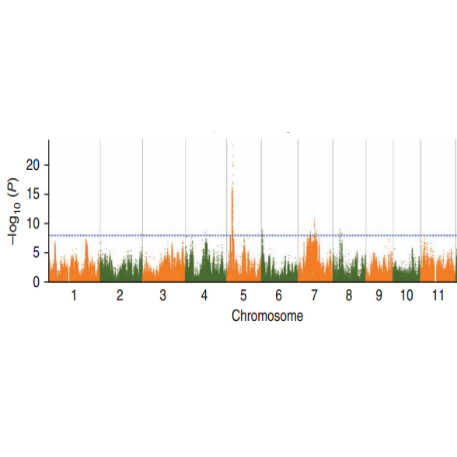
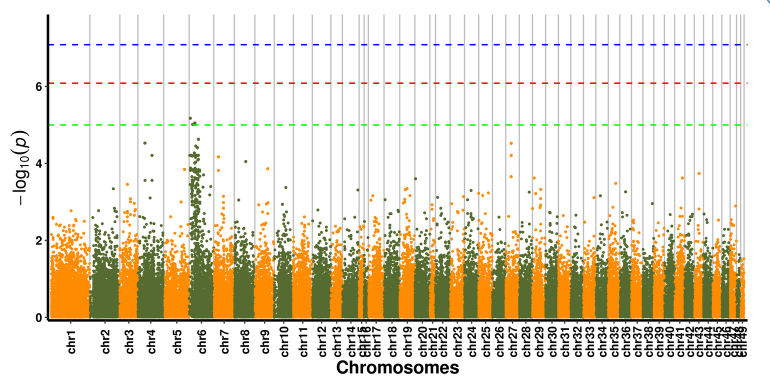
SNP-trækassociationsanalyse – Manhattan plot
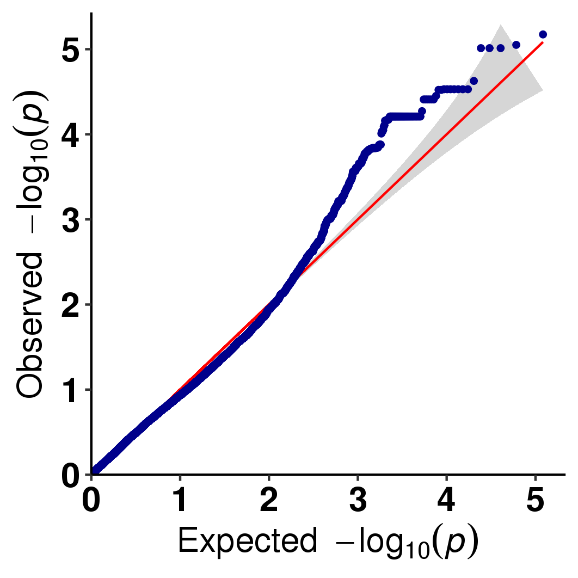
SNP-træk associationsanalyse – QQ plot
Udforsk de fremskridt, der er lettet af BMKGene's de GWAS-tjenester gennem en kurateret samling af publikationer:
Lv, L. et al. (2023) 'Indsigt i det genetiske grundlag for ammoniaktolerance i barbermusling Sinonovacula constricta ved en genomomfattende associationsundersøgelse',Akvakultur569, s. 739351. doi: 10.1016/J.AQUACULTURE.2023.739351.
Li, X. et al. (2022) "Multi-omics analyser af 398 rævehale hirse accessioner afslører genomiske regioner forbundet med domesticering, metabolittræk og antiinflammatoriske effekter",Molekylær plante, 15(8), s. 1367-1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) 'Genomomfattende foreningskortlægning af hulless knapt fænotyper i tørkemiljø',Grænser i plantevidenskab, 13, s. 924892. doi: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) 'GmST1, som koder for en sulfotransferase, giver resistens over for sojabønnemosaikvirusstammer G2 og G3',Plant, Cell & Miljø, 44(8), s. 2777-2792. doi: 10.1111/PCE.14066.