Fuld-længde mRNA-sekventering-Nanopore
Funktioner
● Indfangning af poly-A-mRNA efterfulgt af cDNA-syntese og biblioteksforberedelse
● Rækkefølge af transskriptionerne i fuld længde
● Bioinformatisk analyse baseret på tilpasning til et referencegenom
● Bioinformatisk analyse omfatter ikke kun ekspression på gen- og isoform-niveau, men også analyse af lncRNA, genfusioner, polyadenylering og genstruktur
Service fordele
●Kvantificering af udtryk på isoformniveau: muliggør detaljeret og nøjagtig ekspressionsanalyse, afsløring af ændringer, der kan maskeres, når man analyserer hele genekspressionen
●Reducerede datakrav:Sammenlignet med Next-Generation Sequencing (NGS), udviser Nanopore-sekventering lavere datakrav, hvilket giver mulighed for tilsvarende niveauer af genekspression kvantificeringsmætning med mindre data.
●Højere nøjagtighed af udtryk kvantificering: både på gen- og isoformniveau
●Identifikation af yderligere transkriptomisk information: alternativ polyadenylering, fusionsgener og lcnRNA og deres målgener
●Omfattende ekspertise: Vores team bringer et væld af erfaring til hvert projekt, efter at have gennemført over 850 Nanopore fuldlængde transkriptomprojekter og behandlet over 8.000 prøver.
●Support efter salg: Vores engagement strækker sig ud over projektafslutning med en 3-måneders eftersalgsserviceperiode. I løbet af denne tid tilbyder vi projektopfølgning, fejlfindingshjælp og Q&A-sessioner for at løse eventuelle spørgsmål relateret til resultaterne.
Prøvekrav og levering
| Bibliotek | Sekvenseringsstrategi | Data anbefales | Kvalitetskontrol |
| Poly A beriget | Illumina PE150 | 6/12 Gb | Gennemsnitlig kvalitetsscore: Q10 |
Prøvekrav:
Nukleotider:
| Konc.(ng/μl) | Mængde (μg) | Renhed | Integritet |
| ≥ 100 | ≥ 1,0 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Begrænset eller ingen protein- eller DNA-kontamination vist på gel. | For planter: RIN≥7,0; For dyr: RIN≥7,5; 5,0≥28S/18S≥1,0; begrænset eller ingen basislinjestigning |
● Planter:
Rod, stilk eller kronblad: 450 mg
Blad eller frø: 300 mg
Frugt: 1,2 g
● Dyr:
Hjerte eller tarm: 300 mg
Indvolde eller hjerne: 240 mg
Muskel: 450 mg
Knogler, hår eller hud: 1g
● Leddyr:
Insekter: 6g
Krebsdyr: 300 mg
● Fuldblod: 1 rør
● Celler: 106 celler
Anbefalet prøvelevering
Beholder: 2 ml centrifugerør (stanniol anbefales ikke)
Prøvemærkning: Gruppe+replikat f.eks. A1, A2, A3; B1, B2, B3.
Forsendelse:
1. Tøris: Prøver skal pakkes i poser og begraves i tøris.
2. RNA-stabile rør: RNA-prøver kan tørres i RNA-stabiliseringsrør (f.eks. RNAstable®) og sendes i stuetemperatur.
Service Work Flow
Nukleotider:

Prøve levering

Biblioteksbyggeri

Sekvensering

Dataanalyse

Eftersalgsservice
Service Work Flow
Væv:

Eksperimentdesign

Prøve levering

RNA-ekstraktion

Biblioteksbyggeri

Sekvensering

Dataanalyse

Eftersalgsservice
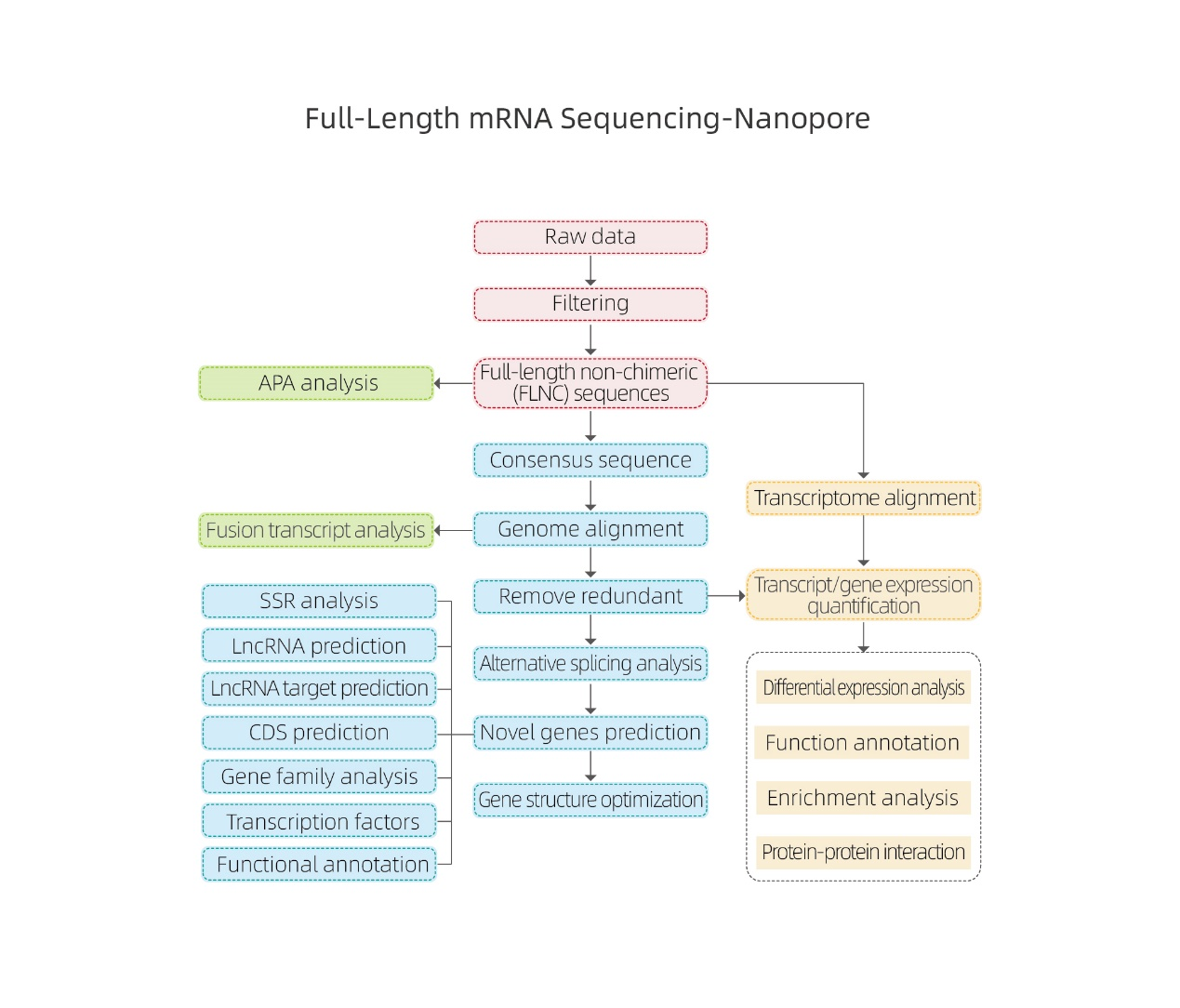
● Rådatabehandling
● Udskriftsidentifikation
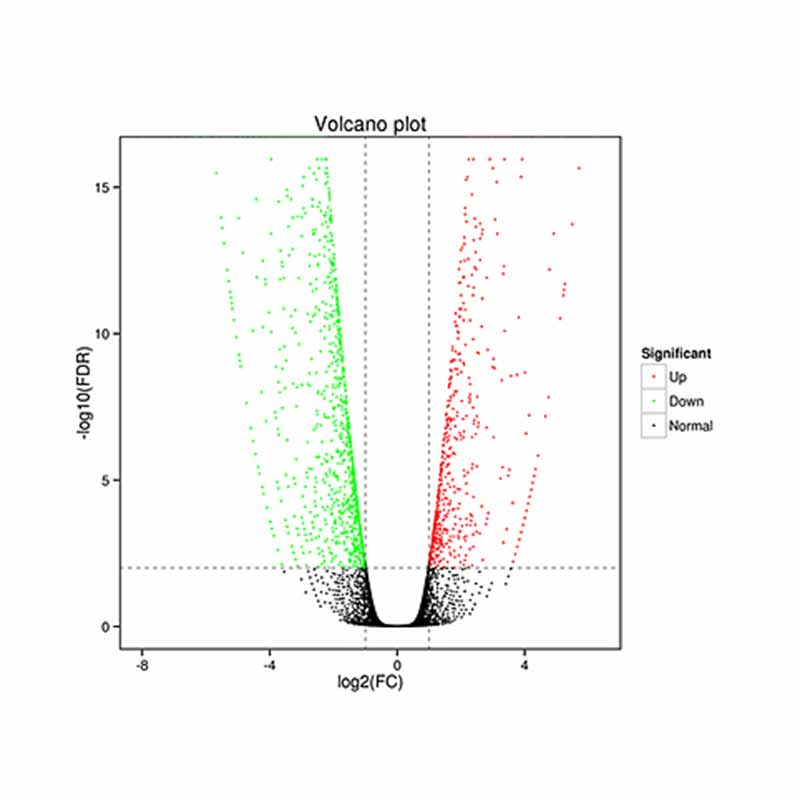
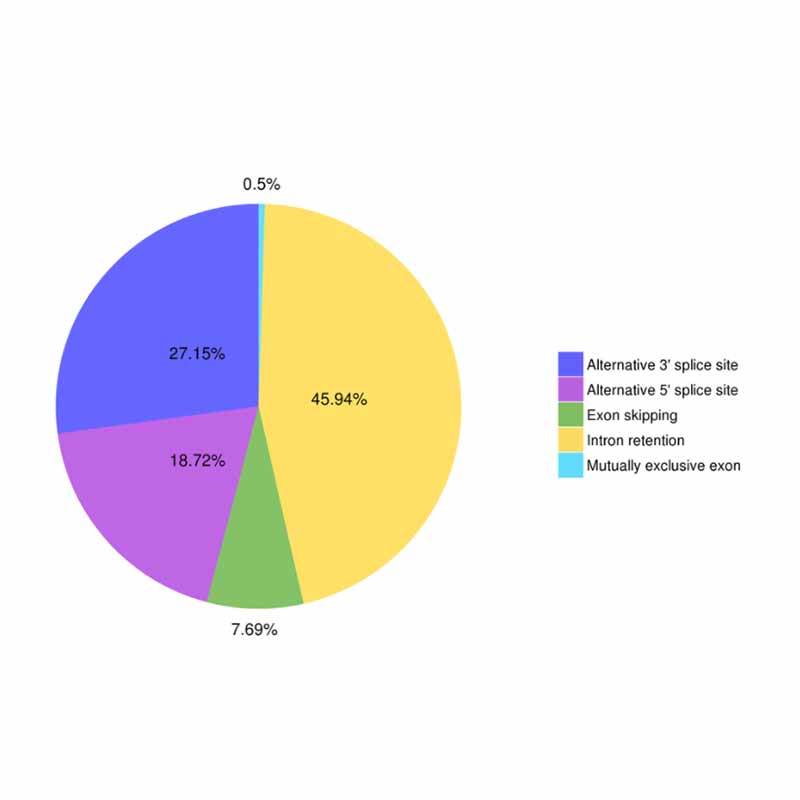
● Alternativ splejsning
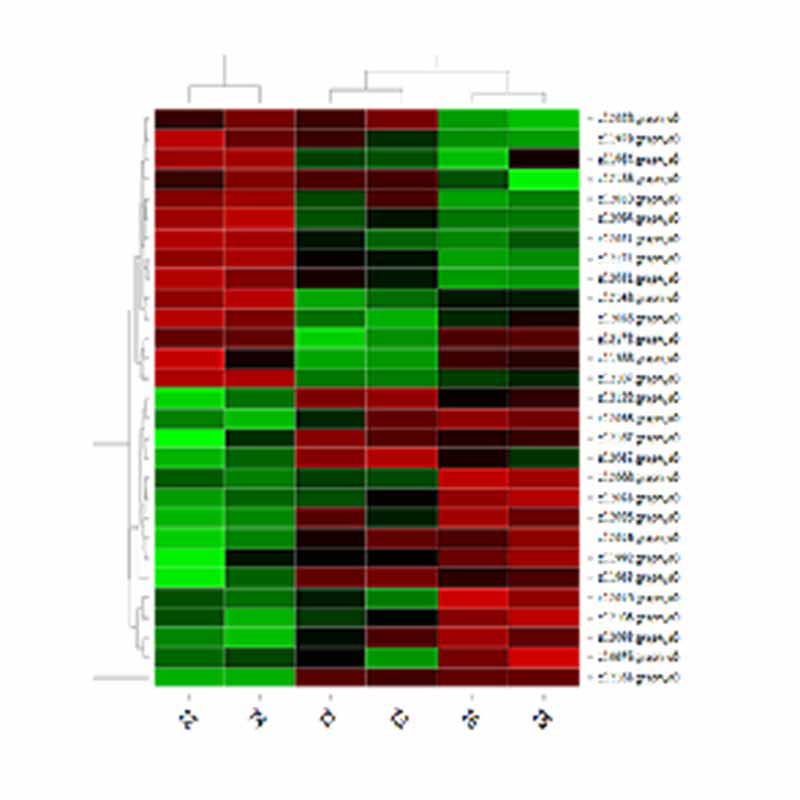
● Ekspressionskvantificering i genniveau og isoformniveau
● Differentiel udtryksanalyse
● Funktionsannotering og berigelse (DEG'er og DET'er)
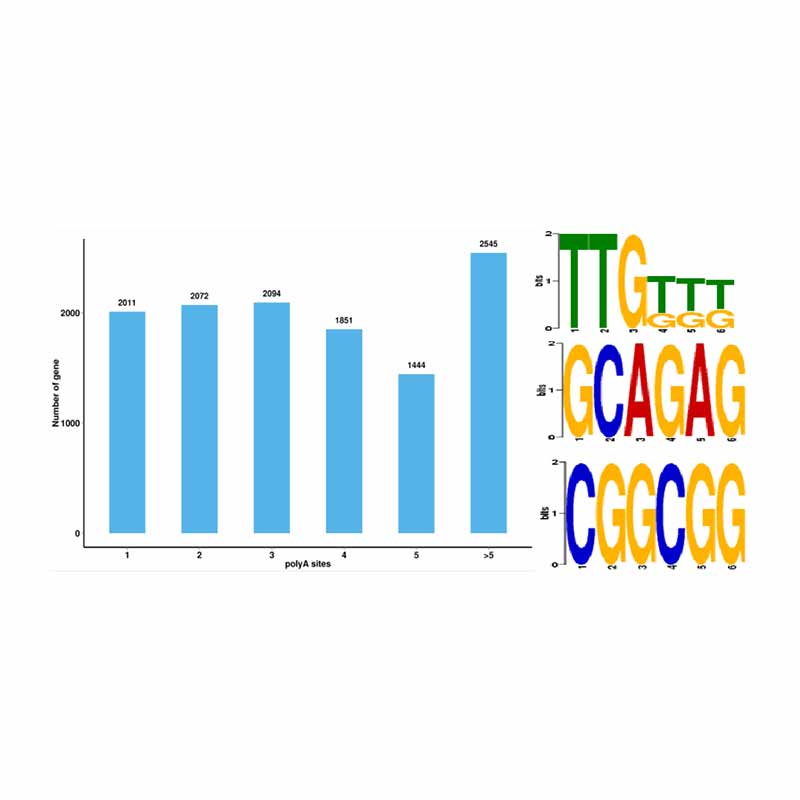
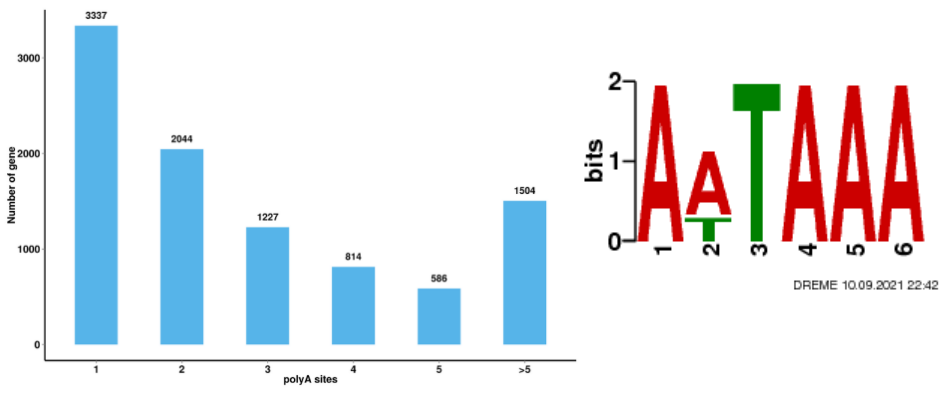
Alternativ splejsningsanalyse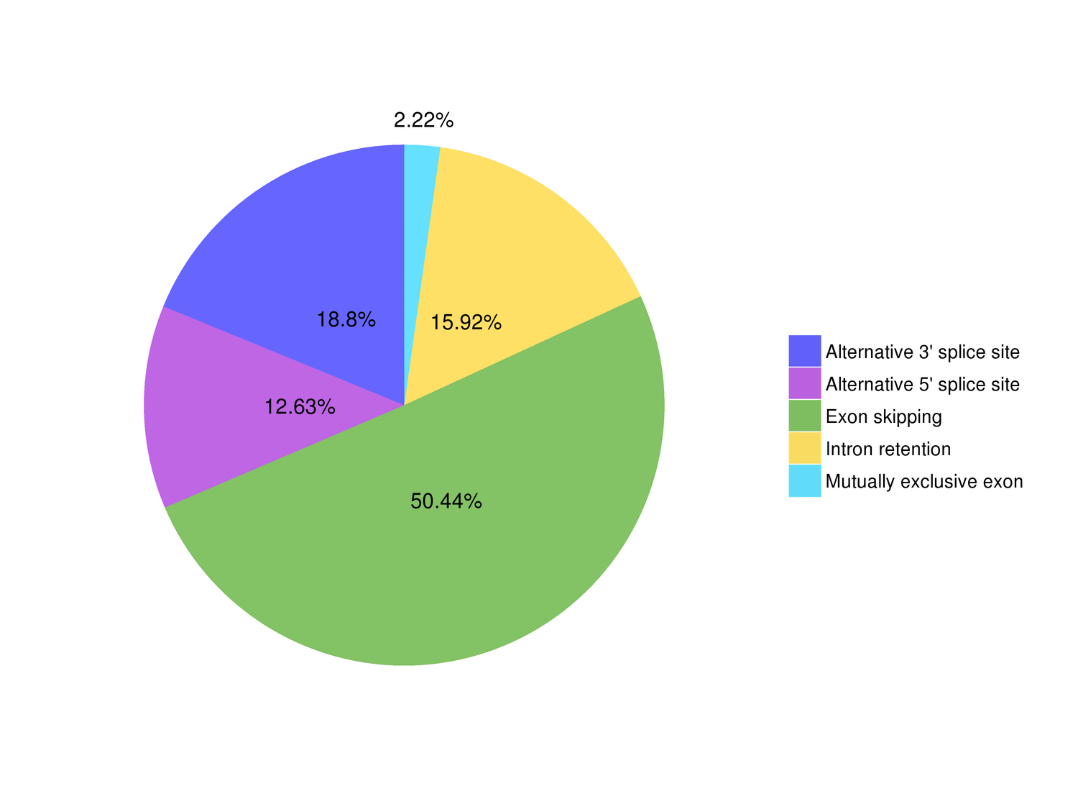 Alternativ polyadenyleringsanalyse (APA)
Alternativ polyadenyleringsanalyse (APA)
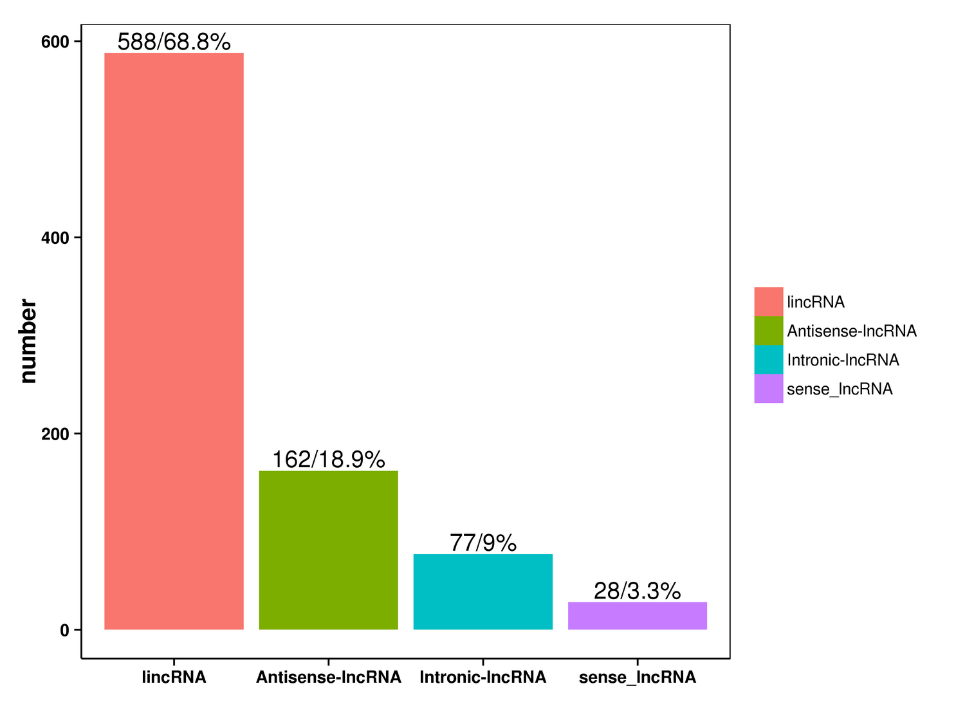
lncRNA forudsigelse
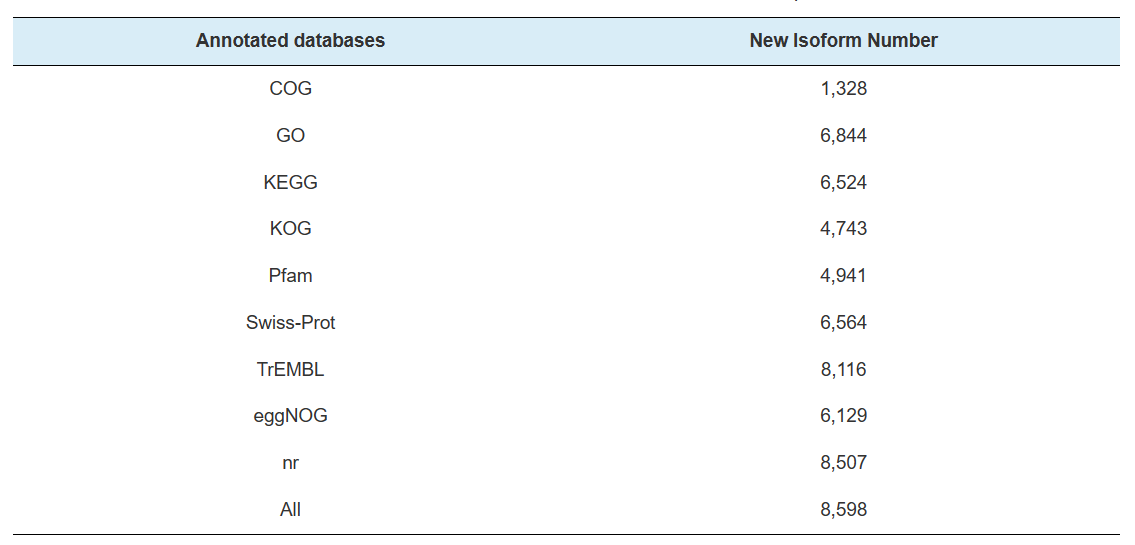
Annotering af nye gener
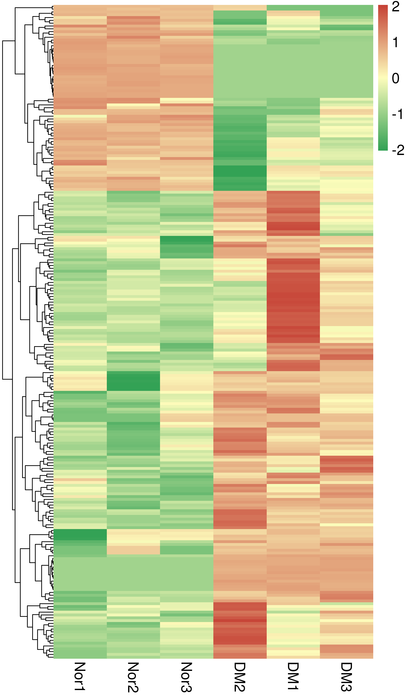
Klynger af DET'er
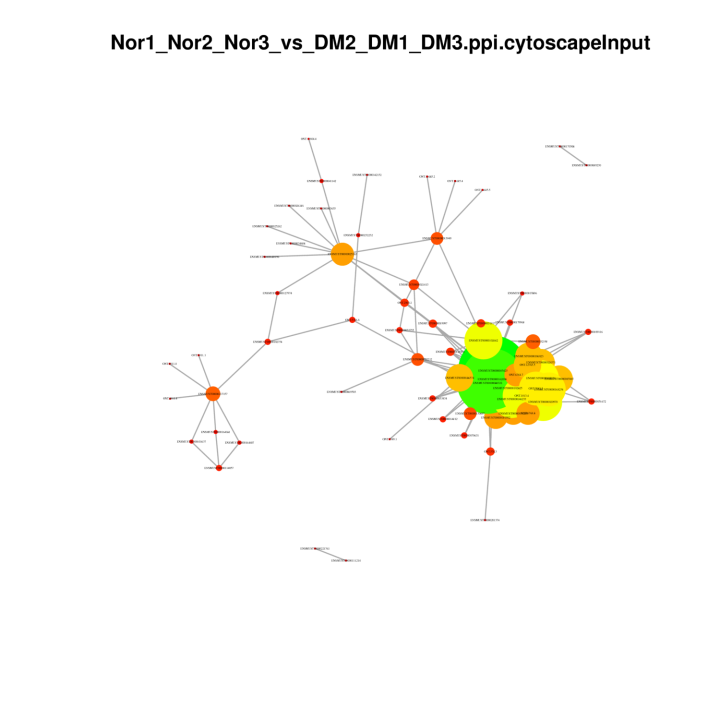
Protein-protein-netværk i DEG'er
Udforsk de fremskridt, der er lettet af BMKGene's Nanopore fuldlængde mRNA-sekventeringstjenester gennem en kurateret samling af publikationer.
Gong, B. et al. (2023) 'Epigenetisk og transkriptionel aktivering af den sekretoriske kinase FAM20C som et onkogen i gliom', Journal of Genetics and Genomics, 50(6), s. 422-433. doi: 10.1016/J.JGG.2023.01.008.
Han, Z. et al. (2023) 'Fuldlængde transkriptomsekventering af lymfocytter reagerer på IFN-γ afslører en Th1-skæv immunreaktion i skrubber (Paralichthys olivaceus)', Fish & Shellfish Immunology, 134, s. 108636. doi: 10.1016/J.FSI.2023.108636.
Ma, Y. et al. (2023) 'Komparativ analyse af PacBio og ONT RNA-sekventeringsmetoder til Nemopilema Nomurai giftidentifikation', Genomics, 115(6), s. 110709. doi: 10.1016/J.YGENO.2023.110709.
Yu, D. et al. (2023) 'Nano-seq-analyse afslører forskellige funktionelle tendenser mellem exosomer og mikrovesikler afledt af hUMSC', Stem Cell Research and Therapy, 14(1), s. 1-13. doi: 10.1186/S13287-023-03491-5/TABELLER/6.