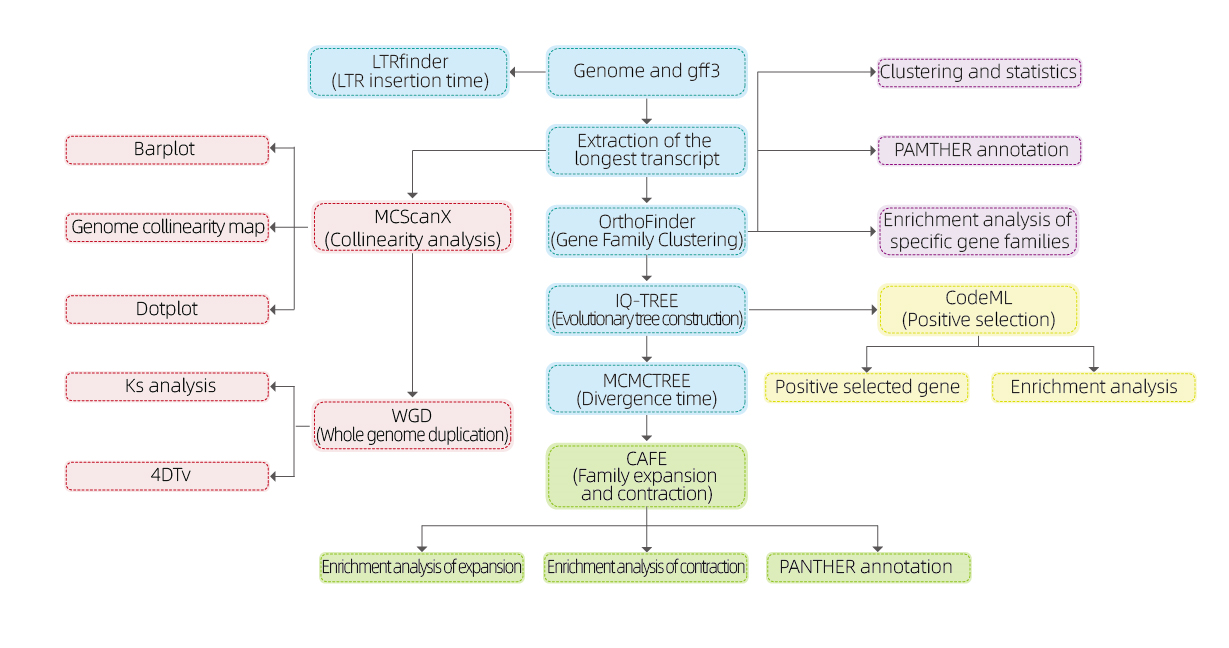
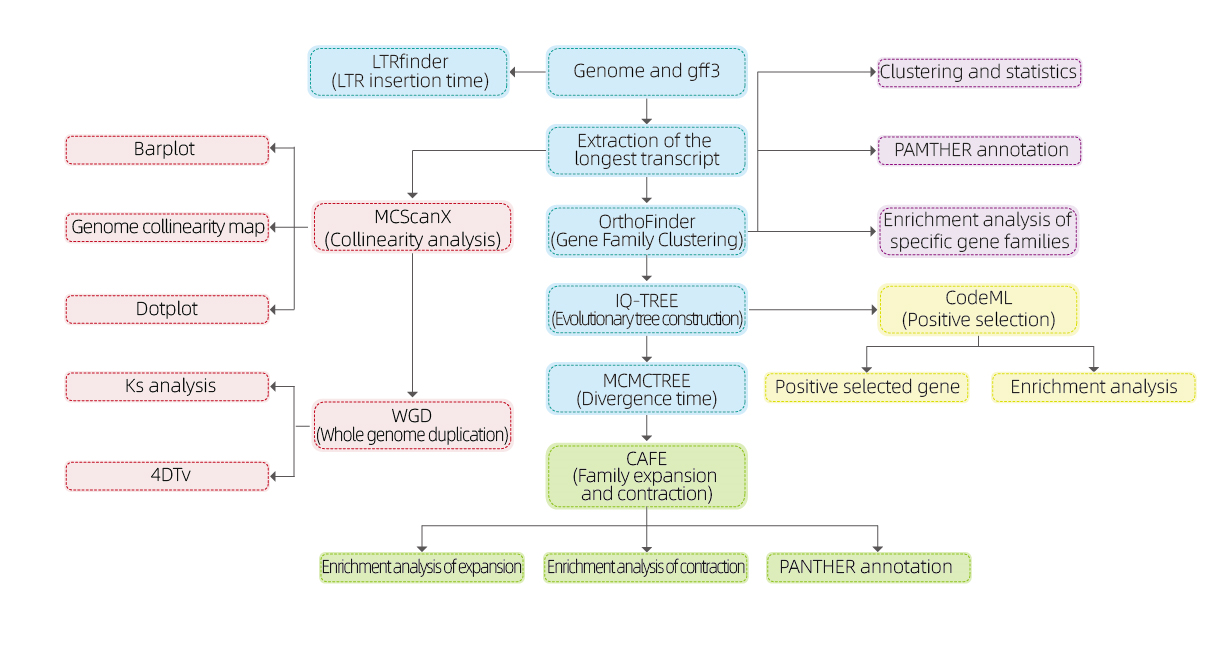
Sammenlignende genomik
Servicefordele
●Omfattende ekspertise og publikationsregistre: Med akkumuleret har BMKGENE afsluttet over 90 komparative genomikprojekter, med en kumulativ påvirkningsfaktor nået 900.
●Omfattende bioinformatikanalyse: Analyserpakken indeholder de otte mest krævede analyser, der giver godt designet klar til offentliggørelse og muliggør en let fortolkning af resultaterne
●Højt kvalificeret bioinformatik team og kort analysecyklus: Med stor erfaring med sammenlignende genomikanalyse opfylder Bmkgene's team forskellige personaliserede analysekrav på en kort omdrejningstid
●Support efter salg:Vores forpligtelse strækker sig ud over projektets afslutning med en 3-måneders serviceperiode efter salg. I løbet af denne tid tilbyder vi projektopfølgning, fejlfinding af hjælp og Q & A-sessioner til at tackle eventuelle forespørgsler relateret til resultaterne.
Servicespecifikationer
| Estimeret omdrejningstid | Antal arter | Analyser |
| 30 arbejdsdage | 6 - 12 | Gene Family Clustering Genfamilieudvidelse og sammentrækning Fylogenetisk træ konstruktion Estimering af divergenstid (fossil kalibrering krævet) LTR -indsættelsestid (for planter) Hele genomduplikation (til planter) Selektivt pres Synteny -analyse |
Bioinformatikanalyser
● Gene -familie
● Phylogenetics
● Divergenstid
● Selektivt pres
● Synteny -analyse
Eksempelkrav og levering
Eksempelkrav:
Væv eller DNA til genomsekventering og samling
Til væv
| Arter | Væv | Kortlægge | Pacbio CCS |
| Dyr | Visceralt væv | 0,5 ~ 1 g | ≥ 3,5 g |
| Muskelvæv | |||
| ≥ 5,0 g | |||
| ≥ 5,0 ml | |||
| Pattedyrblod | |||
| ≥ 0,5 ml | |||
| Fjerkræ/fiskeblod | |||
| Plante | Frisk blad | 1 ~ 2 g | ≥ 5,0 g |
| Kronblad/stilk | 1 ~ 2 g | ≥ 10,0 g | |
| Rod/frø | 1 ~ 2 g | ≥ 20,0 g | |
| Celler | Dyrket celle | - | ≥ 1 x 108 |
Data
Genomsekvensfiler (.fasta) og annotationsfiler (.gff3) af tæt beslægtede arter
Service Work Flow

Eksperimentdesign

Eksempel på levering

Bibliotekskonstruktion

Sekventering

Dataanalyse

Eftersalg tjenester
*Demo -resultater, der er vist her, er alle fra genomer offentliggjort med Biomarker -teknologier
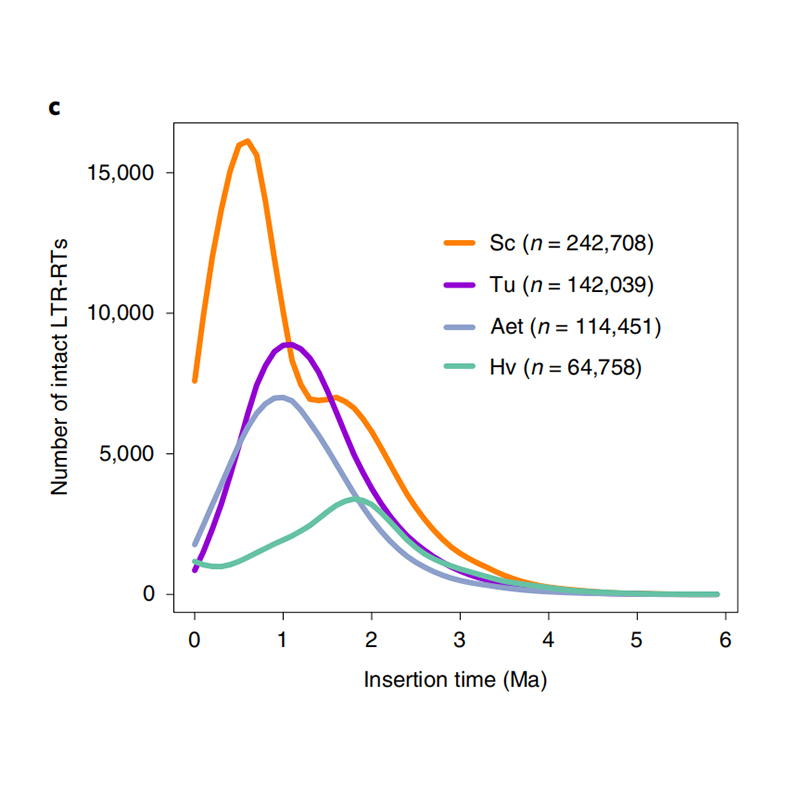
1.LTR INDSTILLING TID ESTIMATION: Figuren viste en unik bimodal fordeling i LTR-RTS-indsættelsestider i Weining Rye Genome sammenlignet med andre arter. Den seneste top optrådte for omkring 0,5 millioner år siden.
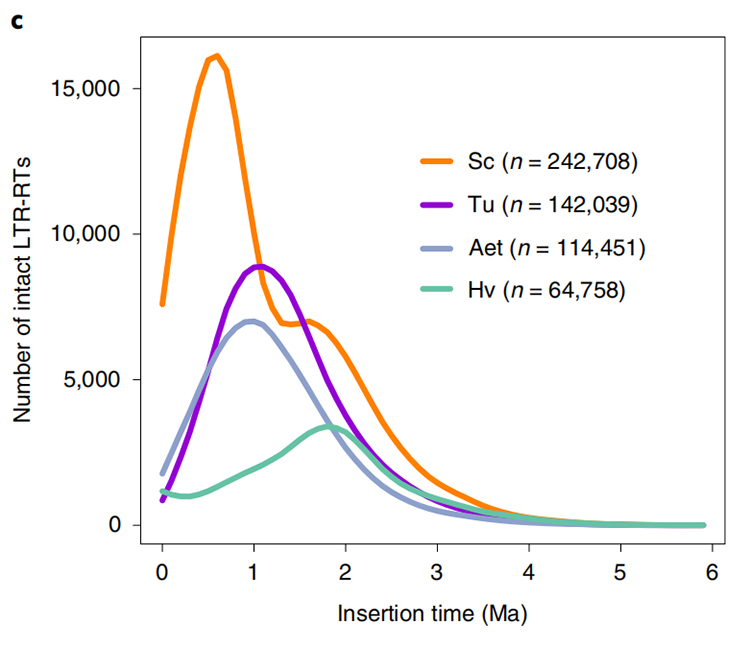
Li Guang et al.,Naturgenetik, 2021
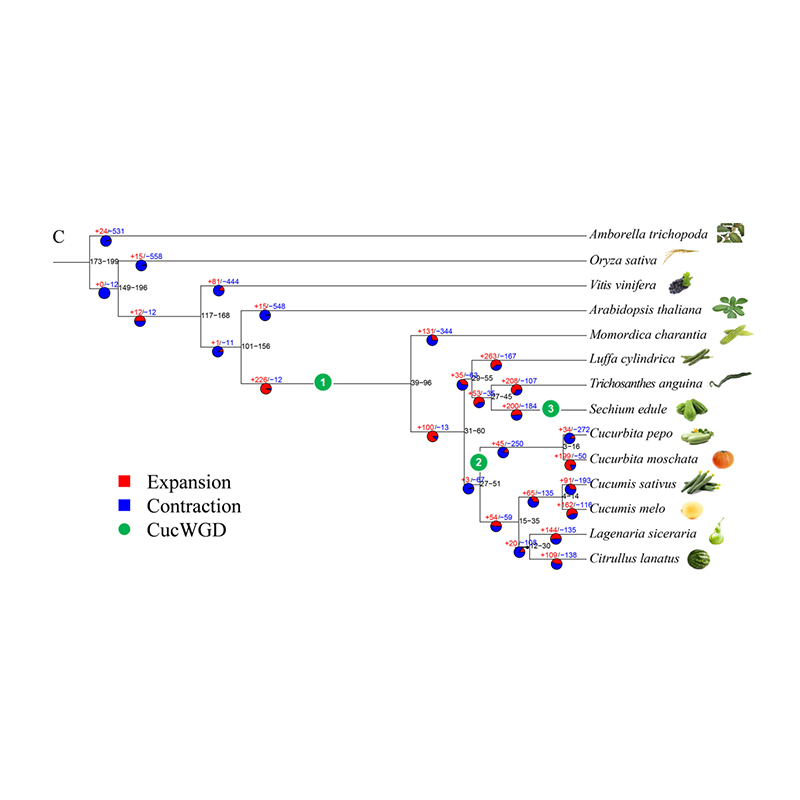
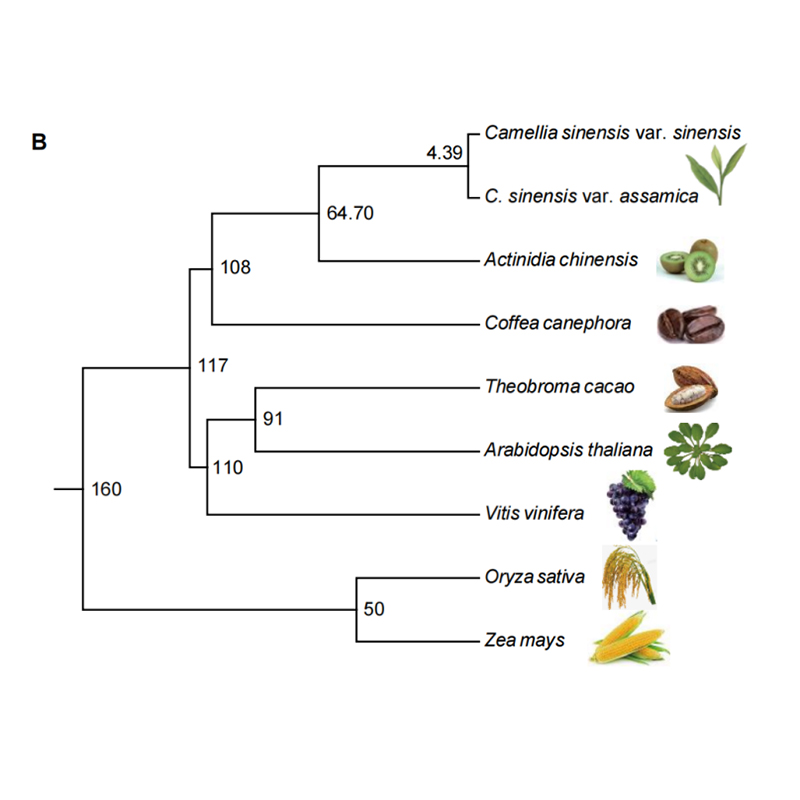
2.Phylogeny og genfamilieanalyse på Chayote (sechium -edule): Ved at analysere Chayote og de andre 13 relaterede arter i genfamilien blev Chayote fundet at være mest relateret til slangekurd (Trichosanthes Anguina). Chayote afledt af slange-kalebas i omkring 27-45 mya og helgenomduplikation (WGD) blev observeret i Chayote i 25 ± 4 MyA, som er den tredje WGD-begivenhed i Cucuibitaceae.
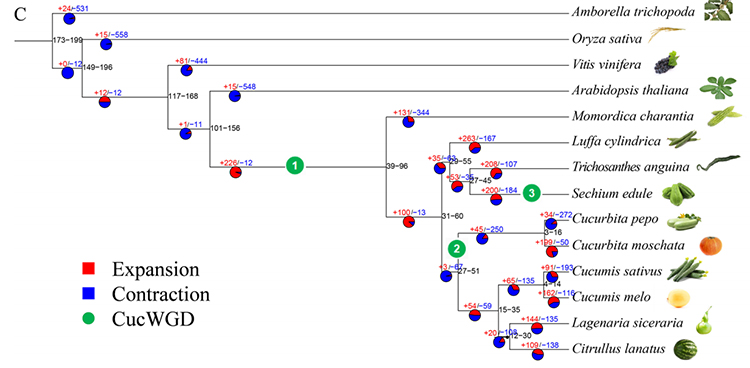
Fu A et al.,Havebrugsforskning, 2021
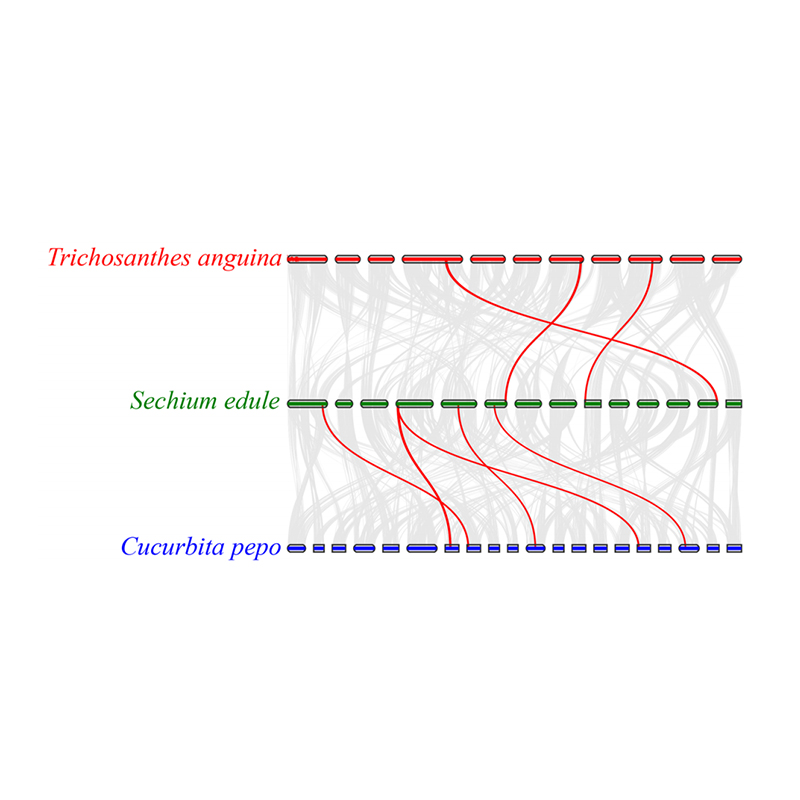
3.Synteny -analyse: Nogle gener relateret til phytohormoner i frugtudvikling blev fundet i Chayote, slange -kalebas og squash. Korrelation mellem chayote og squash er lidt højere end mellem Chayote og Snake Gourd.
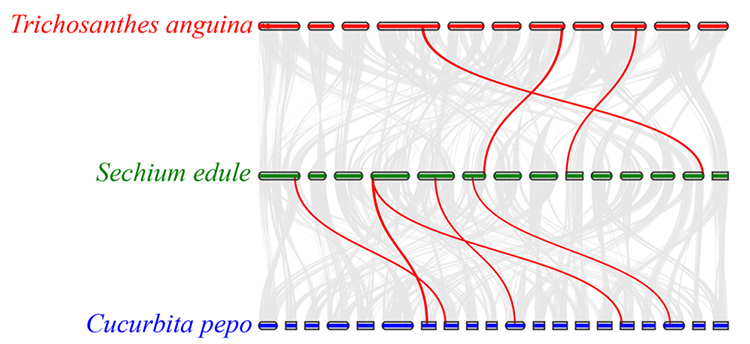
Fu A et al.,Havebrugsforskning, 2021
4.Gene Familieanalyse: KEGG -berigelse om genfamilieudvidelse og sammentrækning i G.Thurberi og G.Davidsonii -genomer vist, at steroidbiosyntese og brassinosteroidbiosyntese -relaterede gener blev udvidet.
Yang Z et al.,BMC Biologi, 2021
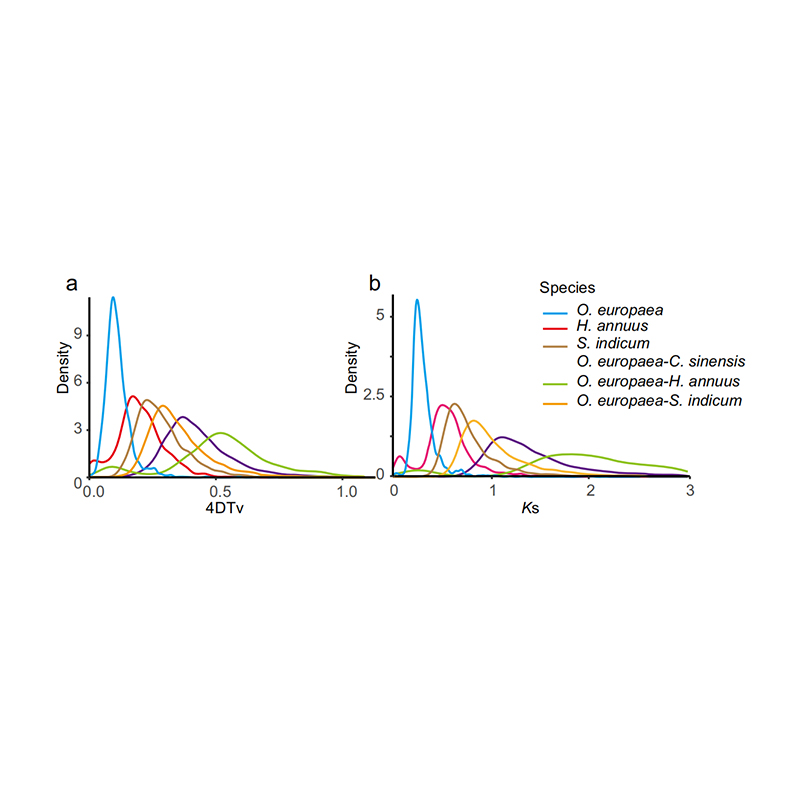
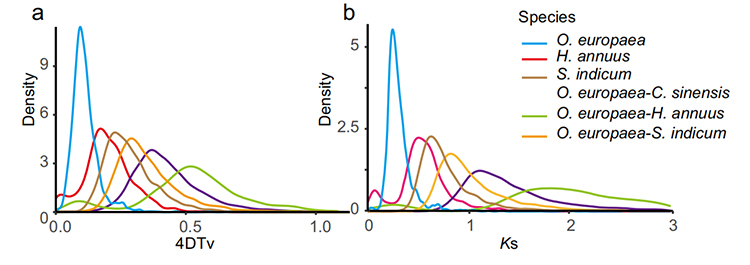
5. Hele genomduplikationsanalyse: 4DTV og KS Distribution Analyse vist hele genomduplikationsbegivenhed. Toppe af intraspecies viste duplikeringsbegivenheder. Toppe af viste interspecies viste speciationbegivenheder. Analysen indikerede, at sammenligning med de andre tre tæt beslægtede arter, O. Europaea gennemgik en storskala -gumplikering for nylig.

Rao G et al.,Havebrugsforskning, 2021
BMK -sag
Rose uden stikkende: Genomisk indsigt knyttet til fugttilpasning
Udgivet: National Science Review, 2021
Sekventeringsstrategi:
'Basye'sTornløs'(R.Wichurainan) genom:
Ca. 93 x pacbio + ca. 90 x nanopore + 267 x Illumina
Nøgleresultater
1. Høj kvalitet R.Wichuraiana genom blev konstrueret under anvendelse af langlæst sekventeringsteknikker, som giver en samling på 530,07 MB (estimeret genomstørrelse var ca. 525,9 MB ved flowcytometri og 525,5 ved genomundersøgelse ; Heterozygositet var omkring 1,03%). Busco estimerede score var 93,9%. Sammenlignet med "gammel rødme" (Haploob) blev kvaliteten og fuldstændigheden af dette genom bekræftet ved basispasen nøjagtighed og LTR-samlingsindeks (LAI = 20.03). R.Wichuraiana -genomet indeholder 32.674 proteinkodende gener.
2.Multi-omics fælles analyse, bestående af komparativ genomik, transkriptomik, QTL-analyse af genetisk population, afslørede den afgørende specifikation mellem R. wichuraiana og Rosa chinensis. Ekspressionsvariation af relaterede gener i QTL var sandsynligvis forbundet med stamprikkle -mønstring.

Sammenlignende genomik anaysis mellem basyes tornløse og rosa chinensis inklusive synteny -analyse, genfamilieklynge, ekspansion og sammentrækningsanalyse afslørede et stort antal variationer, der relaterede til afgørende træk i roser. Den unikke ekspansion i NAC og FAR1/FRS -genfamilien var meget sandsynligt forbundet med modstand mod sort plet.
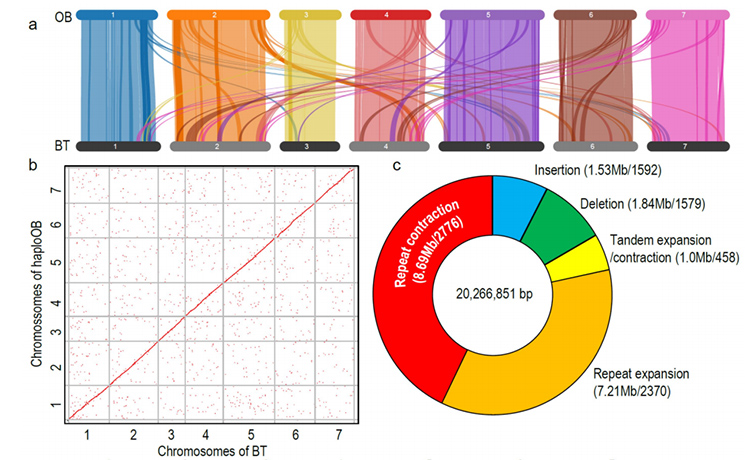

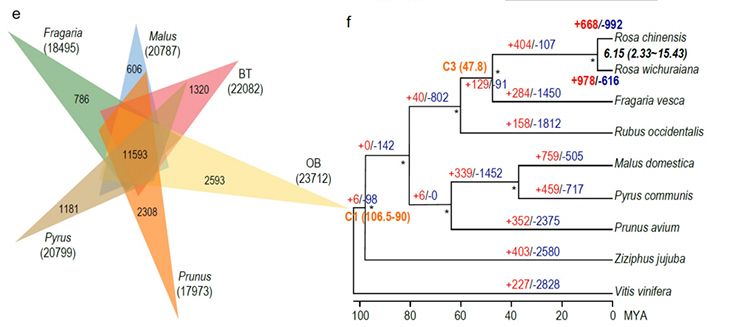
Sammenlignende genomisk analyse mellem BT og Haploob -genomer.
Zhong, M., et al. “Rose uden Prickle: Genomisk indsigt knyttet til fugttilpasning”National Science Review, 2021;, NWAB092.