BMKMANU S1000 Spatial Transcriptome
BMKMANU S1000 Spatial Transcriptome Technical Scheme
Funktioner
● Opløsning: 5 µM
● Spotdiameter: 2,5 µM
● Antal spots: ca. 2 mio
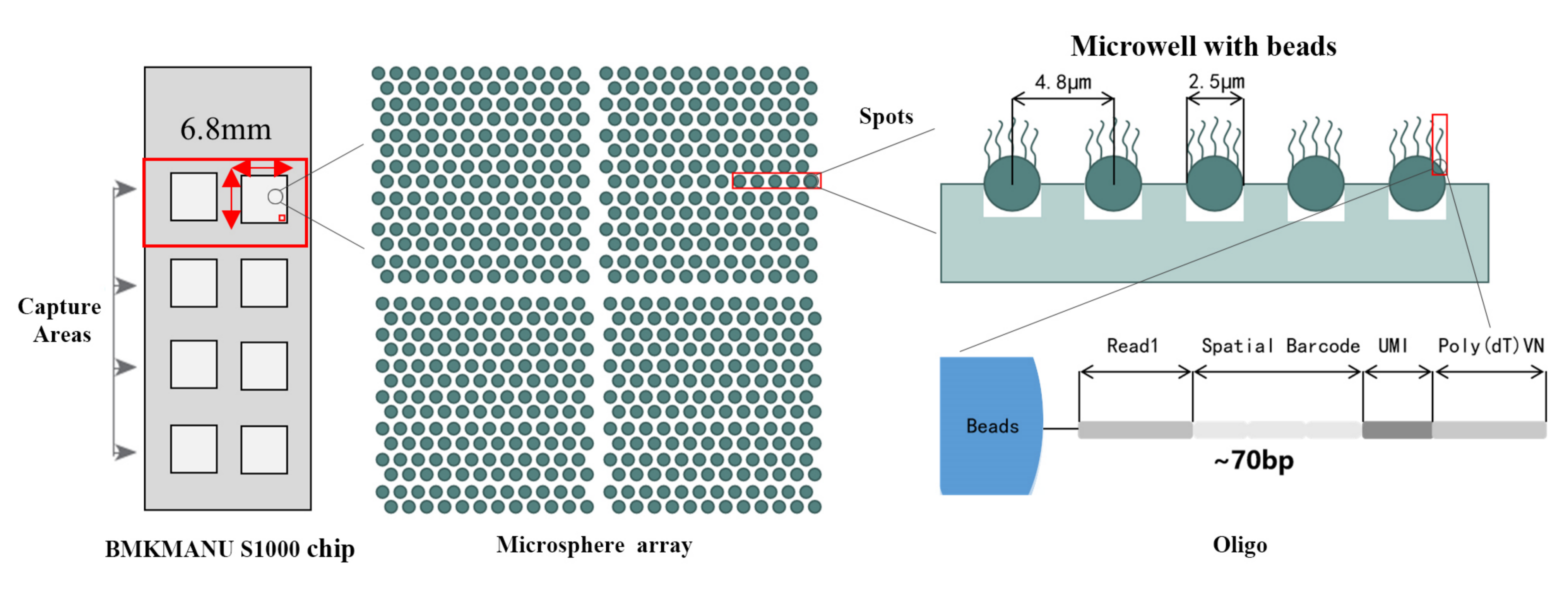
● 3 mulige optageområdeformater: 6,8 mm * 6,8 mm, 11 mm * 11 mm eller 15 mm * 20 mm
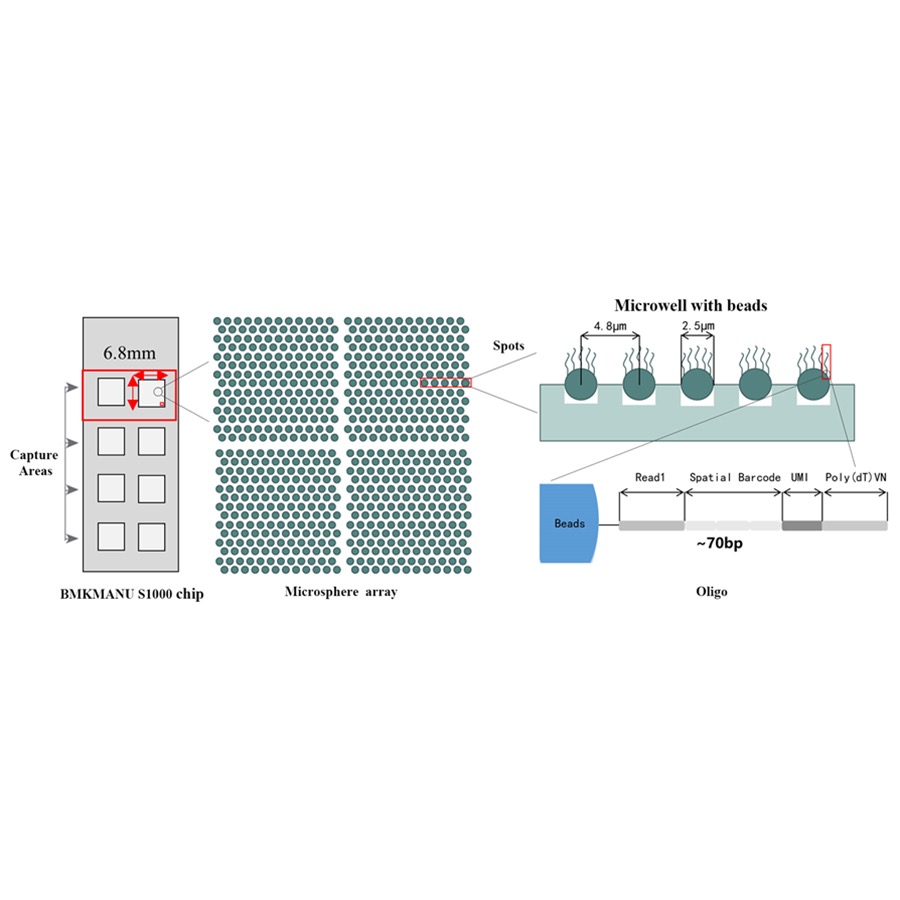
● Hver stregkodet perle er fyldt med primere sammensat af 4 sektioner:
poly(dT)-hale til mRNA-priming og cDNA-syntese
Unik Molecular Identifier (UMI) til at korrigere amplifikationsbias
Rumlig stregkode
Bindingssekvens af delvis læst 1 sekventeringsprimer
● H&E og fluorescerende farvning af snit
● Mulighed for brugcellesegmenteringsteknologi: integration af H&E-farvning, fluorescerende farvning og RNA-sekventering for at bestemme grænserne for hver celle og korrekt tildele genekspression til hver celle.
Fordele ved BMKMANU S1000
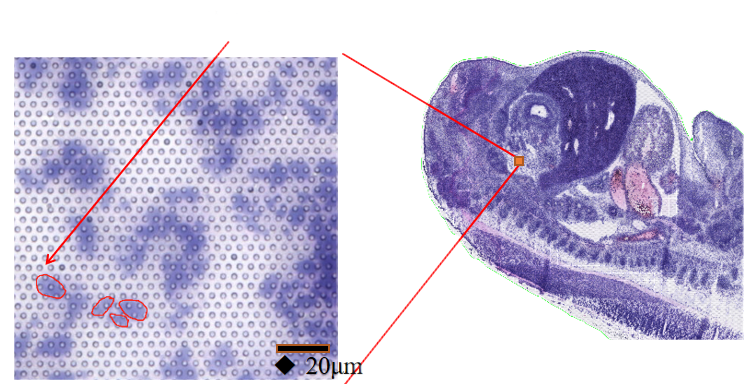
●Subcellulær opløsning: Hvert indfangningsområde indeholdt >2 millioner rumlige stregkodede pletter med en diameter på 2,5 µm og en afstand på 5 µm mellem spotcentre, hvilket muliggjorde rumlig transkriptomanalyse med subcellulær opløsning (5 µm).
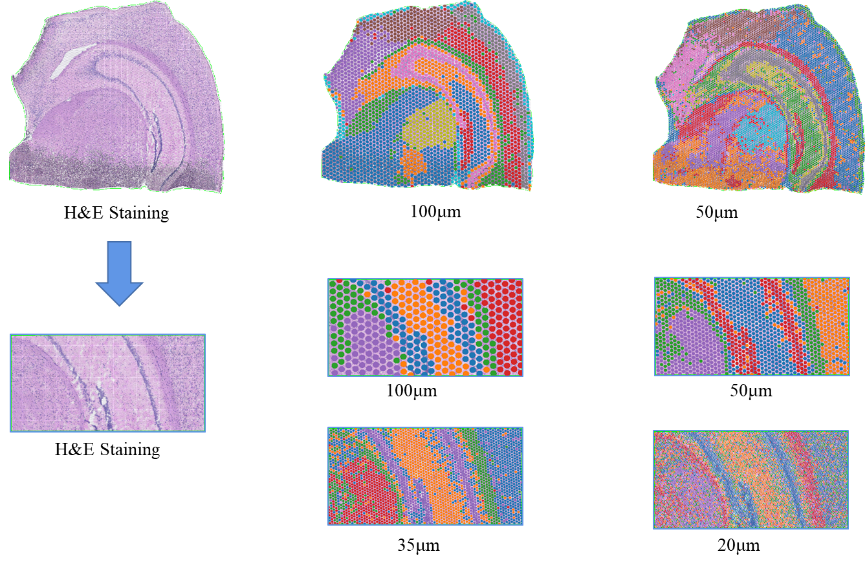
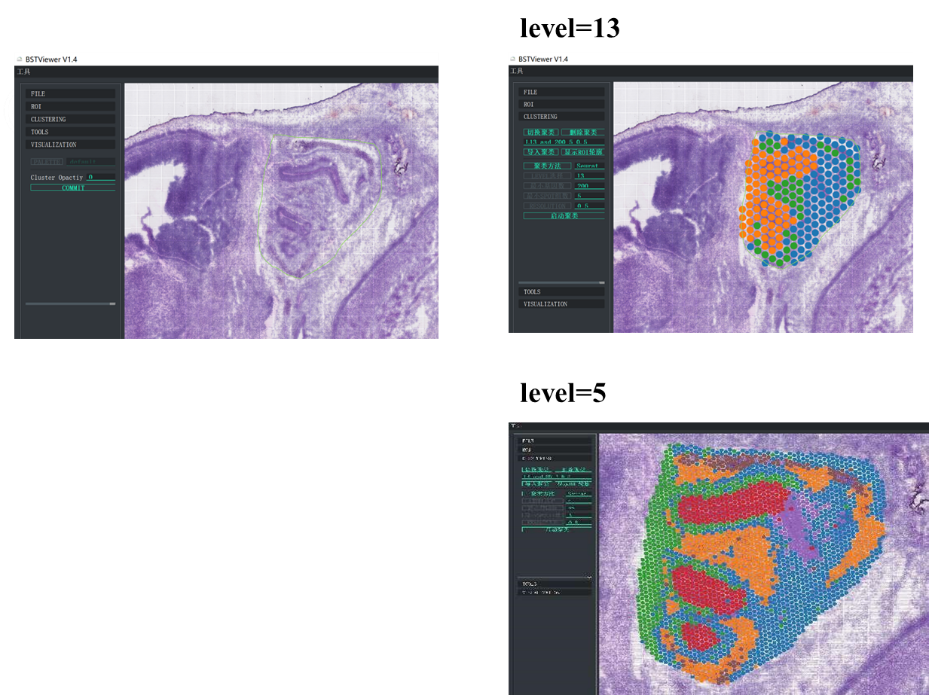
●Multi-level opløsningsanalyse:Fleksibel multi-level analyse fra 100 μm til 5 μm for at løse forskellige vævstræk ved optimal opløsning.
● Mulighed for at bruge "Tre i ét dias" cellesegmenteringsteknologi:Ved at kombinere fluorescensfarvning, H&E-farvning og RNA-sekventering på et enkelt objektglas giver vores "tre-i-en" analysealgoritme mulighed for at identificere cellegrænser for efterfølgende cellebaseret transkriptomik.
●Kompatibel med flere sekventeringsplatforme: Både NGS og langlæst sekvensering tilgængelig.
●Fleksibelt design af 1-8 Active Capture Area: Størrelsen på optagelsesområdet er fleksibel, idet det er muligt at bruge 3 formater (6,8 mm * 6,8 mm., 11 mm * 11 mm og 15 mm * 20 mm)
●One-stop service: Det integrerer alle erfarings- og færdighedsbaserede trin, herunder kryo-sektionering, farvning, vævsoptimering, rumlig stregkodning, biblioteksforberedelse, sekventering og bioinformatik.
●Omfattende bioinformatik og brugervenlig visualisering af resultater:Pakken indeholder 29 analyser og 100+ figurer af høj kvalitet, kombineret med brugen af internt udviklet software til at visualisere og tilpasse celleopdeling og spot-klynger.
●Tilpasset dataanalyse og visualisering: tilgængelig for forskellige forskningsanmodninger
●Højt kvalificeret teknisk team: med erfaring i over 250 vævstyper og 100+ arter, herunder mennesker, mus, pattedyr, fisk og planter.
●Opdateringer i realtid på hele projektet: med fuld kontrol over eksperimentelle fremskridt.
●Valgfri fælles analyse med enkeltcellet mRNA-sekventering
Service specifikationer
|
Prøve Krav
| Bibliotek |
Sekvenseringsstrategi
| Data anbefales | Kvalitetskontrol |
| OCT-indlejrede kryoprøver, 3 blokke pr. prøve | S1000 cDNA-bibliotek | Illumina PE150 (andre tilgængelige platforme) | 100K PE-aflæsninger pr. 100 uM (60-150 Gb) | RIN>7 |
For flere detaljer om prøveforberedelsesvejledning og serviceworkflow er du velkommen til at tale med enBMKGENE ekspert
Service Work Flow
I prøveforberedelsesfasen udføres et indledende bulk-RNA-ekstraktionsforsøg for at sikre, at et RNA af høj kvalitet kan opnås. I vævsoptimeringsstadiet farves og visualiseres snittene, og permeabiliseringsbetingelserne for mRNA-frigivelse fra væv optimeres. Den optimerede protokol anvendes derefter under bibliotekskonstruktion, efterfulgt af sekventering og dataanalyse.
Det komplette service-workflow involverer opdateringer i realtid og klientbekræftelser for at opretholde en responsiv feedback-loop, hvilket sikrer problemfri projektudførelse.
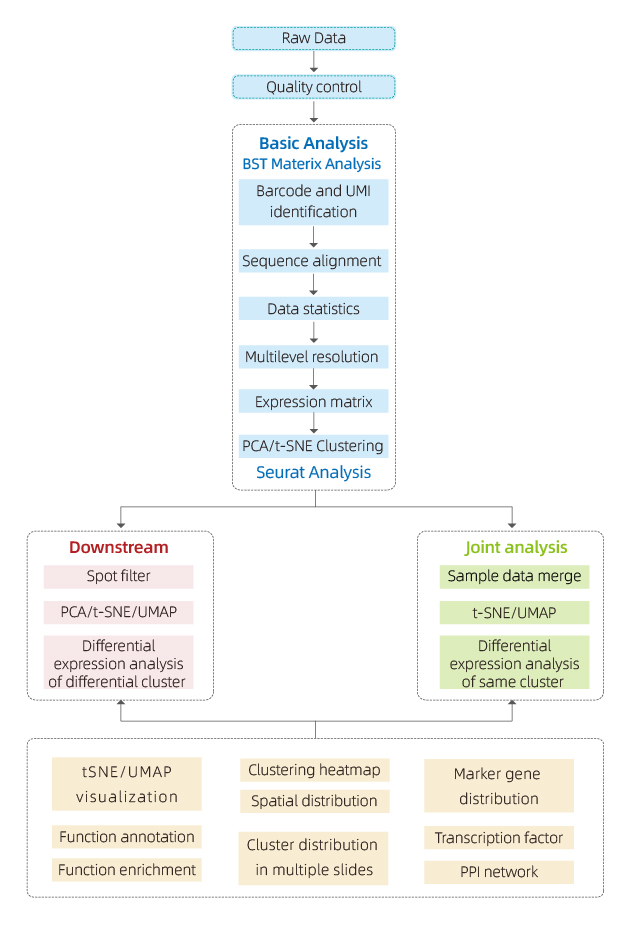
Dataene genereret af BMKMANU S1000 analyseres ved hjælp af softwaren "BSTMatrix", som er uafhængigt designet af BMKGENE, der genererer en genekspressionsmatrix. Derfra genereres en standardrapport, der inkluderer datakvalitetskontrol, intern prøveanalyse og inter-gruppe analyse.
● Datakvalitetskontrol:
Dataoutput og kvalitetsscorefordeling
Gendetektion pr. plet
Vævsdækning
● Indre prøveanalyse:
Genrigdom
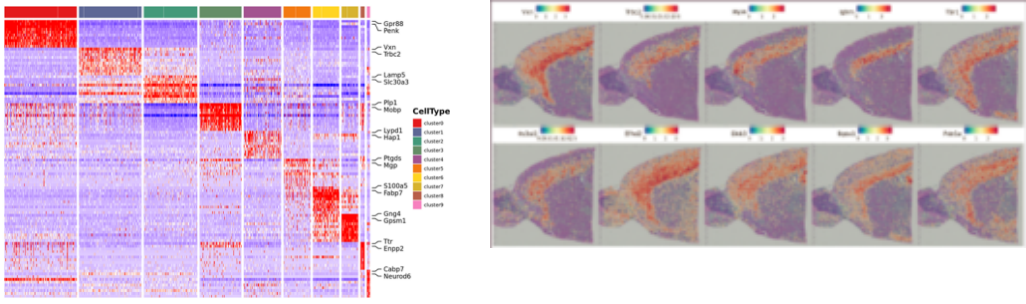
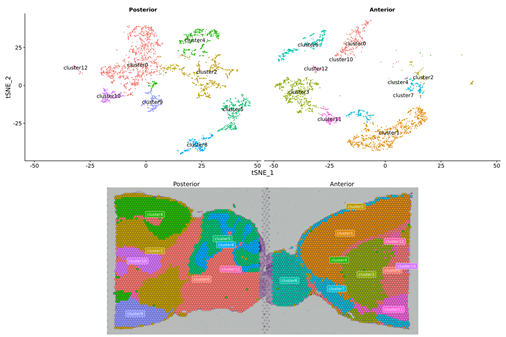
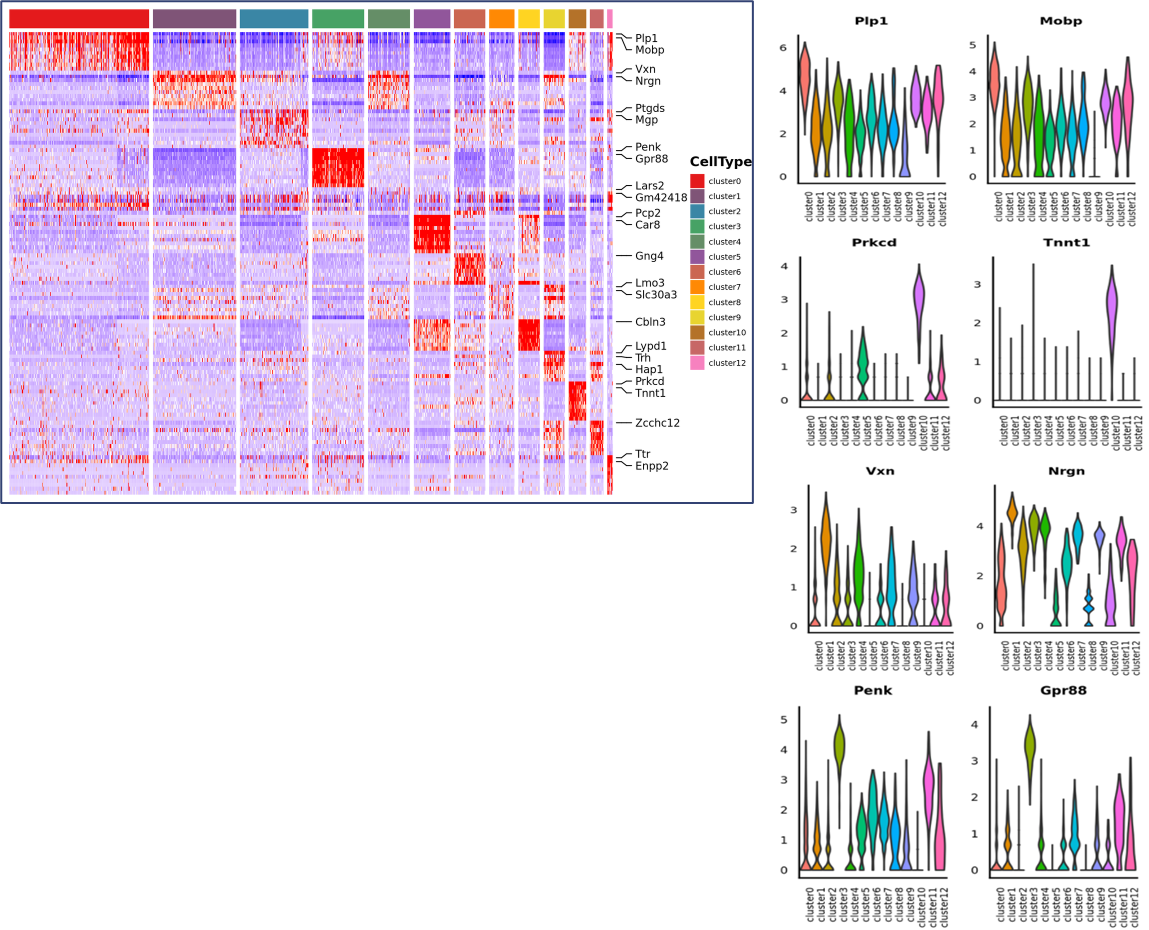
Spot clustering, herunder reduceret dimensionsanalyse
Differentiel ekspressionsanalyse mellem klynger: identifikation af markørgener
Funktionel annotering og berigelse af markørgener
● Inter-gruppe analyse:
Re-kombination af pletter fra både prøver (f.eks. syge og kontrol) og re-cluster
Identifikation af markørgener for hver klynge
Funktionel annotering og berigelse af markørgener
Differentiel Udtryk af den samme klynge mellem grupper
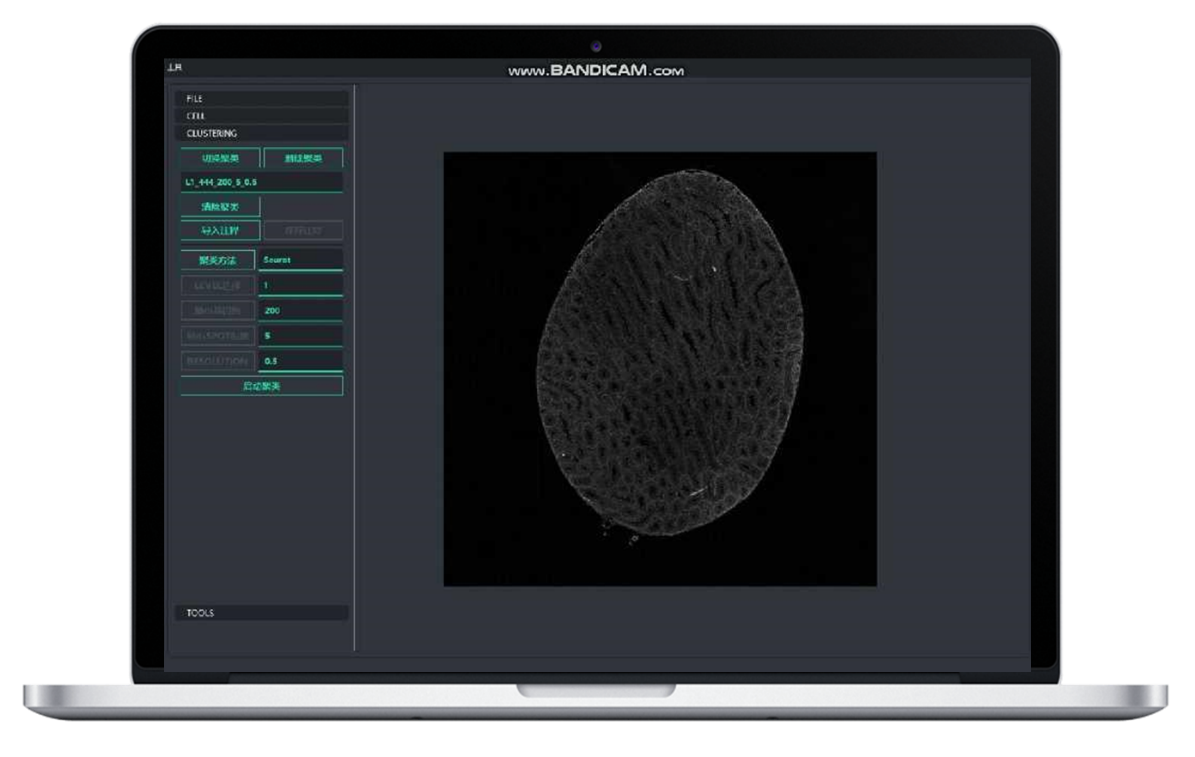
Derudover er BMKGENE udviklet "BSTViewer" et brugervenligt værktøj, der gør det muligt for brugeren at visualisere genekspressionen og pletklynger i forskellige opløsninger.
BMKGene udviklede software til brugervenlig visualisering
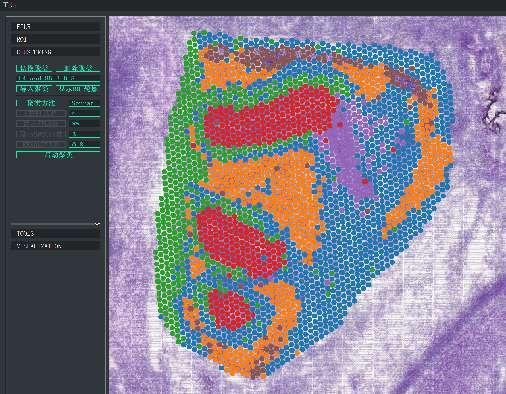
BSTViewer spot clustering ved multi-level opløsning
BSTcellViewer: automatisk og manuel celleopdeling
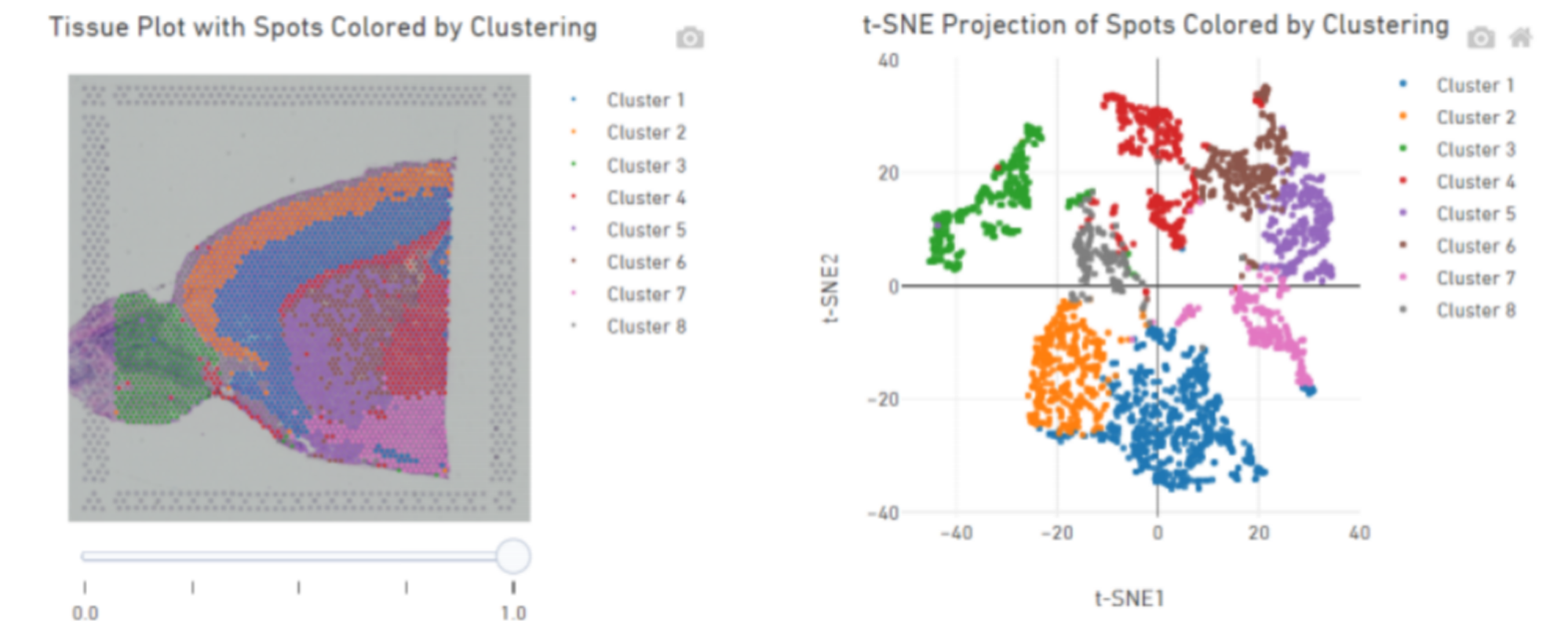
Analyse af indre prøve
Punktklynger:
Markørgener identifikation og rumlig fordeling:
Inter-gruppe Analyse
Datakombination fra begge grupper og re-cluster:
Markørgener for nye klynger:
Udforsk de fremskridt, der er lettet af BMKGenes rumlige transkriptomiske tjenester med BMKManu S1000-teknologien i denne fremhævede publikation:
Song, X. et al. (2023) 'Spatial transcriptomics afslører lys-inducerede chlorenchyma celler involveret i at fremme skudregenerering i tomat callus',Proceedings of the National Academy of Sciences of the United States of America120(38), s. e2310163120. doi: 10.1073/pnas.2310163120
Du, Y. et al. (2023) 'Systematisk sammenligning af sekventeringsbaserede rumlige transkriptomiske metoder',bioRxiv, s. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.