10x Genomics Visium Spatial Transcriptome
Teknisk ordning

Funktioner
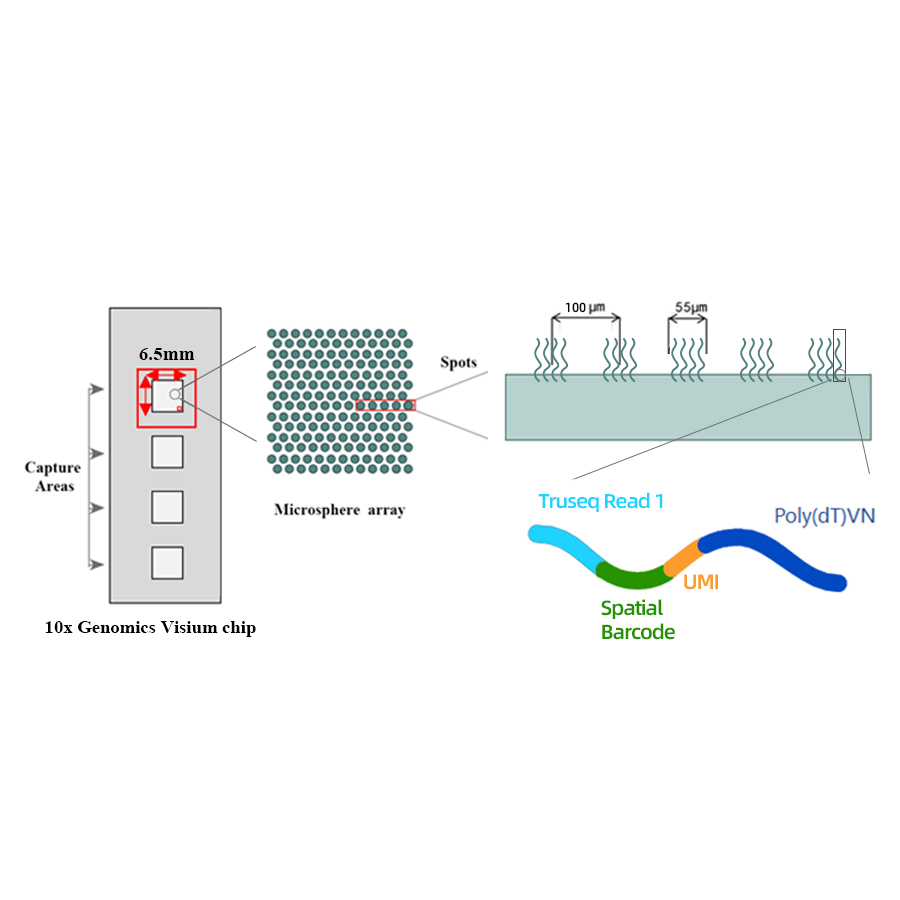
● Opløsning: 100 µM
● Spotdiameter: 55 µM
● Antal pladser: 4992
● Optagelsesområde: 6,5 x 6,5 mm
● Hvert stregkodested er fyldt med primere sammensat af 4 sektioner:
- poly(dT)-hale til mRNA-priming og cDNA-syntese
- Unik Molecular Identifier (UMI) til at korrigere amplifikationsbias
- Rumlig stregkode
- Bindingssekvens af delvis læst 1 sekventeringsprimer
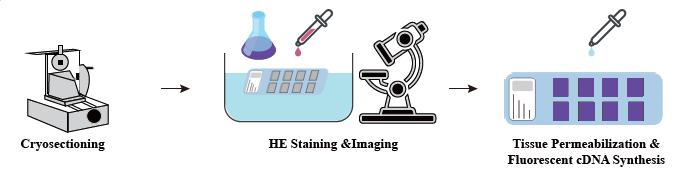
● H&E-farvning af sektioner
Fordele
●One-stop service: integrerer alle erfarings- og færdighedsbaserede trin, herunder kryo-sektionering, farvning, vævsoptimering, rumlig stregkodning, biblioteksforberedelse, sekventering og bioinformatik.
● Højt kvalificeret teknisk team: med erfaring i over 250 vævstyper og 100+ arter, herunder mennesker, mus, pattedyr, fisk og planter.
●Opdatering i realtid på hele projektet: med fuld kontrol over eksperimentelle fremskridt.
●Omfattende standard bioinformatik:Pakken indeholder 29 analyser og 100+ højkvalitetsfigurer.
●Tilpasset dataanalyse og visualisering: tilgængelig for forskellige forskningsanmodninger.
●Valgfri fælles analyse med enkeltcellet mRNA-sekventering
Specifikationer
| Prøvekrav | Bibliotek | Sekvenseringsstrategi | Data anbefales | Kvalitetskontrol |
| OCT-indlejrede kryoprøver (Optimal diameter: ca. 6x6x6 mm³) 2 blokke pr. prøve | 10X Visium cDNA-bibliotek | Illumina PE150 | 50K PE-aflæsninger pr. spot (60 Gb) | RIN > 7 |
For flere detaljer om prøveforberedelsesvejledning og serviceworkflow er du velkommen til at tale med enBMKGENE ekspert
Service Workflow
I prøveforberedelsesfasen udføres et indledende bulk-RNA-ekstraktionsforsøg for at sikre, at et RNA af høj kvalitet kan opnås. I vævsoptimeringsstadiet farves og visualiseres snittene, og permeabiliseringsbetingelserne for mRNA-frigivelse fra væv optimeres. Den optimerede protokol anvendes derefter under bibliotekskonstruktion, efterfulgt af sekventering og dataanalyse.
Det komplette service-workflow involverer opdateringer i realtid og klientbekræftelser for at opretholde en responsiv feedback-loop, hvilket sikrer problemfri projektudførelse.

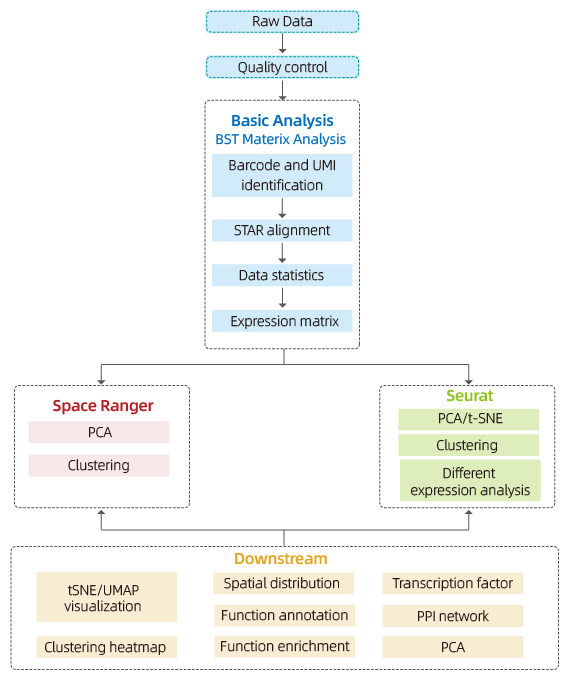
Indeholder følgende analyse:
Datakvalitetskontrol:
o Dataoutput og kvalitetsscorefordeling
o Gendetektion pr. plet
o Vævsdækning
Analyse af indre prøve:
o Genrigdom
o Spot clustering, herunder reduceret dimensionsanalyse
o Differentiel ekspressionsanalyse mellem klynger: identifikation af markørgener
o Funktionel annotering og berigelse af markørgener
Inter-gruppe Analyse
o Re-kombination af pletter fra både prøver (f.eks. syge og kontrol) og re-cluster
o Identifikation af markørgener for hver klynge
o Funktionel annotering og berigelse af markørgener
o Differentiel udtryk for den samme klynge mellem grupper
Analyse af indre prøve
Pletklynger

Markørgener identifikation og rumlig fordeling


Inter-gruppe Analyse
Datakombination fra begge grupper og re-cluster
Markørgener for nye klynger
Udforsk de fremskridt, der er lettet af BMKGenes rumlige transcriptomics-tjeneste af 10X Visium I disse fremhævede publikationer:
Chen, D. et al. (2023) 'mthl1, en potentiel Drosophila-homolog af pattedyrsadhæsions-GPCR'er, er involveret i antitumorreaktioner på injicerede onkogene celler i fluer',Proceedings of the National Academy of Sciences of the United States of America120(30), s. e2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'STEEL muliggør afgrænsning i høj opløsning af spatiotemporale transkriptomiske data',Briefinger i bioinformatik, 24(2), s. 1-10. doi: 10.1093/BIB/BBAD068.
Liu, C. et al. (2022) 'Et spatiotemporalt atlas over organogenese i udviklingen af orkidéblomster',Nukleinsyreforskning, 50(17), s. 9724-9737. doi: 10.1093/NAR/GKAC773.
Wang, J. et al. (2023) 'Integration Spatial Transcriptomics and Single-nucleus RNA Sequencing afslører de potentielle terapeutiske strategier for uterin leiomyom',International Journal of Biological Sciences, 19(8), s. 2515-2530. doi: 10.7150/IJBS.83510.