Malá RNA sekvenování-ilumina
Funkce
● Příprava knihovny zahrnuje krok výběru velikosti
● Bioinformatická analýza soustředěná kolem predikce miRNA a jejich cílů
Výhody služeb
●Komplexní analýza bioinformatiky:Povolení identifikace známých i nových miRNA, identifikace cílů miRNA a odpovídající funkční anotace a obohacení několika databázemi (KEGG, GO)
●Přísná kontrola kvality: Implementujeme základní kontrolní body ve všech fázích, od přípravy vzorku a knihovny po sekvenování a bioinformatiku. Toto pečlivé monitorování zajišťuje dodávání trvale vysoce kvalitních výsledků.
●Podpora po prodeji: Náš závazek přesahuje dokončení projektu s 3měsíční obdobím servisního období. Během této doby nabízíme sledování projektů, pomoc při řešení problémů a sezení otázek a odpovědí k řešení jakýchkoli dotazů souvisejících s výsledky.
●Rozsáhlé odborné znalosti: S výsledkem úspěšného uzavření nad několika projekty SRNA pokrývající více než 300 druhů v různých výzkumných oblastech přináší náš tým do každého projektu bohaté zkušenosti.
Ukázkové požadavky a doručení
| Knihovna | Platforma | Doporučená data | Data QC |
| Vybraná velikost | Illumina Se50 | 10m-20m čtení | Q30≥85% |
Ukázkové požadavky:
Nukleotidy:
| Konc. (Ng/μl) | Částka (μg) | Čistota | Integrita |
| ≥ 80 | ≥ 0,8 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Omezená nebo žádná kontaminace proteinu nebo DNA na gelu. | Rin≥6.0; 5,0≥28s/18s≥1,0; omezená nebo žádná výška výchozího linie |
● Rostliny:
Kořen, stonek nebo okvětní lístek: 450 mg
List nebo semeno: 300 mg
Ovoce: 1,2 g
● Zvíře:
Srdce nebo střevo: 450 mg
Viscera nebo mozek: 240 mg
Sval: 600 mg
Kosti, vlasy nebo kůže: 1,5 g
● Artropody:
Hmyz: 9G
Crustacea: 450 mg
● Celá krev: 2 trubice
● Buňky: 106 buňky
● Sérum a plazma:6 ml
Doporučené doručení vzorku
Kontejner: 2 ml odstředivé trubice (cínová fólie se nedoporučuje)
Značení vzorků: skupina+replikujte EG A1, A2, A3; B1, B2, B3.
Zásilka:
1. Suchý led: Vzorky musí být zabaleny do pytlů a pohřbeny do suchého ledu.
2. Rnastabilní zkumavky: Vzorky RNA mohou být vysušeny ve stabilizační trubici RNA (např. RNASTABLE®) a dodávány v pokojové teplotě.
Tok servisní práce

Návrh experimentu

Ukázka doručení

Extrakce RNA

Knihovna konstrukce

Sekvenování

Analýza dat

Poprodejní služby
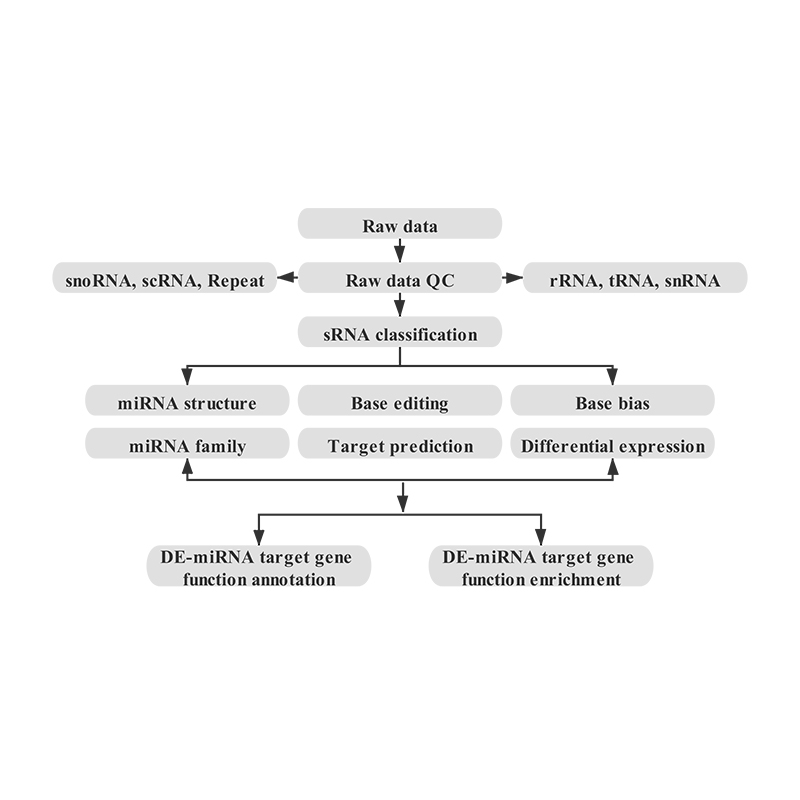
Bioinformatika
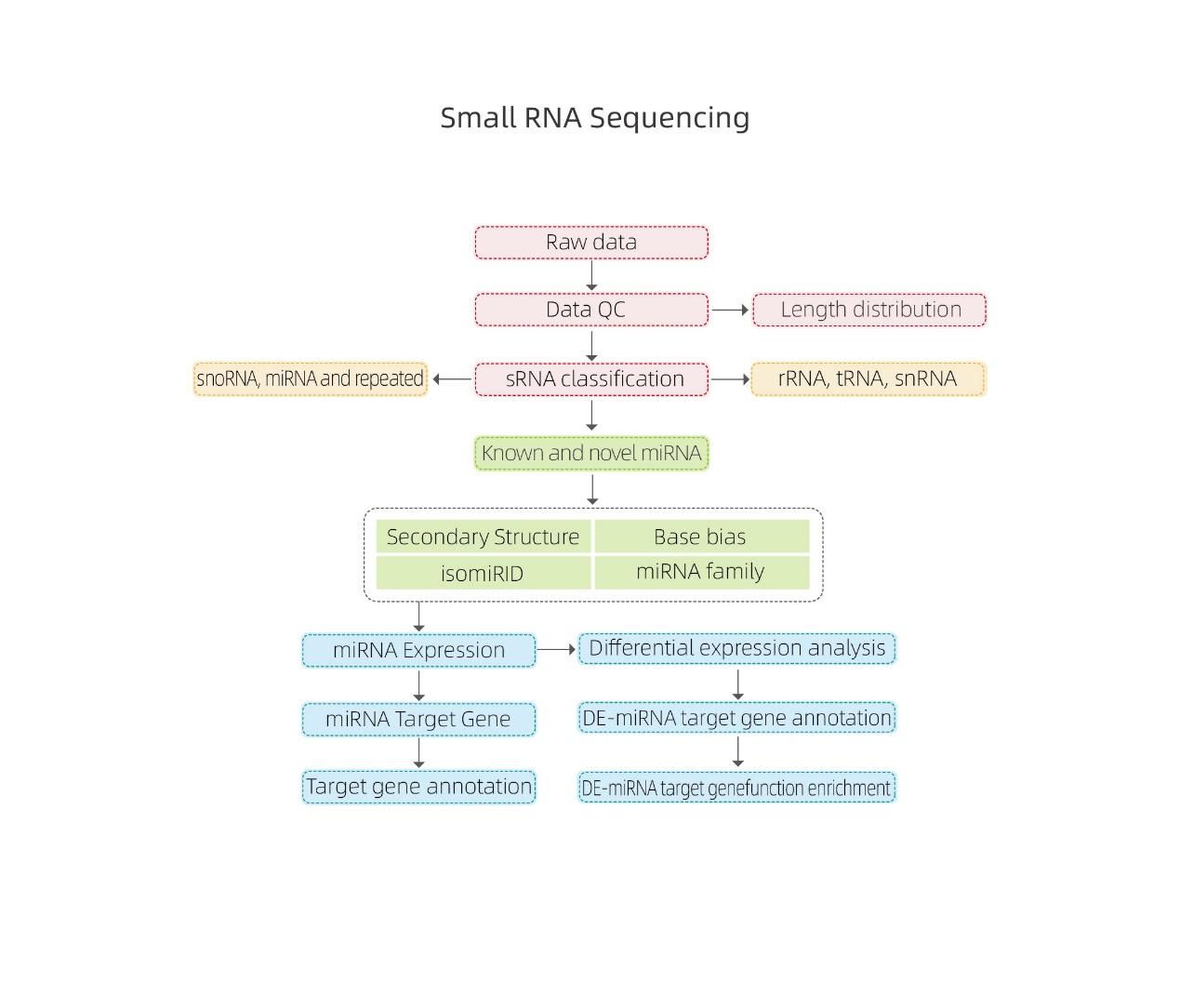 ● Řízení kvality dat nezpracovaných
● Řízení kvality dat nezpracovaných
● Klasifikace sRNA
● Zarovnání s referenčním genomem
● Identifikace známé a nové miRNA
● Diferenciální analýza exprese miRNA
● Funkční anotace cílů miRNA
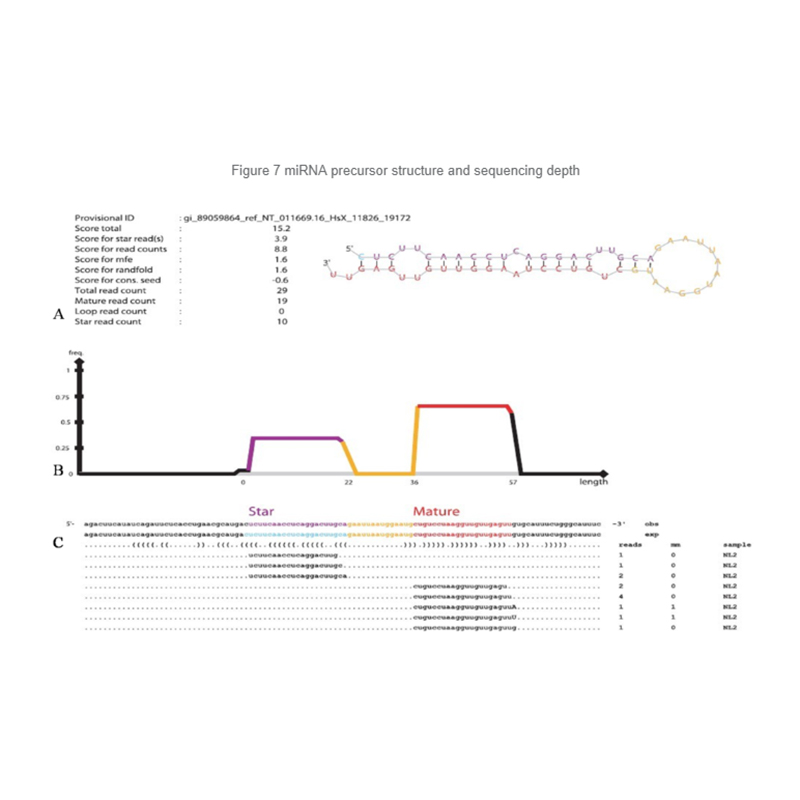
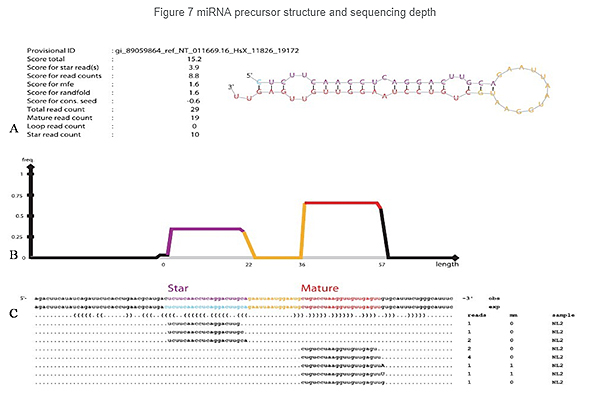
Identifikace miRNA: Struktura a hloubka
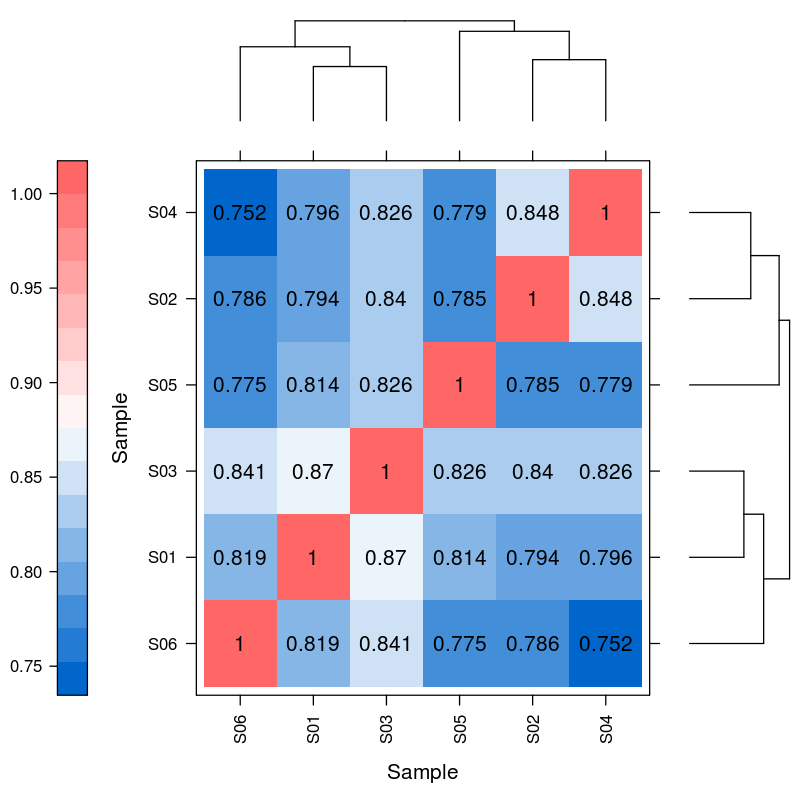
Diferenciální vyjádření miRNA - Hiearchical Clustering
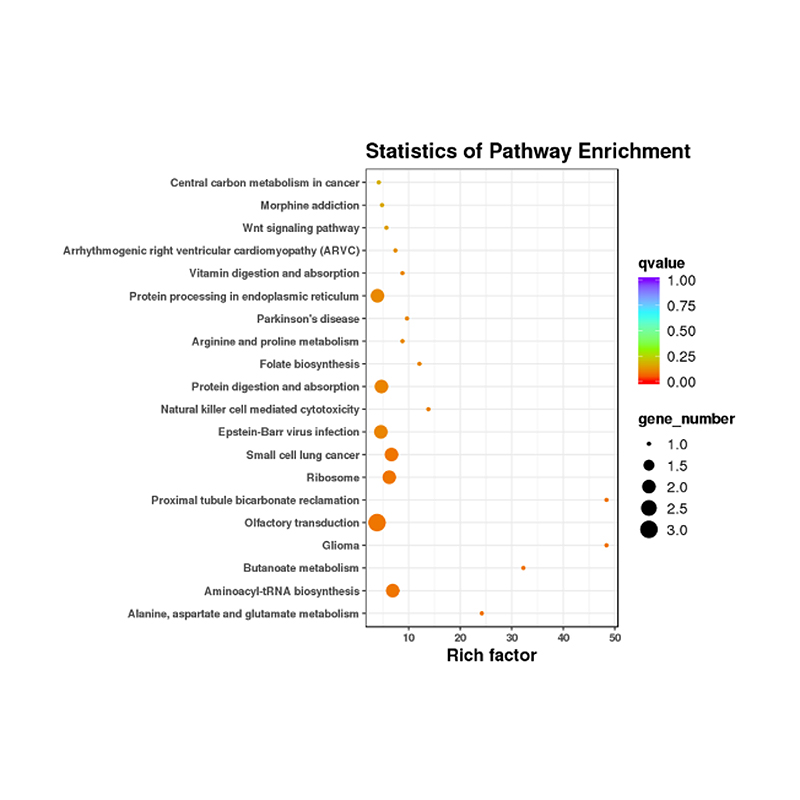
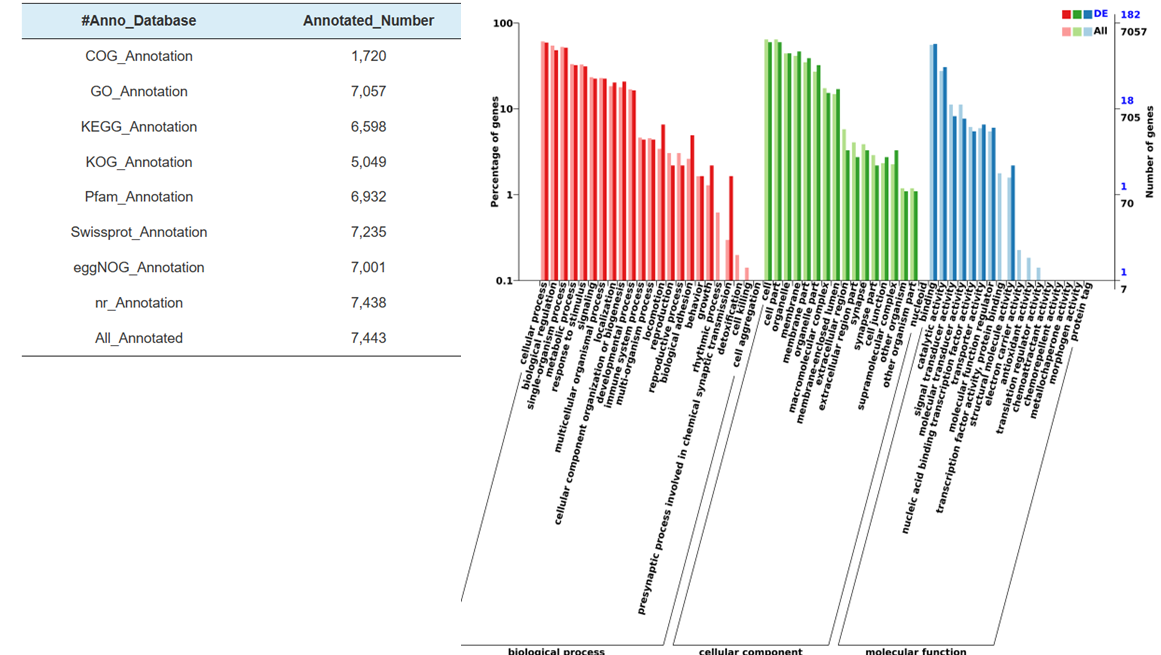
Funkční anotace cíle odlišně exprimovaných miRNA
Prozkoumejte výzkumné pokroky usnadněné sekvenčními službami SRNA BMKGene prostřednictvím kurátorské sbírky publikací.
Chen, H. a kol. (2023) „Virové infekce inhibují biosyntézu a fotosyntézu saponinu v Panax notoginseng“, fyziologii rostlin a biochemie, 203, s. 203, s. 108038. Doi: 10.1016/j.Plaphy.2023.108038.
Li, H. a kol. (2023) „Protein obsahující doménu obsahující FYVE FYVE FREEIN Free1 se spojuje s mikroprocesorovými složkami k potlačení biogeneze miRNA“, zprávy EMBO, 24 (1). doi: 10.15252/emmpt.202255037/suppl_file/emmpt202255037-SUP-0004-SDATAFIG4.TIF.
Yu, J. et al. (2023) „MikroRNA AME-BANTAM-3P řídí vývoj larevných kusů zaměřením na více epidermálních domén podobných růstovým faktorem 8 (MEGF8) v včele, APIS Mellifera“, International Journal of Molecular Sciences, 24 (6), p p . 5726. Doi: 10,3390/ijms24065726/s1.
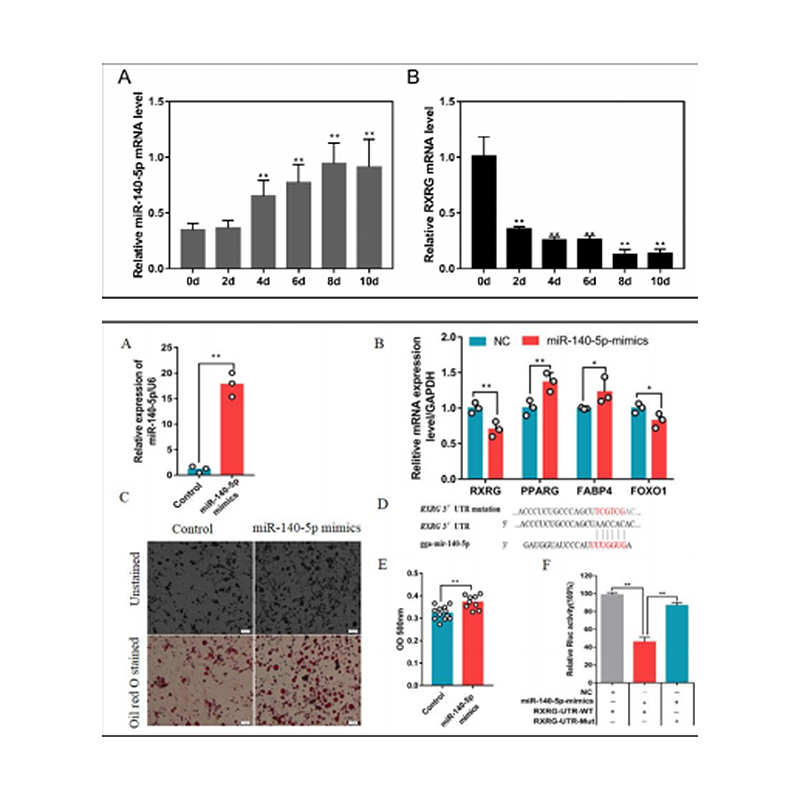
Zhang, M. a kol. (2018) „Integrovaná analýza miRNA a genů spojených s kvalitou masa ukazuje, že GGA-MIR-140-5p ovlivňuje intramuskulární ukládání tuků u kuřat, buněčné fyziologie a biochemie, 46 (6), s. 2421–2433. doi: 10.1159/000489649.