Sekvenování mRNA sekvenční nanopory plné délky
Funkce
● Zachycení poly-A mRNA následované syntézou cDNA a přípravou knihovny
● Sekvenování přepisů po celé délce
● Bioinformatická analýza založená na zarovnání s referenčním genomem
● Bioinformatická analýza zahrnuje nejen expresi na úrovni genu a izoformy, ale také analýzu lncRNA, genových fúzí, polyadenylace a struktury genů
Výhody služeb
●Kvantifikace exprese na úrovni izoformy: Povolení podrobné a přesné analýzy exprese, odhalení změny, která může být maskována při analýze celé exprese genu
●Snížené požadavky na údaje:Ve srovnání se sekvenováním nové generace (NGS) vykazuje sekvenování Nanopore nižší požadavky na údaje, což umožňuje ekvivalentní úrovně nasycení kvantifikace genové exprese menšími daty.
●Vyšší přesnost kvantifikace exprese: jak na úrovni genu, tak izoformy
●Identifikace dalších transkriptomických informací: Alternativní polyadenylace, fúzní geny a LCNRNA a jejich cílové geny
●Rozsáhlé odborné znalosti: Náš tým přináší do každého projektu bohaté zkušenosti, poté, co dokončil více než 850 projektů transkriptomu nanopore a zpracovával přes 8 000 vzorků.
●Podpora po prodeji: Náš závazek přesahuje dokončení projektu s 3měsíční obdobím servisního období. Během této doby nabízíme sledování projektů, pomoc při řešení problémů a sezení otázek a odpovědí k řešení jakýchkoli dotazů souvisejících s výsledky.
Ukázkové požadavky a doručení
| Knihovna | Sekvenční strategie | Doporučená data | Kontrola kvality |
| Poly A obohacené | Illumina PE150 | 6/12 GB | Průměrné skóre kvality: Q10 |
Ukázkové požadavky:
Nukleotidy:
| Konc. (Ng/μl) | Částka (μg) | Čistota | Integrita |
| ≥ 100 | ≥ 1,0 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Omezená nebo žádná kontaminace proteinu nebo DNA na gelu. | Pro rostliny: Rin≥7,0; Pro zvířata: Rin≥7,5; 5,0≥28s/18s≥1,0; omezená nebo žádná výška výchozího linie |
● Rostliny:
Kořen, stonek nebo okvětní lístek: 450 mg
List nebo semeno: 300 mg
Ovoce: 1,2 g
● Zvíře:
Srdce nebo střevo: 300 mg
Viscera nebo mozek: 240 mg
Sval: 450 mg
Kosti, vlasy nebo kůže: 1G
● Artropody:
Hmyz: 6G
Crustacea: 300 mg
● Celá krev: 1 trubice
● Buňky: 106 buňky
Doporučené doručení vzorku
Kontejner: 2 ml odstředivé trubice (cínová fólie se nedoporučuje)
Značení vzorků: skupina+replikujte EG A1, A2, A3; B1, B2, B3.
Zásilka:
1. Suchý led: Vzorky musí být zabaleny do pytlů a pohřbeny do suchého ledu.
2. Rnastabilní zkumavky: Vzorky RNA mohou být vysušeny ve stabilizační trubici RNA (např. RNASTABLE®) a dodávány v pokojové teplotě.
Tok servisní práce
Nukleotidy:

Ukázka doručení

Knihovna konstrukce

Sekvenování

Analýza dat

Poprodejní služby
Tok servisní práce
Tkáň:

Návrh experimentu

Ukázka doručení

Extrakce RNA

Knihovna konstrukce

Sekvenování

Analýza dat

Poprodejní služby
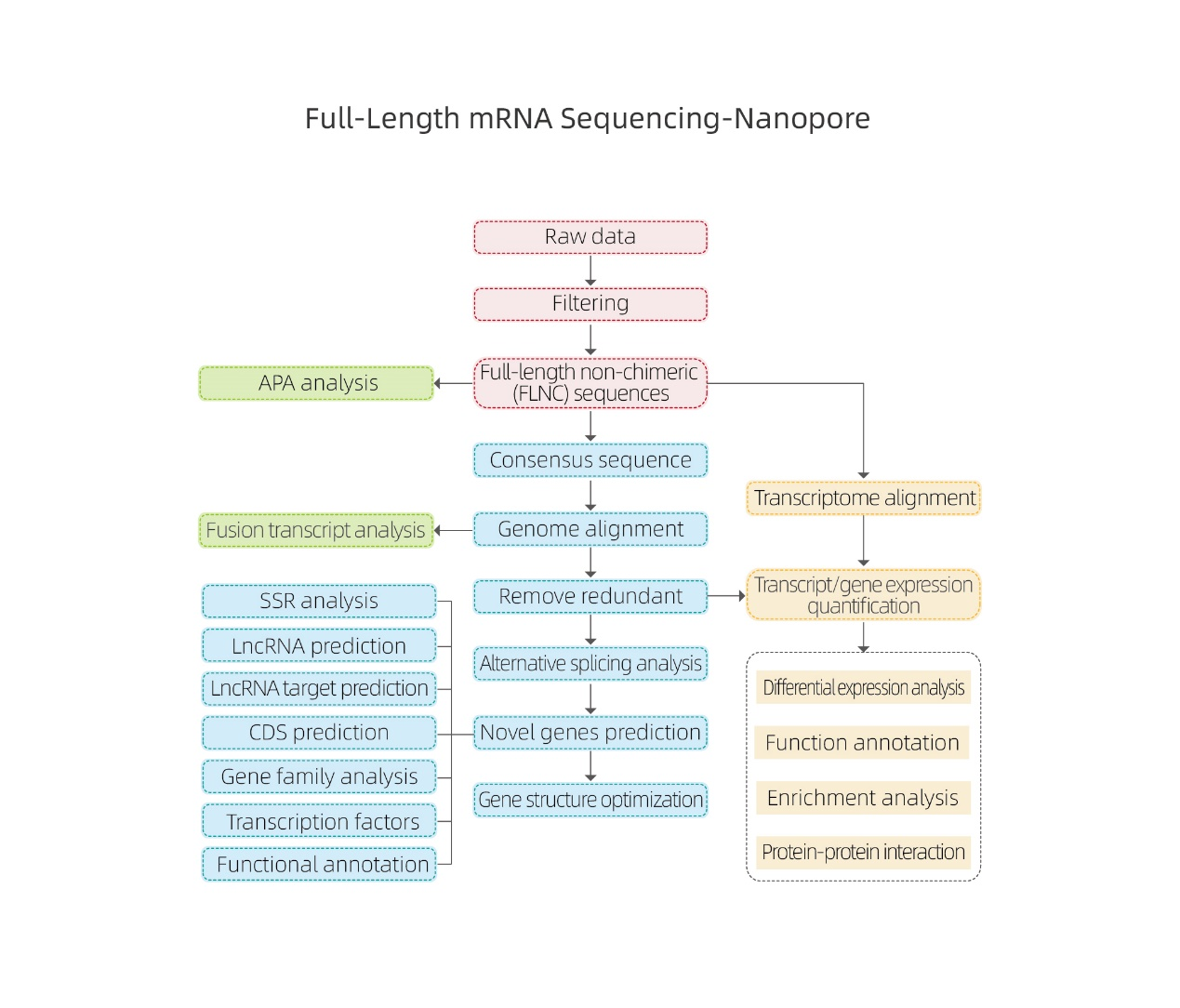
● Zpracování nezpracovaných dat
● Identifikace přepisu
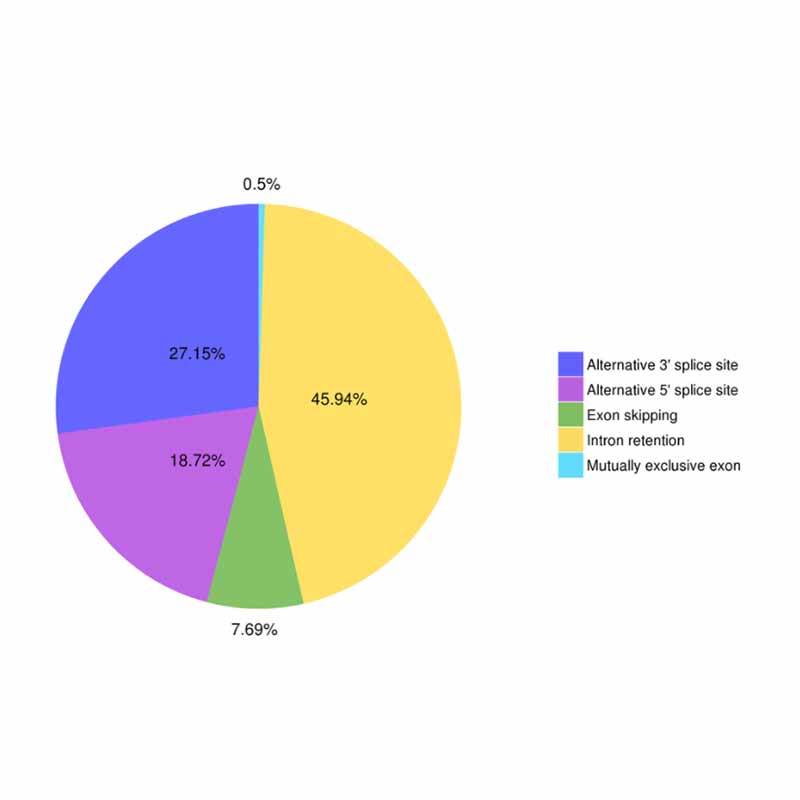
● Alternativní sestřih
● Kvantifikace exprese na úrovni genu a izoformy
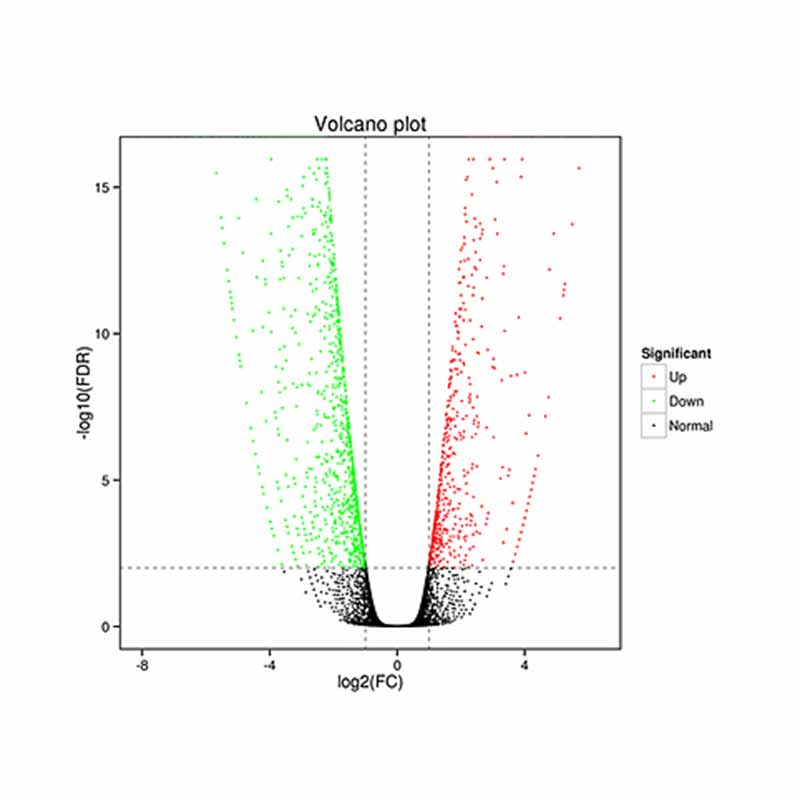
● Analýza diferenciálního výrazu
● Anotace a obohacení funkce (DEGS a Dets)
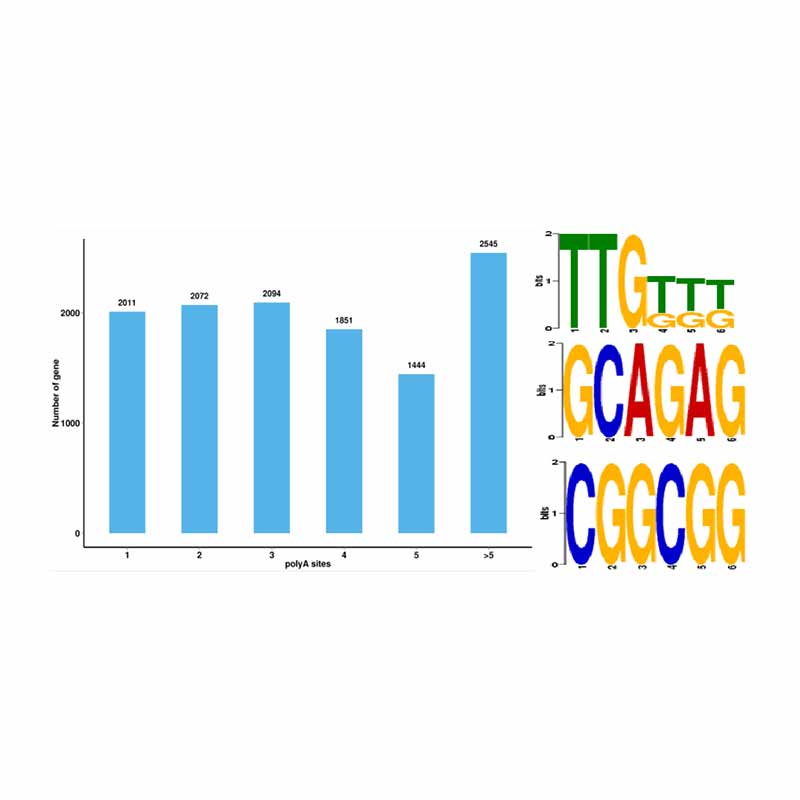
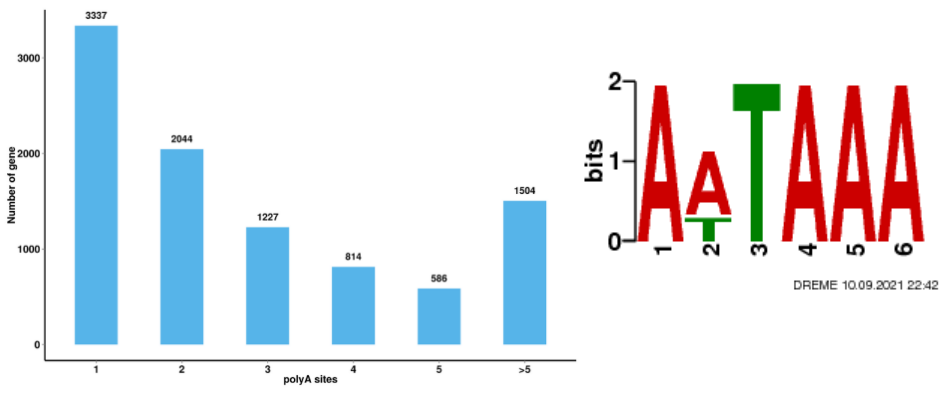
Alternativní analýza sestřihu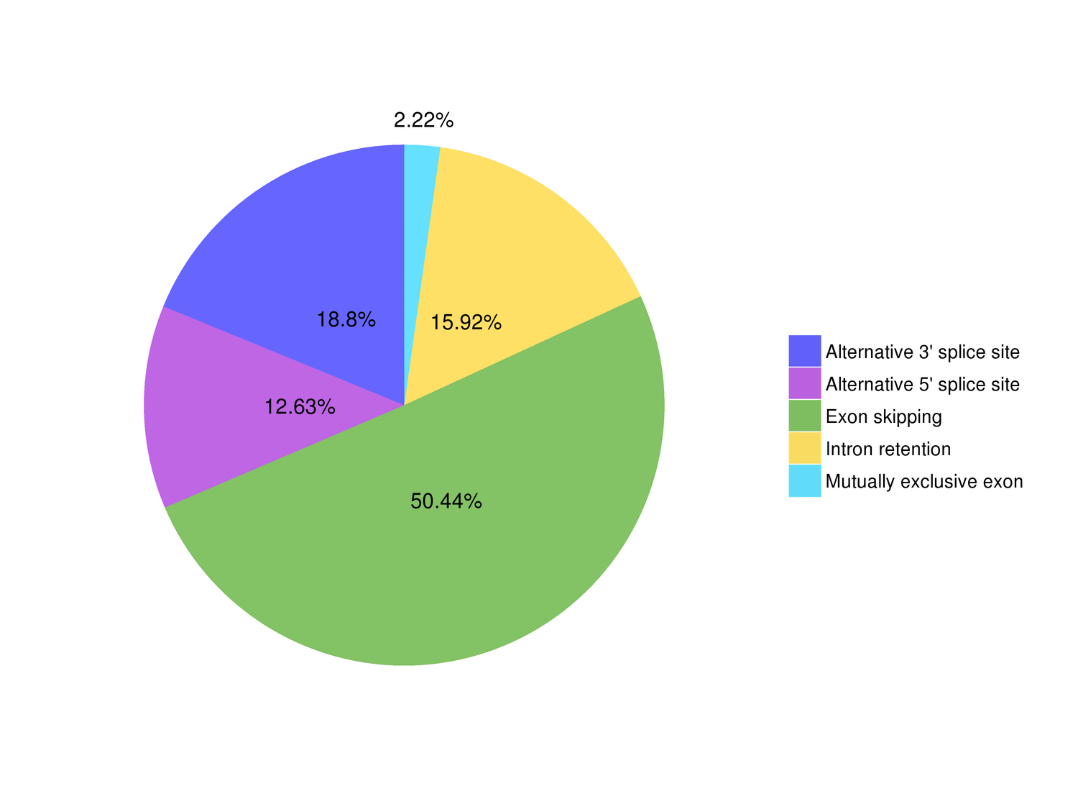 Alternativní analýza polyadenylace (APA)
Alternativní analýza polyadenylace (APA)
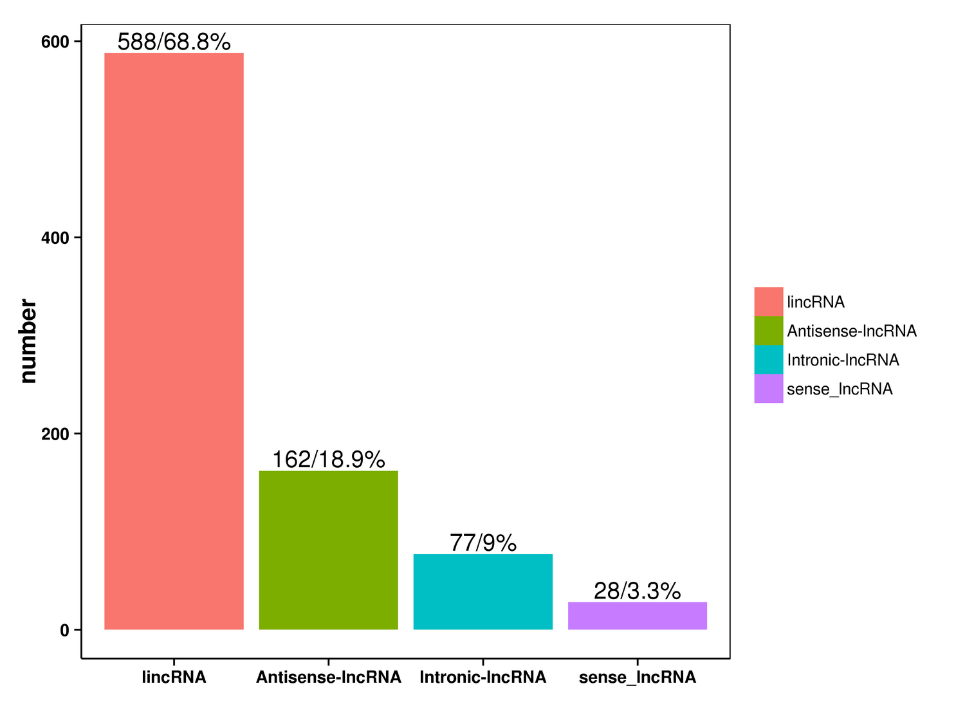
Predikce lncRNA
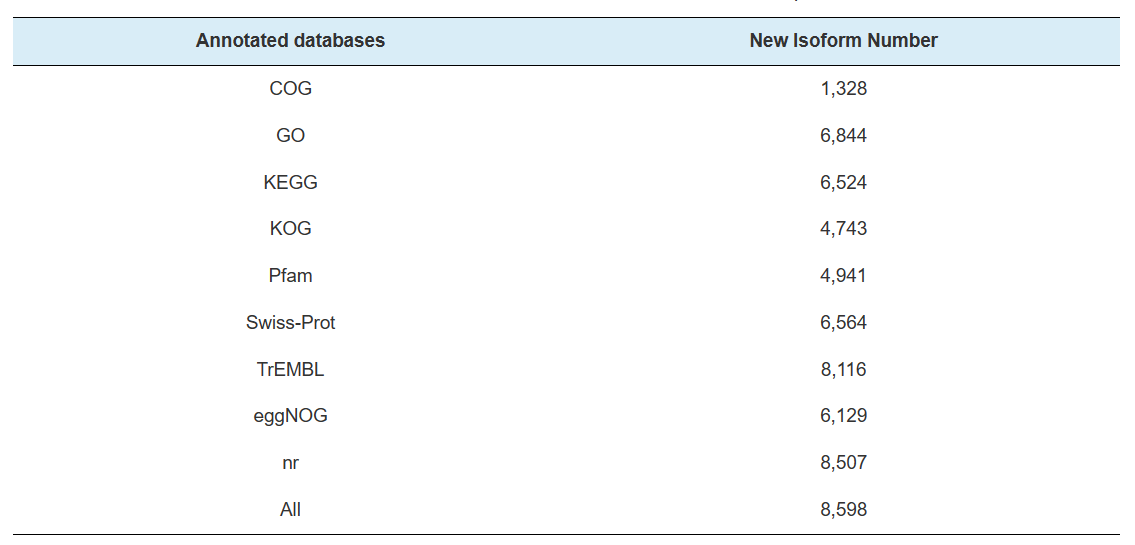
Anotace nových genů
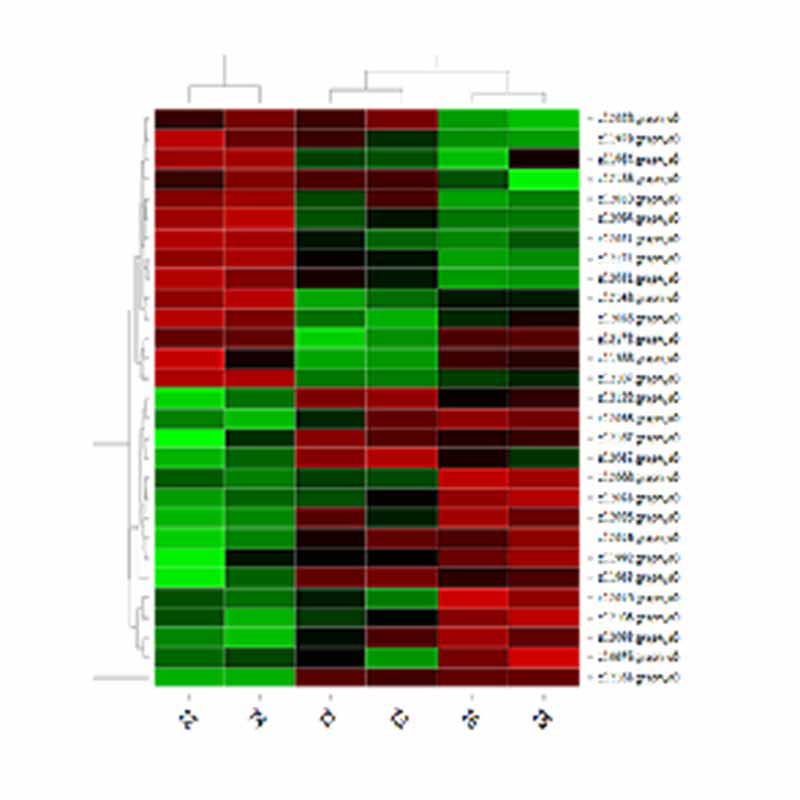
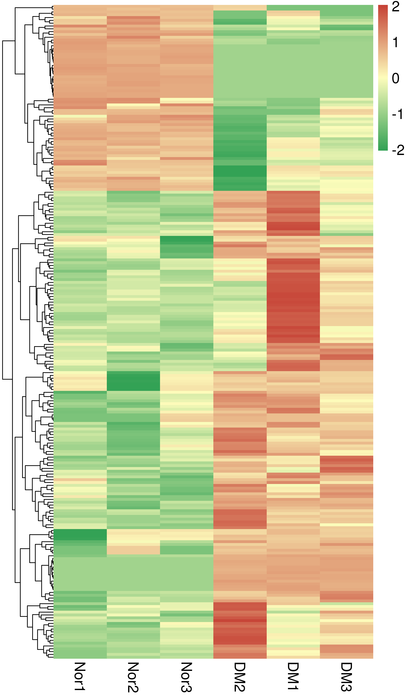
Shlukování dets
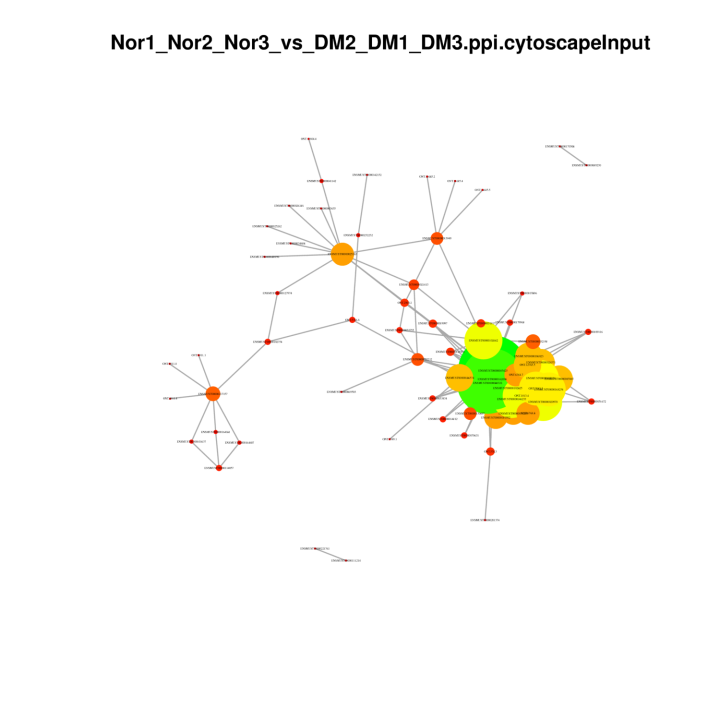
Sítě protein-protein ve stupních
Prozkoumejte pokroky usnadněné službami sekvencování mRNA nanopore BMKGene prostřednictvím kurátorské sbírky publikací.
Gong, B. a kol. (2023) „Epigenetická a transkripční aktivace sekreční kinázy FAM20C jako onkogenu v gliomu“, Journal of Genetics and Genomics, 50 (6), s. 422–433. doi: 10.1016/j.jgg.2023.01.008.
On, Z. et al. (2023) „Transcriptomové sekvenování lymfocytů v plné délce reaguje na IFN-y odhaluje imunitní odpověď skewed Th1 ve Flounderu (Paralichthys Olivaceus)“, Imunologie Fish & Memifish, 134, str. 108636. Doi: 10,016/j.fsi.2023.108636.
Ma, Y. a kol. (2023) „Srovnávací analýza metod sekvenování PACBIO a ONT RNA pro identifikaci Nemopilema Nomurai Venom“, Genomics, 115 (6), str. 110709. Doi: 10.1016/j.ygeno.2023.110709.
Yu, D. et al. (2023) „Analýza NANO-SEQ odhaluje odlišnou funkční tendenci mezi exosomy a mikrovezikuly odvozenými z HumSC“, výzkumu a terapie kmenových buněk, 14 (1), s. 1–13. doi: 10.1186/s13287-023-03491-5/tabulky/6.