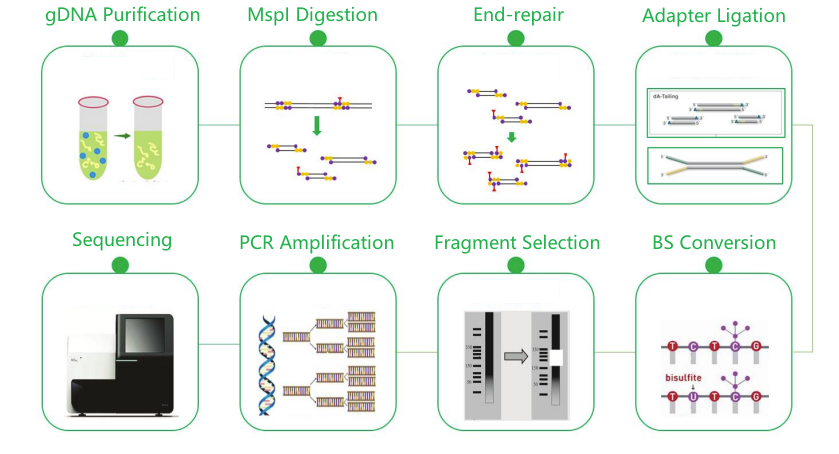
Sequenza di bisulfite à rappresentazione ridotta (RRBS)
Funzioni di serviziu
● Esige un genoma di riferimentu.
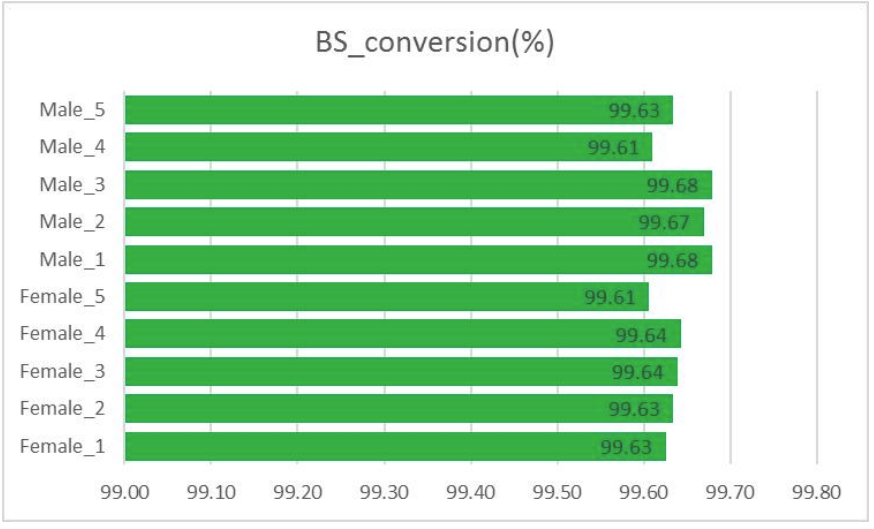
● Lambda DNA hè utilizatu per monitorà l'efficienza di cunversione di bisulfite.
● L'efficienza di a digestione MspI hè ancu monitorata.
● Doppia digestione enzyme per i campioni di e piante.
● Sequenza nantu à Illumina NovaSeq.
Vantaghji di serviziu
●Alternativa à u costu è Efficace à WGBS: permette à l'analisi di esse realizatu à un costu più bassu è cù esigenze di mostra più bassi.
●Piattaforma cumpleta:furnisce un serviziu eccellente unicu da a trasfurmazione di campioni, a custruzzione di a biblioteca è a sequenza à l'analisi bioinformatica.
●Vasta Competenza: cù i prughjetti di sequenza di RRBS cumpletati cù successu in una varietà diversa di spezie, BMKGENE porta più di una dicada di sperienza, una squadra di analisi altamente qualificata, cuntenutu cumpletu è un eccellente supportu post-vendita.
Specificazioni di serviziu
| Biblioteca | Strategia di sequenza | Risultato di dati cunsigliatu | Controlu di qualità |
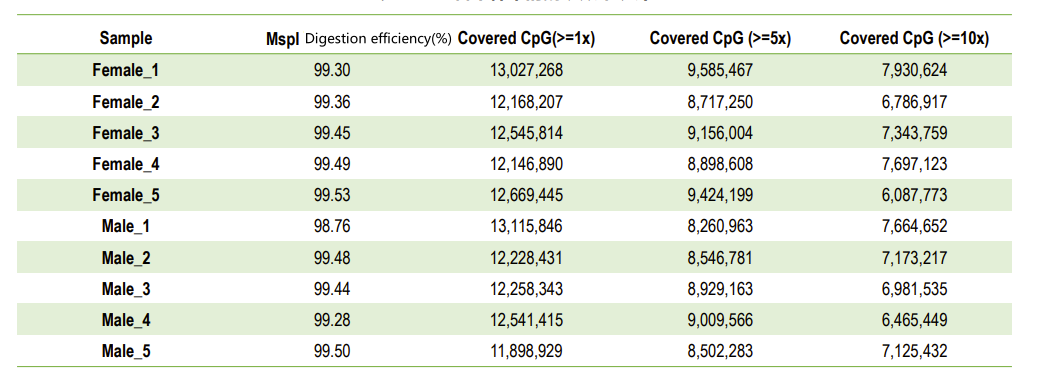
| Biblioteca digerita MspI è trattata bisulfite | Illumina PE150 | 8 Gb | Q30 ≥ 85% cunversione bisulfite> 99% Efficienza di taglio MspI > 95% |
Requisiti di mostra
| Concentrazione (ng/µL) | Quantità totale (µg) |
| |
| DNA genomicu | ≥ 30 | ≥ 1 | Degradazione limitata o contaminazione |
Flussu di travagliu di serviziu

Consegna di mostra

Custruzzione di biblioteca

Sequencing

Analisi di dati
Servizi post-vendita
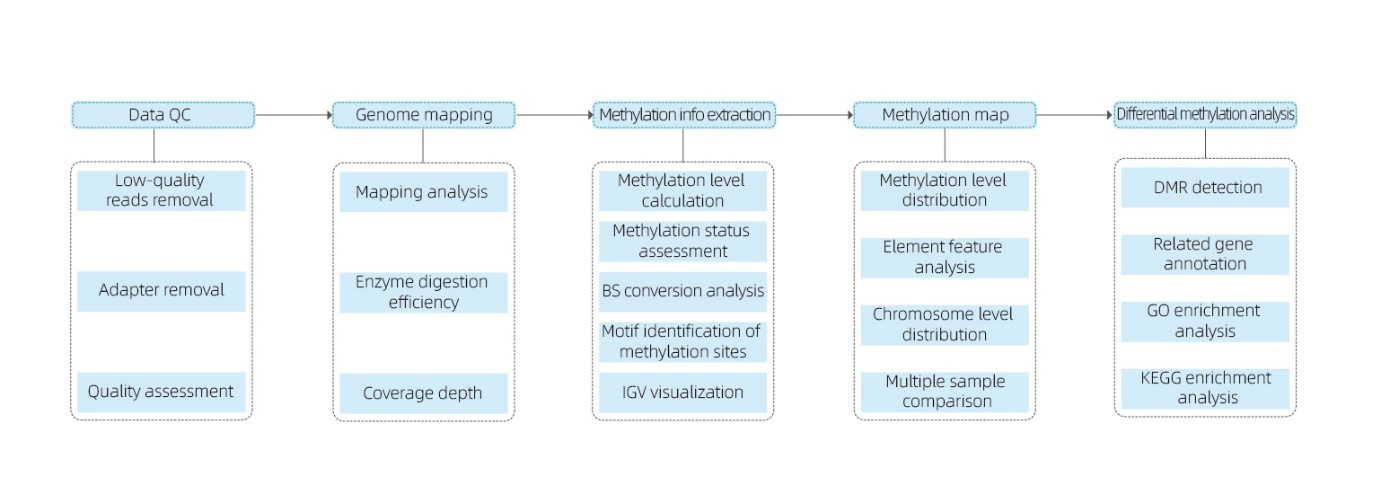
Include l'analisi seguente:
● Control di qualità di sequenza cruda;
● Cartografia à u genoma di riferimentu;
● Detection of 5mC methylated basis è identificazione di motivi;
● Analisi di a distribuzione di metilazione è a comparazione di mostra;
● Analisi di e Regioni Methylated Differentially (DMR);
● Annotazione funzionale di i geni assuciati à i DMR.
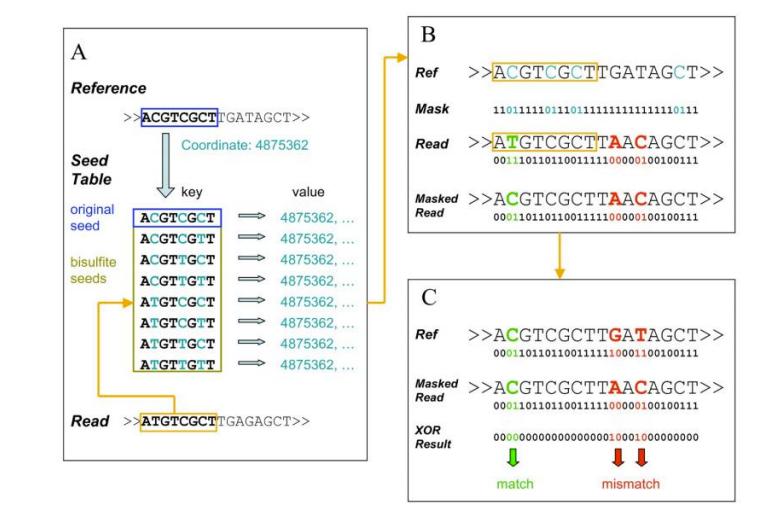
Cuntrolu di qualità: efficienza di a digestione (in a mappa di u genoma)
Controlu di qualità: cunversione di bisulfite (in estrazione di informazioni di metilazione)
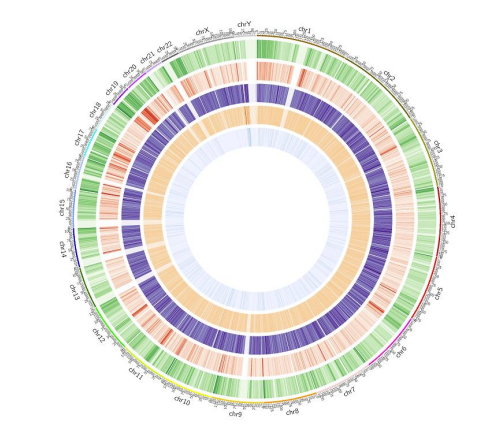
Mappa di metilazione: distribuzione 5mC di metilazione in tuttu u genoma
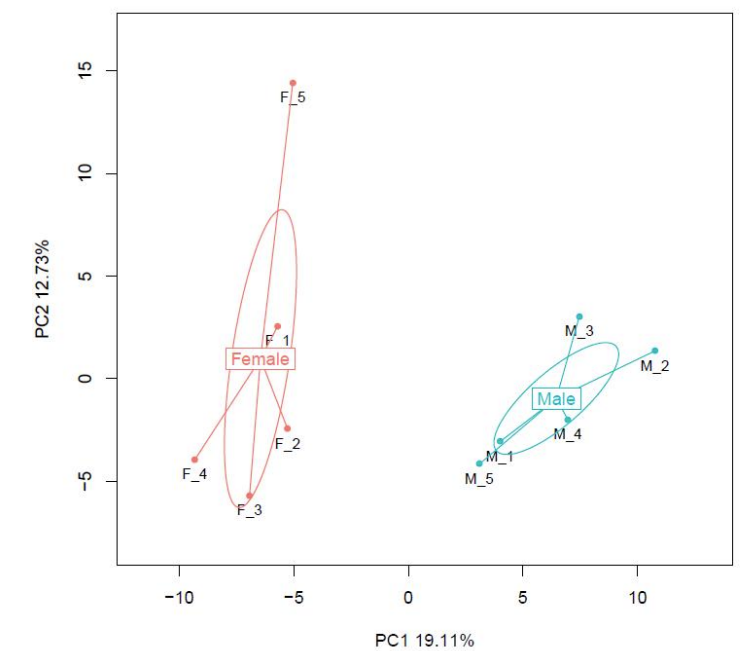
Sample comparison: Analisi di cumpunenti principali
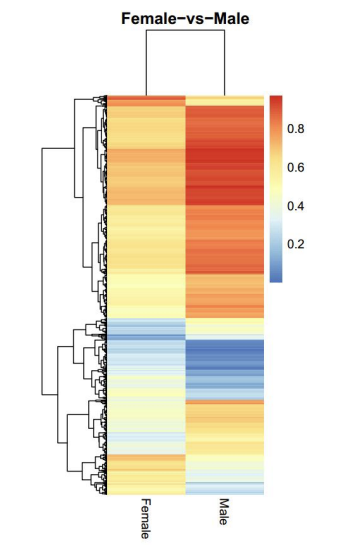
Analisi di Regioni Methylated Differentially (DMRs): heatmap
Esplora l'avanzamenti di ricerca facilitati da i servizii di sequenza di bisulfite di u genoma interu di BMKGene attraversu una cullizzioni curata di publicazioni.
Li, Z. et al. (2022) "Riprogrammazione di alta fedeltà in cellule simili a Leydig per attivazione CRISPR è fattori paracrini",PNAS Nexus, 1 (4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. et al. (2023) "Analisi di metilazione di DNA in tuttu u genoma di a cumpusizioni di u corpu in i gemelli monozigoti cinesi",Revista Europea di Investigazione Clinica, 53(11), p. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) "A metilazione di DNA è u rapportu cintura-anca: un studiu di associazione epigenomica in gemelli monozigoti cinesi",Journal of Endocrinological Investigation, 45 (12), pp. 2365-2376. doi: 10.1007/S40618-022-01878-4.