Analisi setegerante imbottiva
Vantaghji di serviziu

Takagi et al., U Ghjurnale di a Spegna, 2013
● localizazione precisa: mischjà bulche cù 30 + 30 à 200 + 200 individui per minimizà u rumore di fondo; PREDICZIONE NOTIMINATICA DI MUTIMU DI MUTIMU.
● Analisi Comprensiva: In profonda candidata per l'anno di funzione gene, cumprese nr, SwissProt, vai, Kegg, COG, kog, etc.
● Tempu di fornitu più veloce: u rapidu gene localizazione in 45 ghjorni di travagliu.
● Esperienza estensiva: BMK hà cuntribuitu in millaie di tagliati in località, copre diversa cume culturi, prudutti aquatici, fiori, frutti, etc.
Specificazioni di serviziu
Pupulazione:
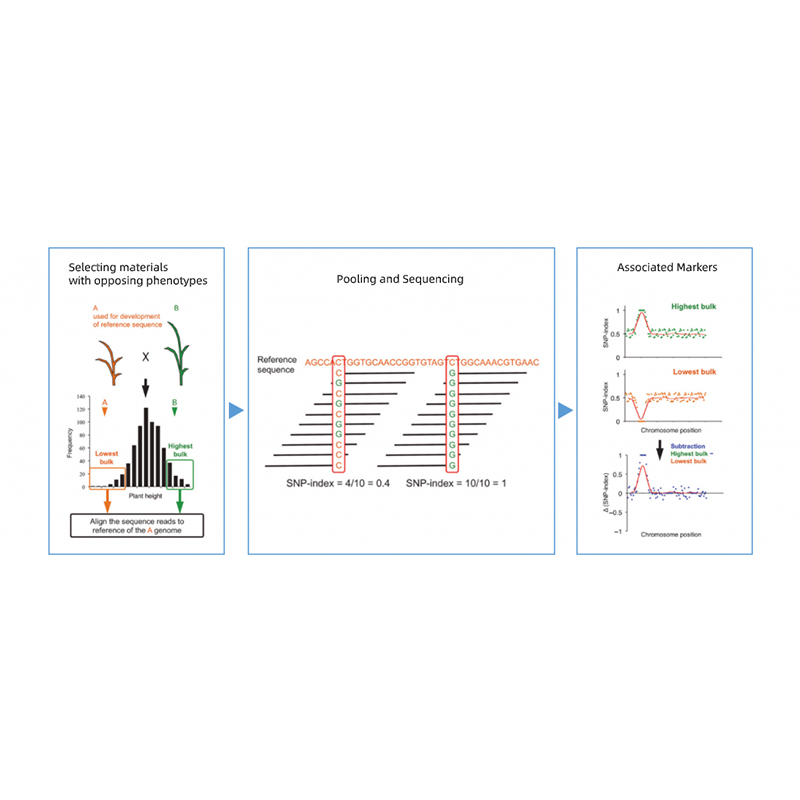
Segregating Progeny di i genitori cù fenotipi opposti.
EG F2 PROGENY, BACKCROSSTING (BC), Linea Inbred Rincominante (Ril)
Piscina mixing
Per i tratti Qualificativi: 30 à 50 individui (minimu 20) / ingrossu
Per TRatis quantitative: Top 5% à 10% individui cù fenotipes estremi in tutta a pupulazione (minimu 30 + 30).
Prufundità di sequenza cunsigliata
Almenu 20x / Parent è 1x è di a prole di u mischju di u mischju di 30 + 30 individuali, a prufundità di sequenza serà 30x per ingrossa)
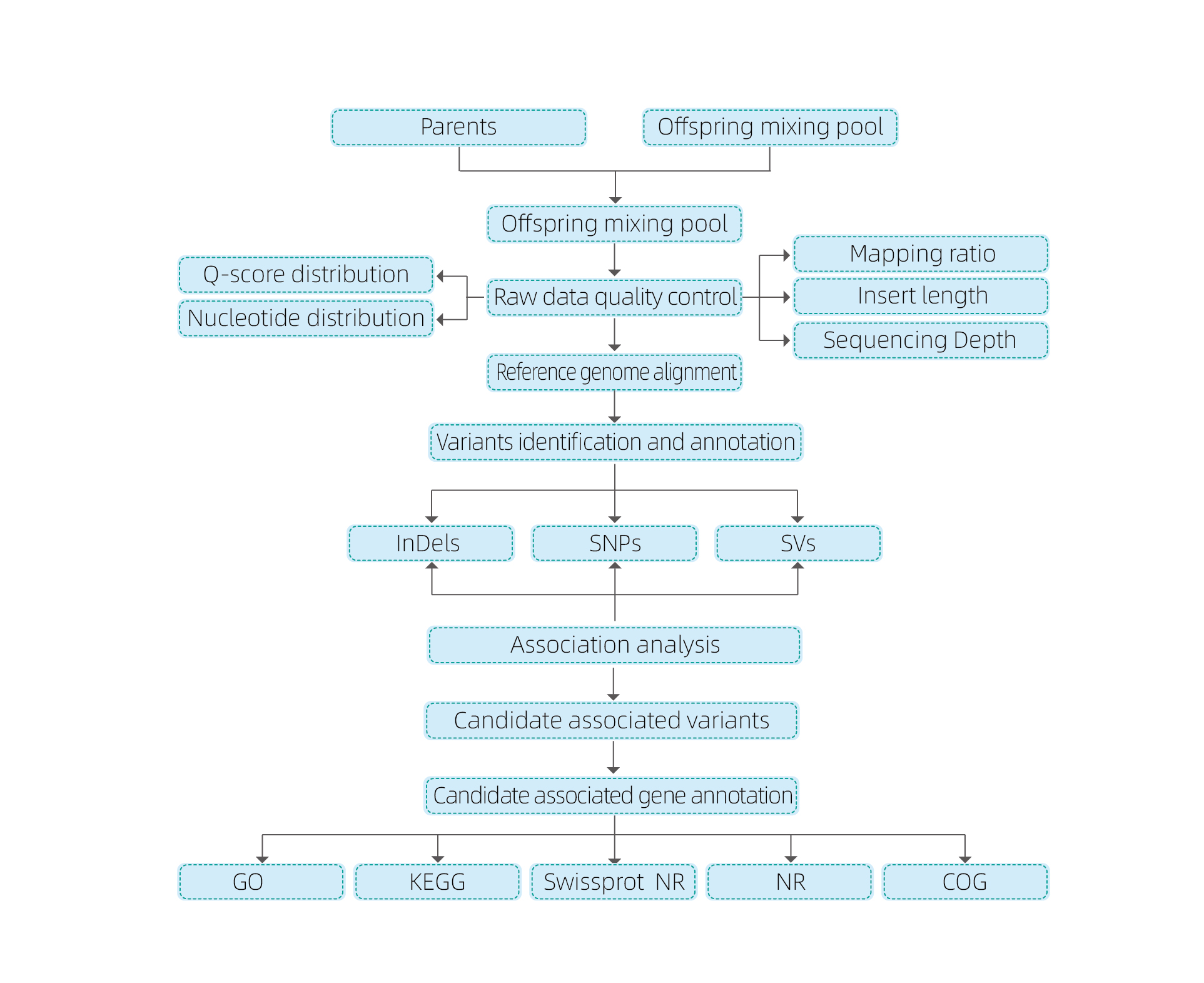
Analisi bioinformatica
● u Genoma di Genomi
● Processazione di dati
● SNP / Induve chjama
● Screening Regione Candidata
● Funzione di a funzione Gene à Candidate

Esigenze è consegna
Requisiti di mostra:
Nucleotidi:
| Sample GDNA | Mostra di tessulu |
| CONCENTRAZIONE: ≥30 ng / μl | Piante: 1-2 g |
| Quantità: ≥2 μg (volunno ≥15 μl) | Animali: 0,5-1 G |
| Purità: Od260 / 280 = 1.6-2,5 | Sangue sanu: 1,5 ml |
U flussu di travagliu di serviziu

Design di sperimentariu

Spedizione di mostra

Extrazzione rna

Custruzione di Biblioteca

SEQUENZZA

Analisi di dati

Servizi dopu in vendita
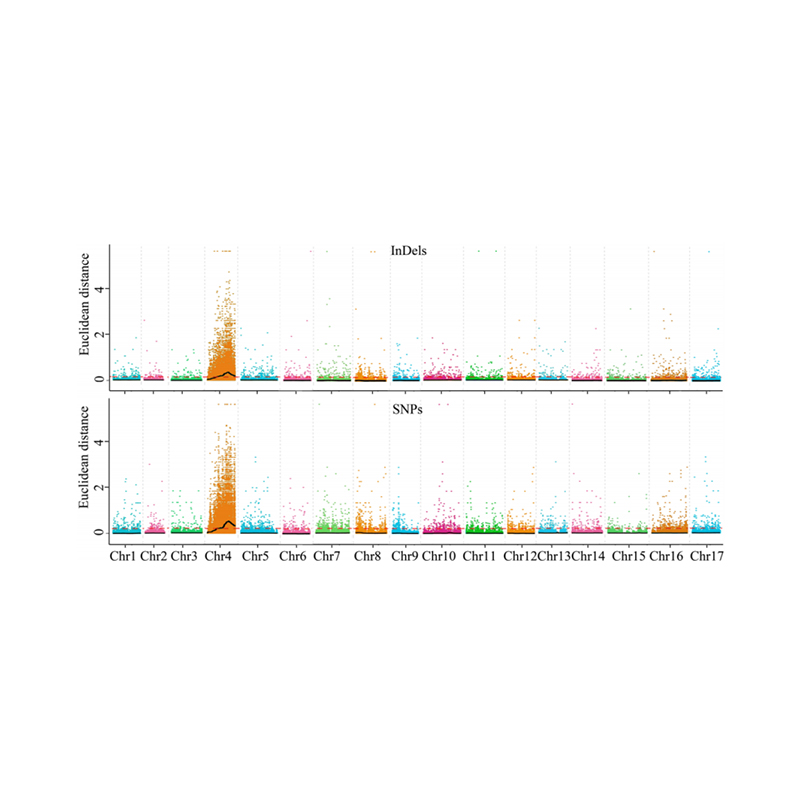
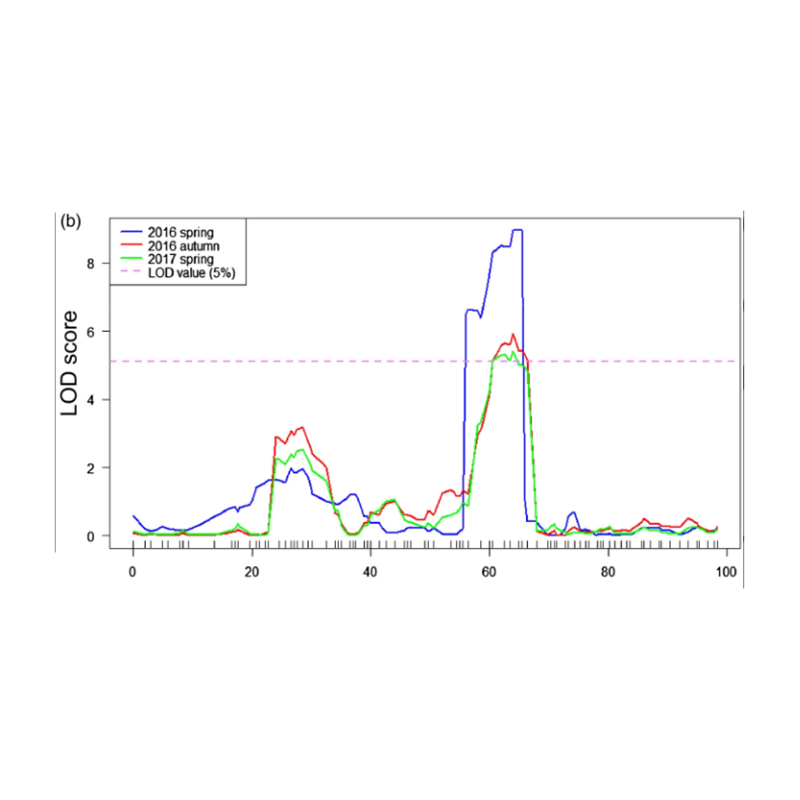
1. Basioni analisi di 1. Se follà a distanza euclidiana (ED) per identificà a regione di u candidatu. In a seguente figura
X-AXIS: Numeru di cromosoma; Ogni puntu rapprisenta un valore ed di un snp. A linea negra currisponde à u valore di l'addombu. Un valore più altu indica una associazione più significativa trà u situ è u fenotipu. A Linea di Dash Rossa rapprisenta a soglia di l'associu significativu.

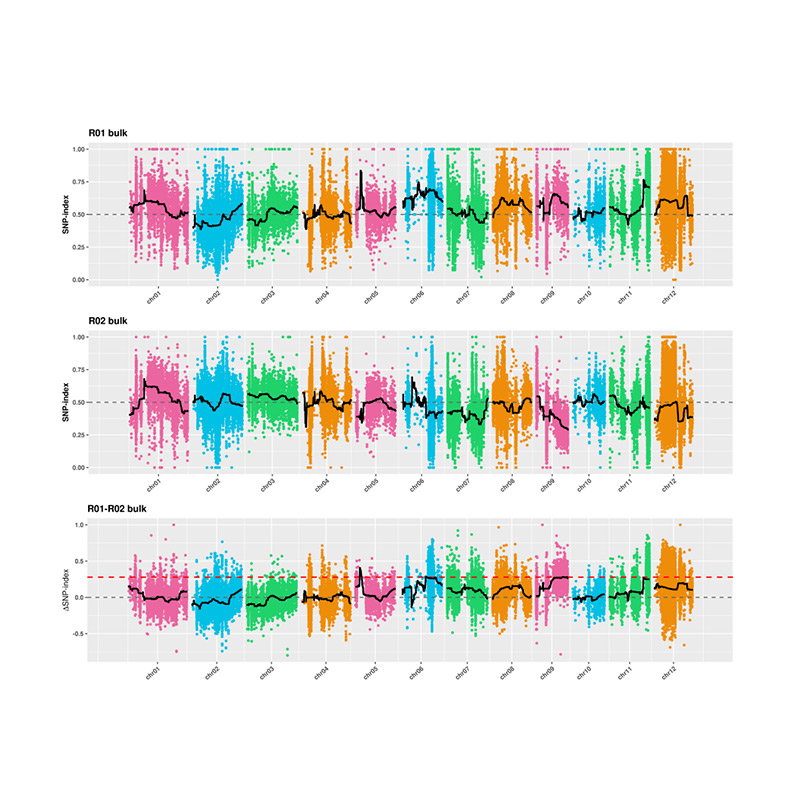
L'analisi di 2,avantazioni basatu senza un indice snp
X-AXIS: Numeru di cromosoma; Ogni puntu rapprisenta u valore di l'indice snp. A linea negra ferma per u valore di l'indice snp adattatu. U valore più grande hè, u più significativu l'associu hè.

Casu BMK
U Major-EXC..0 FNL7.1 codificate una proteina abbundante abbundante assuciata cù a lunghezza di u collu di frutti in Cucumber
Publicatu: Plant Neachottnology Journal, 2020
Strategia Sequencing:
Genitori (jin5-508, Yn): Generale di Genome di u geniu di 34 × è 20 ×.
DNA Pooles (50 - Anziatu è 50 cuncricutu è 50 più pocu di dicurità): Scancimentu di 61 × è 52 ×
Risultati chjave chjave
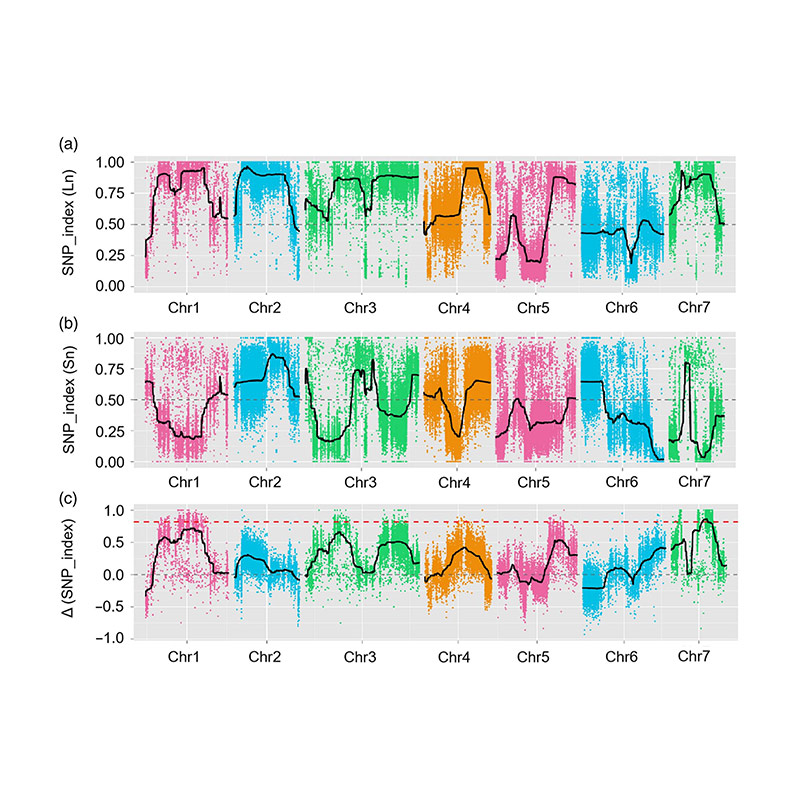
In stu studiu, a pupulazione segregata (F2 è F2: 3) hè stata generata da attraversà u paturu di u pulizzeru di u paturu à longu u collu Dui piscine DNA sò stati custruiti da 50 individui di longa estrema longa è 50 estremi di u collu Mai-Effettu QTL hè statu identificatu nantu à u chr07 da l'analisi di BSA è a cartografia di QTL tradiziunale. A regione di u Candidata era più strettu down in messe-mazona di gene espressivi è esperimenti transressivi, chì revuenu a genua chjave in cuntrullandu u collu, csfnl7.1. Inoltre, polimorfisimu in csfnl7.1 Regione Promotter hè stata trovata per esse assuciata cun espressione currispondente. L'analisi fisicinetici più suggerì chì Fnl7.1 Locanu hè assai prubabile di esse originata da l'India.
 Qtl-mapping in l'analisi di BSA per identificà a regione di u candidatu assuciatu à a lunghezza di u collu |  LOGFT PROFILLI DI U CUCCU U BLACK-DENNU SKLY Identificatu nantu à u CHR07 |
Xu, X., et al. "L'effettu tutale tutale fnitativu fnitativu fnl7.1 Codificate l'embrugeni di a Proteina abbundante assuciata cù a lunghezza di u collu di fruttu in Cucumber." Plant journovia di biotechnologia 18,7 (2020).