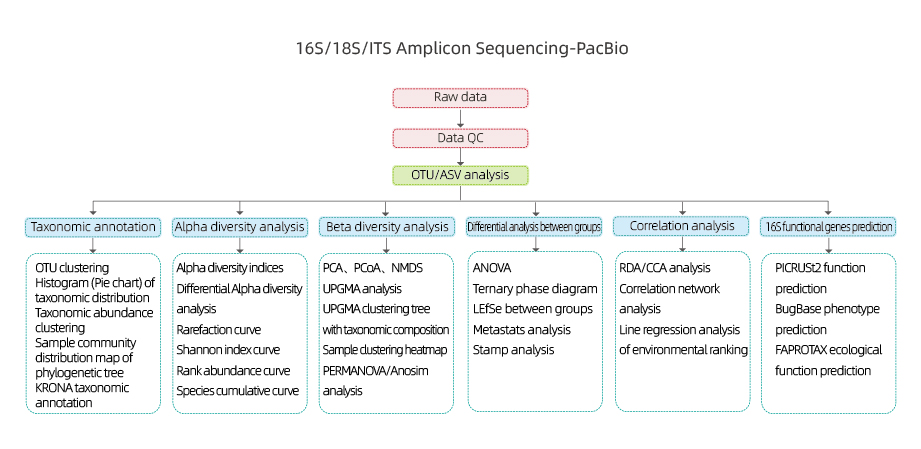
16S/18S/ITS Amplicon Sequencing-PacBio
Funzioni di serviziu
● Piattaforma di sequenza: PacBio Revio
● Modu di sequenza: CCS (lecture HiFi)
● Amplificazione di a regione di destinazione seguita da a cunnessione in tandem di ampliconi prima di a preparazione di a biblioteca di campana HiFi SMRT
Vantaghji di serviziu
●Risoluzione tassonomica più alta: Than sequenza di ampliconu cortu,chì permette tassi di classificazione OTU più altu à u livellu di spezie.
●Chjama di basa assai precisa: Sequenza di u modu PacBio CCS (lecture HiFi).
●Senza isolamentu: Identificazione rapida di a cumpusizioni microbica in campioni ambientali.
●Ampiamente applicabile: Diversi studii di a cumunità microbiana.
●Analisi bioinformatica cumpleta: L'ultimu pacchettu QIIME2 (intruduzioni quantitative in l'ecologia microbica) cù diverse analisi in termini di basa di dati, annotazione, OTU / ASV.
●Vasta Competenza: Cù millaie di prughjetti di sequenza di ampliconi realizati annu, BMKGENE porta più di una dicada di sperienza, una squadra di analisi altamente qualificata, cuntenutu cumpletu, è un eccellente supportu post-vendita.
Specificazioni di serviziu
| Biblioteca | Strategia di sequenza | Dati cunsigliatu |
| Amplicon | PacBio Revio | 10/30/50 K tags (CCS) |
Requisiti di mostra
| Concentrazione (ng/µL) | Quantità totale (µg) | Volume (µL) |
| ≥5 | ≥0,3 | ≥20 |
Cunsigliu Cunsigliu di Campione
Freeze i campioni in nitrogenu liquidu per l'ora di 3-4 è almacenà in nitrogenu liquidu o -80 gradi à riservazione longu. U trasportu di mostra cù ghiaccio seccu hè necessariu.
Flussu di travagliu di serviziu

Consegna di mostra

Custruzzione di biblioteca

Sequencing

Analisi di dati

Servizi post-vendita
Include l'analisi seguente:
●Raw cuntrollu di qualità di dati
●OTU clustering/De-noise (ASV)
● annotazione OTU
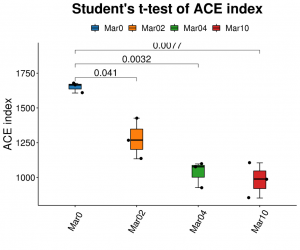
●Alfa analisi diversità: indici multipli, cumpresi Shannon, Simpson è ACE.
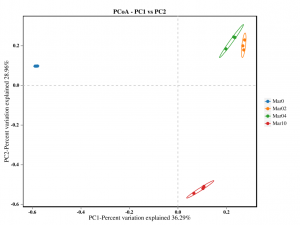
●Beta analisi diversità
●Analisi inter-gruppu
●Correlation analisi: trà fattori ambiente è cumpusizioni OUT è diversità
●16S predizione geni funziunali
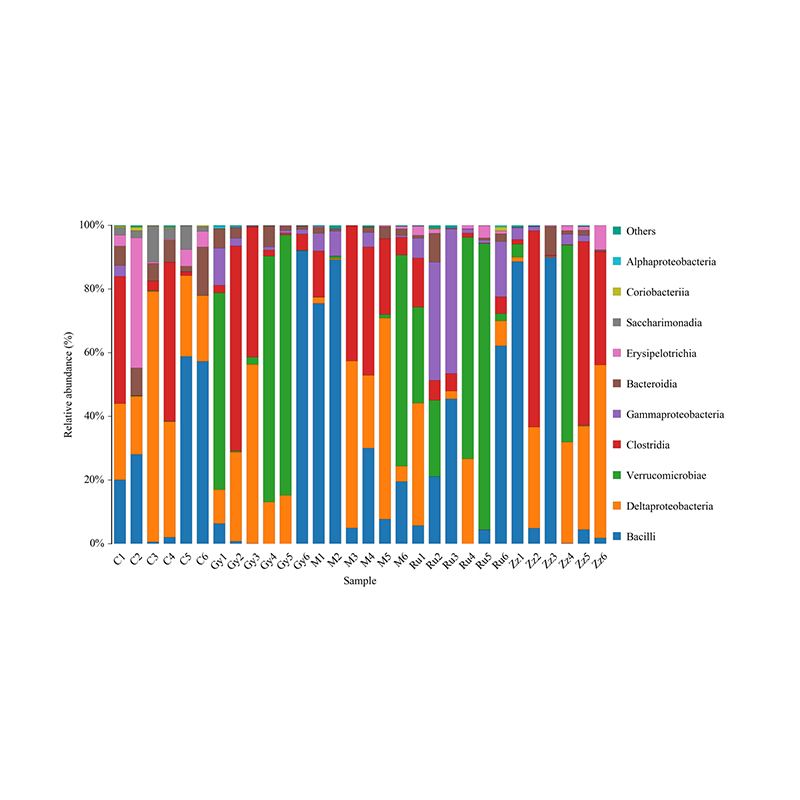
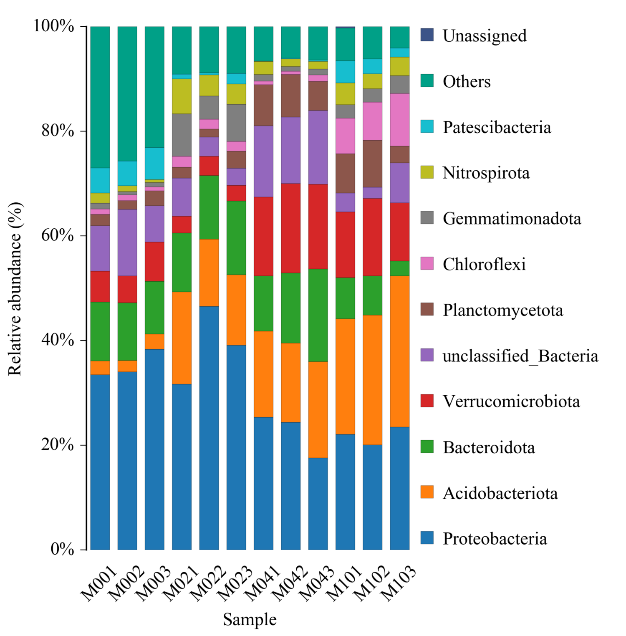
Istogramma di distribuzione tassonomica
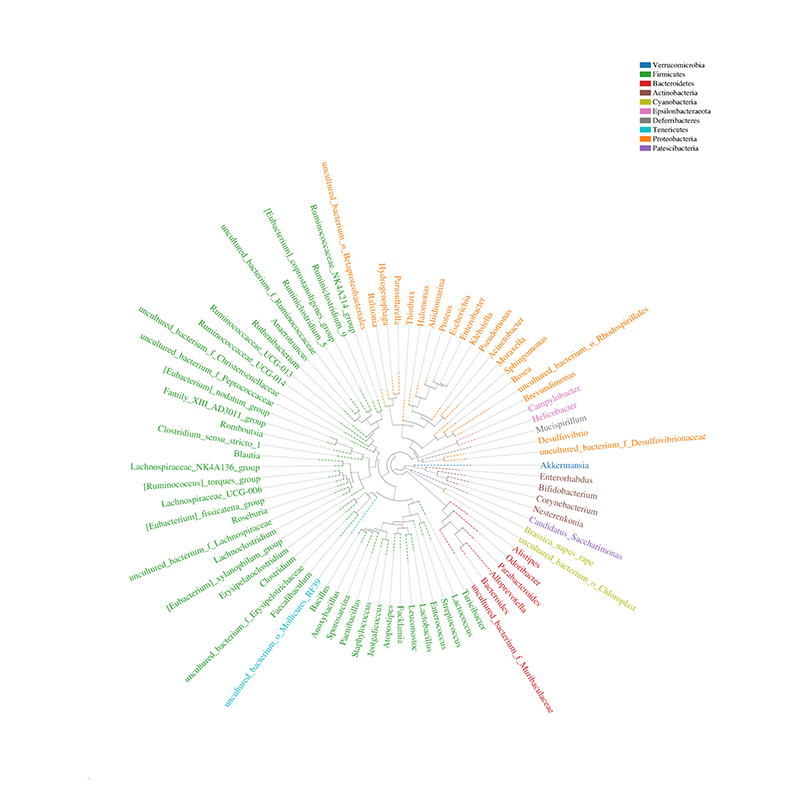
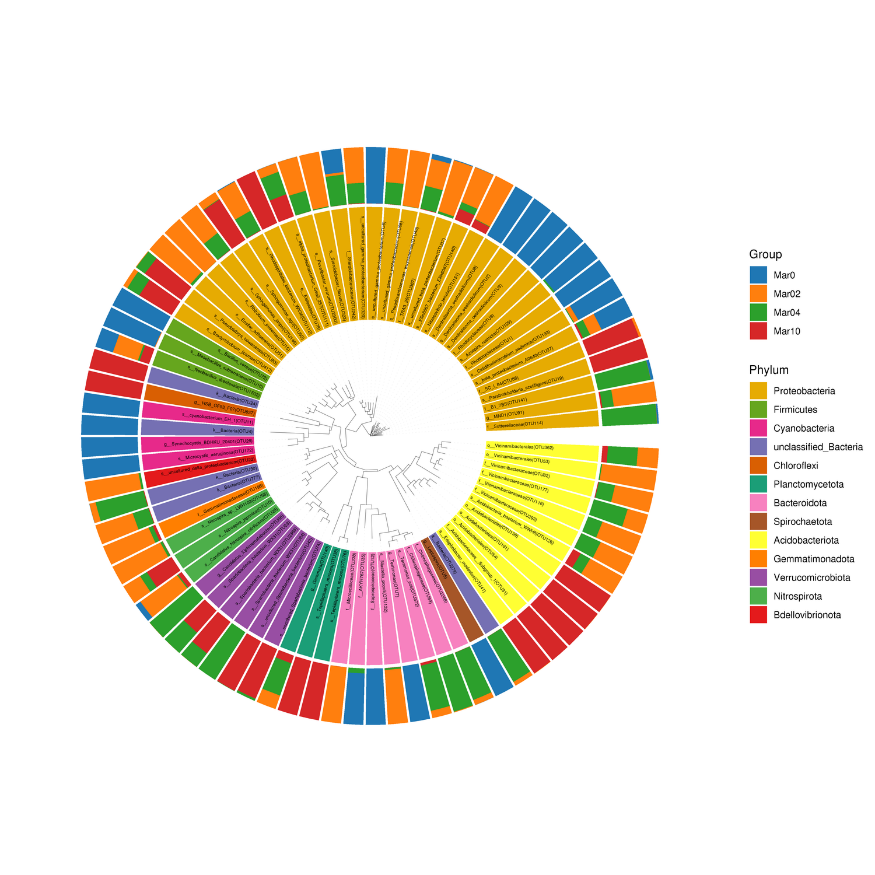
Arbulu filogeneticu di distribuzione di a cumunità
Analisi di a diversità alfa: ACE
Analisi di a diversità Beta: PCoA
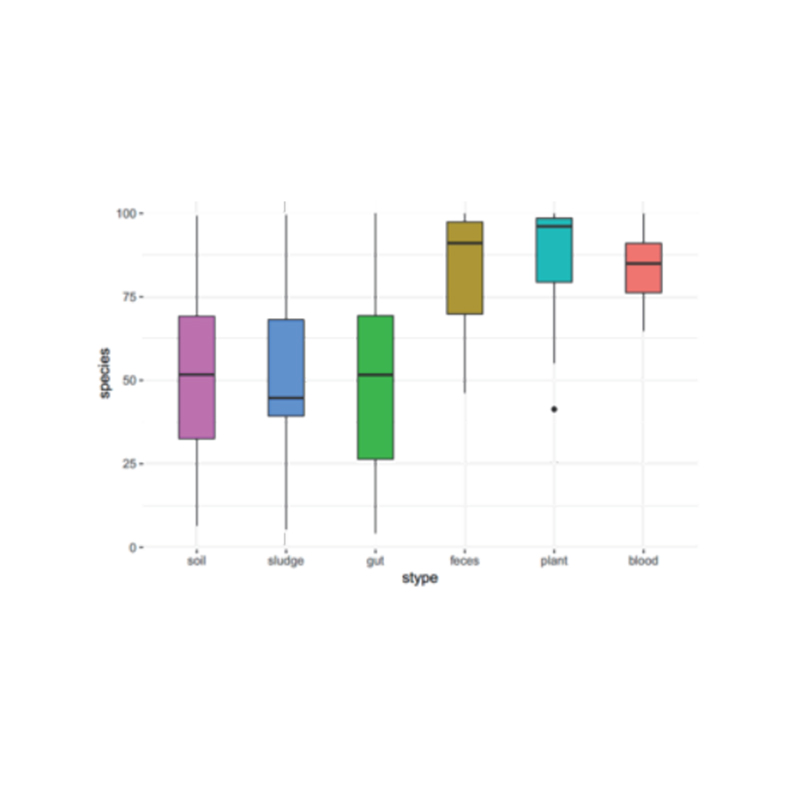
Analisi intergruppu: ANOVA
Esplora l'avanzamenti facilitati da i servizii di sequenza di amplicone di BMKGene cù PacBio attraversu una cullizzioni curata di publicazioni.
Gao, X. è Wang, H. (2023) 'Analisi Comparativa di Profili Bacteriali Rumen è Funzioni durante l'Adaptazione à Diversi Fenologia (Regreen vs. Grassy) in Pecore Merino Alpine cù Dui Stadi di Crescita nantu à un Pratu Alpino', Fermentazione, 9( 1), p. 16. doi : 10.3390/FERMENTATION9010016/S1.
Li, S. et al. (2023) "Capturing the microbial dark matter in desert soils using culturomics-based metagenomics and high-resolution analysis", npj Biofilms and Microbiomes 2023 9: 1, 9 (1), pp. 1-14. doi: 10.1038/s41522-023-00439-8.
Mu, L. et al. (2022) "Effetti di i sali di l'acidu grassu nantu à e caratteristiche di fermentazione, a diversità bacteriana è a stabilità aerobica di l'insilatu mistu preparatu cù alfalfa, paglia di risu è crusca di granu", Journal of the Science of Food and Agriculture, 102 (4), pp. 1475– 1487. doi: 10.1002/JSFA.11482.
Yang, J. et al. (2023) "L'Interazzione trà i Biomarcatori di Stress Oxidative è Gut Microbiota in l'Effetti Antioxidanti di Estratti da Sonchus brachyotus DC. in Oxazolone-Induced Intestinal Oxidative Stress in Adult Zebrafish', Antioxidants 2023, Vol. 12, Pagina 192, 12(1), p. 192. doi : 10.3390/ANTIOX12010192.