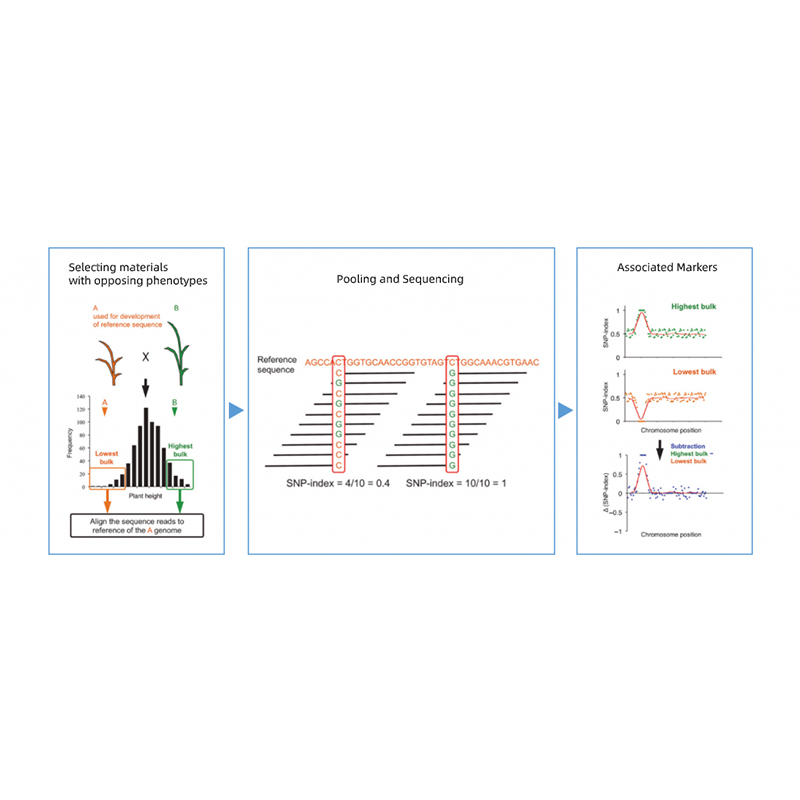
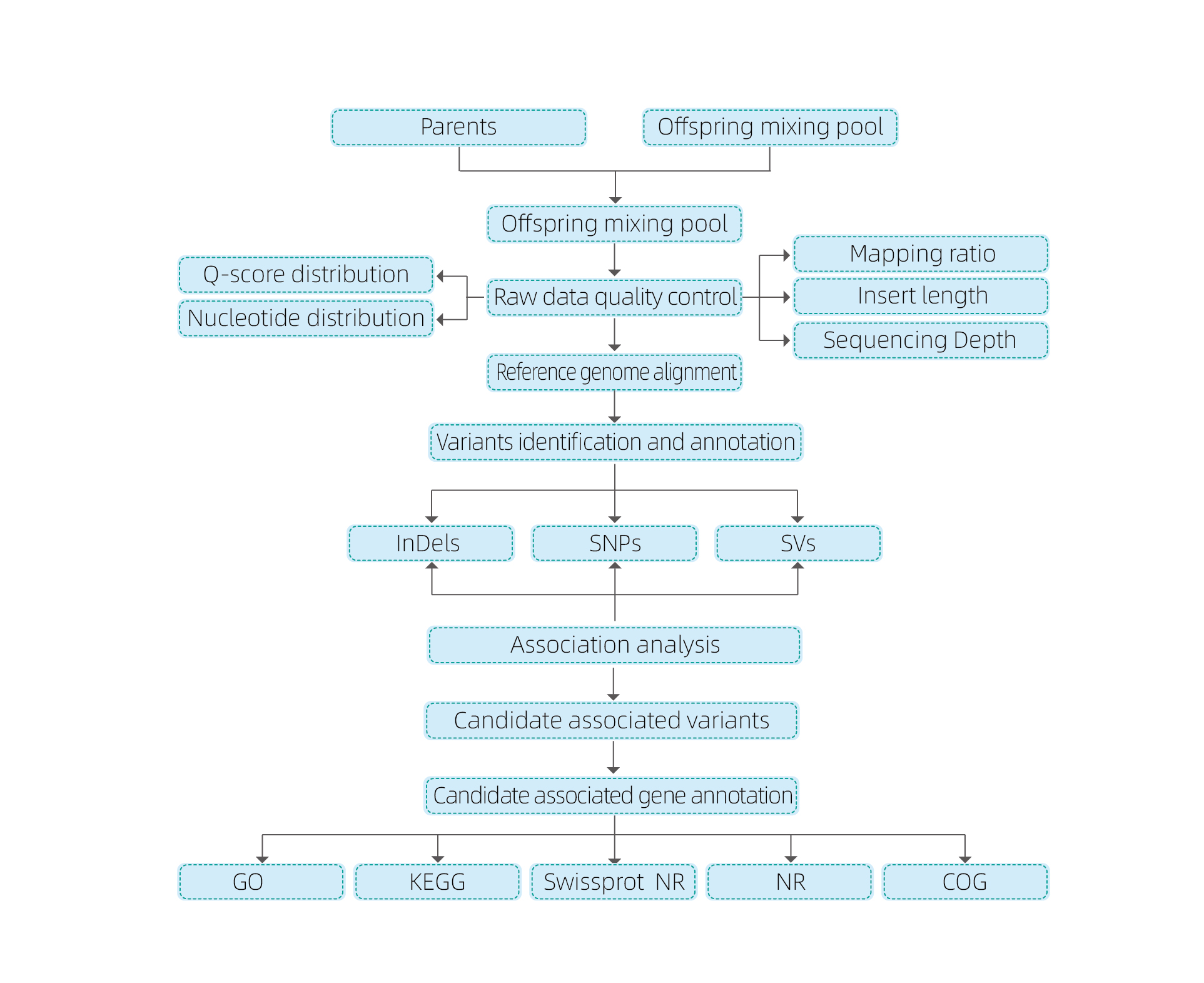
Gisalibay ang pag-analisar sa Segregant
Mga Buluhaton sa Serbisyo

Takagi et al., Ang journal sa tanum, 2013
● Tukma nga lokalisasyon: Pagsagol sa mga Bulk sa 30 + 30 hangtod 200 + 200 nga mga indibidwal aron maminusan ang kasaba sa background; Dili magkasapid nga panagna nga adunay kandidikto nga nakabase sa kandidato.
● Comprehensive analysis: In-depth candidate gene function annotation, including NR, SwissProt, GO, KEGG, COG, KOG ,etc.
● Mas paspas nga oras sa pag-agi: Dali nga localization sa gene sa sulod sa 45 nga mga adlaw sa pagtrabaho.
● Daghang kasinatian: Ang BMK nakaamot sa libu-libong mga kinaiya nga gi-localize, nga naglangkob sa lainlaing mga espisye sama sa mga tanum, mga bulak, bulak, mga bunga, ug uban pa, ug uban pa, ug uban pa
Mga Estudyante sa Serbisyo
Populasyon:
Pag-seguck sa Progeny sa mga Ginikanan nga Adunay Pagsupak sa Phenotypes.
eg F2 Progeny, backcrossing (BC), recombinant inbred Line (Ril)
Pagsagol sa pool
Alang sa mga kwalipikasyon nga mga kinaiya: 30 hangtod 50 nga mga indibidwal (minimum 20) / Daghang
Alang sa Quantitative Tratis: Top 5% hangtod sa 10% nga mga indibidwal nga adunay grabe nga mga phenotypes sa tibuuk nga populasyon (minimum 30 + 30).
Girekomenda nga giladmon
Labing menos 20x / Ginikanan ug 1x / Offspring Indibidwal (pananglitan alang sa mga anak nga nagsagol sa pool nga 30 + 30 nga indibidwal, ang giladmon sa 30x matagbaw)
Mga Bioinformatics Awalizes
● Ang tibuuk nga pag-okupar sa genome
● Pagproseso sa datos
● Snp / Indel calling
● Pag-screening sa Kandidato sa Rehiyon
● Pag-andar sa Kendigo

Mga kinahanglanon nga sample ug paghatud
Mga Kinahanglan nga Sampol:
Nucleotides:
| Sample sa GDNA | Sample sample |
| Konsentrasyon: ≥30 ng / μl | Mga Tanum: 1-2 g |
| Kadaghanan: ≥2 μg (Volumn ≥15 μl) | Mga Hayop: 0.5-1 g |
| Kaputli: OD260 / 280 = 1.6-2.5 | Tibuok nga Dugo: 1.5 ML |
Pag-agos sa Pag-alagad

Disenyo sa Pag-eksperimento

Sample nga Paghatud

Pagkuha sa rna

Pagtukod sa Library

Sunud-sunod

Data Analysis

Pagkahuman sa mga serbisyo nga gibaligya
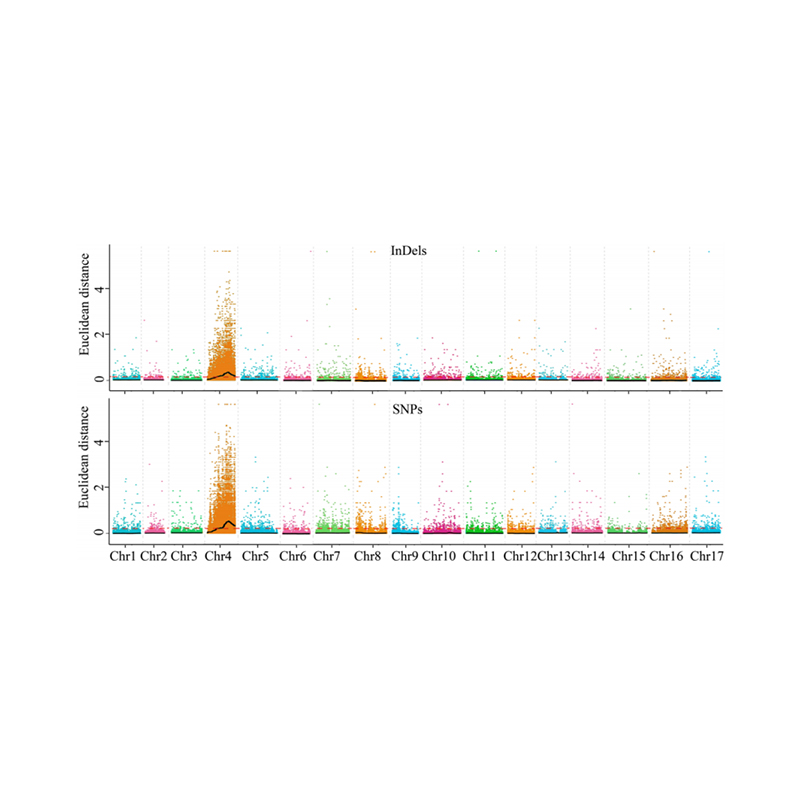
Ang base sa pag-analisar sa 1.Astociation sa Euclidean Distansya (ED) aron mahibal-an ang OPERICATION REGION. Sa mosunud nga numero
X-Axis: Cromosome nga numero; Ang matag tuldok nagrepresentar sa usa ka kantidad nga adunay usa ka SSP. Ang itom nga linya katumbas sa gitaod nga kantidad nga na-fort. Ang usa ka labing taas nga kantidad nga Ed nagpaila sa usa ka labi ka hinungdanon nga panag-uban tali sa site ug sa phenotype. Ang linya sa pula nga dash nagrepresentar sa threshold nga hinungdan sa panag-uban.

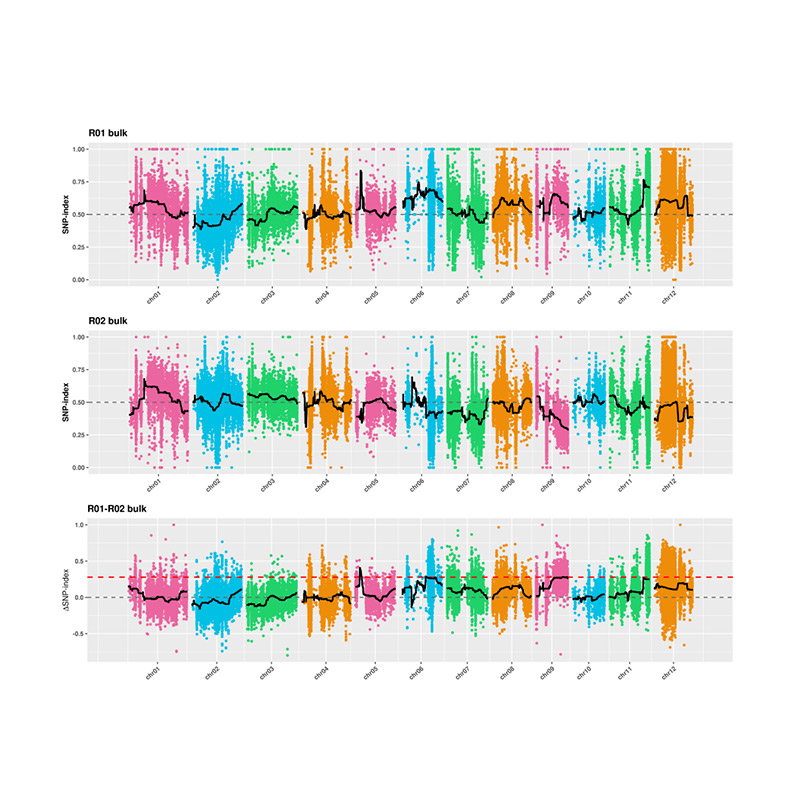
Ang pag-analisar sa 2.Asaciation nga wala'y snp-index
X-Axis: Cromosome nga numero; Ang matag tuldok nagrepresentar sa kantidad sa SNP-Index. Ang itom nga linya nagbarug alang sa angay nga kantidad sa SNP-Index. Ang labi ka dako nga kantidad mao, ang labi ka hinungdanon nga asosasyon mao ang.

Kaso sa bmk
Ang panguna nga epekto nga taas nga trait lolus fnl7.1 nag-encode sa usa ka ulahing bahin sa embryagenesis nga daghang protina nga may kalabutan sa pipino sa prutas
Gipatik: Itanom biotechnology journal, 2020
SEQUENCING SCHATKY:
Mga ginikanan (Jin5-508, Yn): Ang tibuuk nga pag-okupar sa genome alang sa 34 × ug 20 ×.
DNA POOLS (50 Long-NEDED UG 50 SLECK-NEDED): Pag-resekar sa 61 × ug 52 ×
Key Resulta
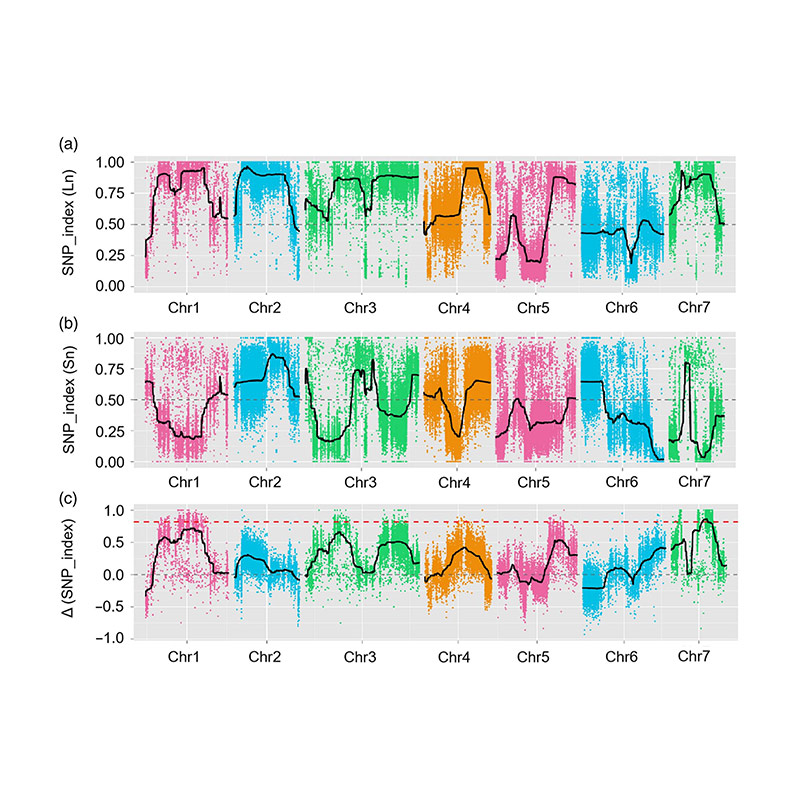
In this study, segregating population(F2 and F2:3) was generated by crossing long-neck cucumber line Jin5-508 and short-neck YN. Duha ka DNA Pools ang gitukod sa 50 ka grabe nga mga tawo nga taas nga liog ug 50 ka grabe nga gagmay nga mga tawo sa liog. Ang panguna nga epekto sa QTL giila sa CHR07 sa pag-analisar sa BSA ug tradisyonal nga QTL mapping. Ang Kandidor sa rehiyon labi pa nga gipunting sa fine-mapping, pag-ihap sa gene nga ekspresyon ug mga eksperimento sa transgenic, nga nagpadayag nga Key Gene sa pagpugong sa liog-gitas-on, CSFNLL7.1. Gawas pa, ang Polymorphism sa CSFNLL7.1 Ang rehiyon sa promo sa CSFNLL7.1 nakit-an nga adunay kalabutan sa katugbang nga pagpahayag. Ang dugang nga pag-analisar sa phylogenetic nga gisugyot nga ang FNL7.1 Si Locus lagmit nga gikan sa India.
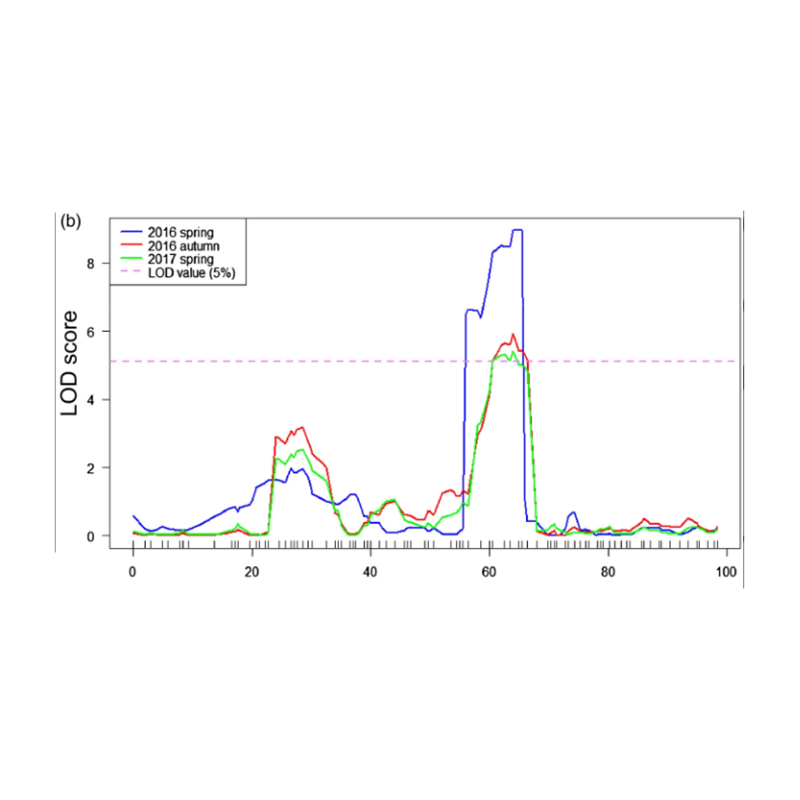
 Ang QTL-Mapping sa pag-analisar sa BSA aron mahibal-an ang Region Region nga may kalabutan sa gitas-on sa cucumber nga gitas-on |  Mga Profile sa Lod sa Cucumber Neck-Length qtl nga giila sa CHR07 |
XU, X., ET AL. "Ang nag-unang epekto nga Trait Trait Locus FNL7.1 nga nag-encode sa usa ka ulahing bahin sa embryagenesis sa daghang protina nga may kalabutan sa pipino." Itanom biotechnology journal 18.7 (2020).