BMKMANU S1000 Spatial Transcriptome
BMKMANU S1000 Spatial Transcriptome Technical Scheme
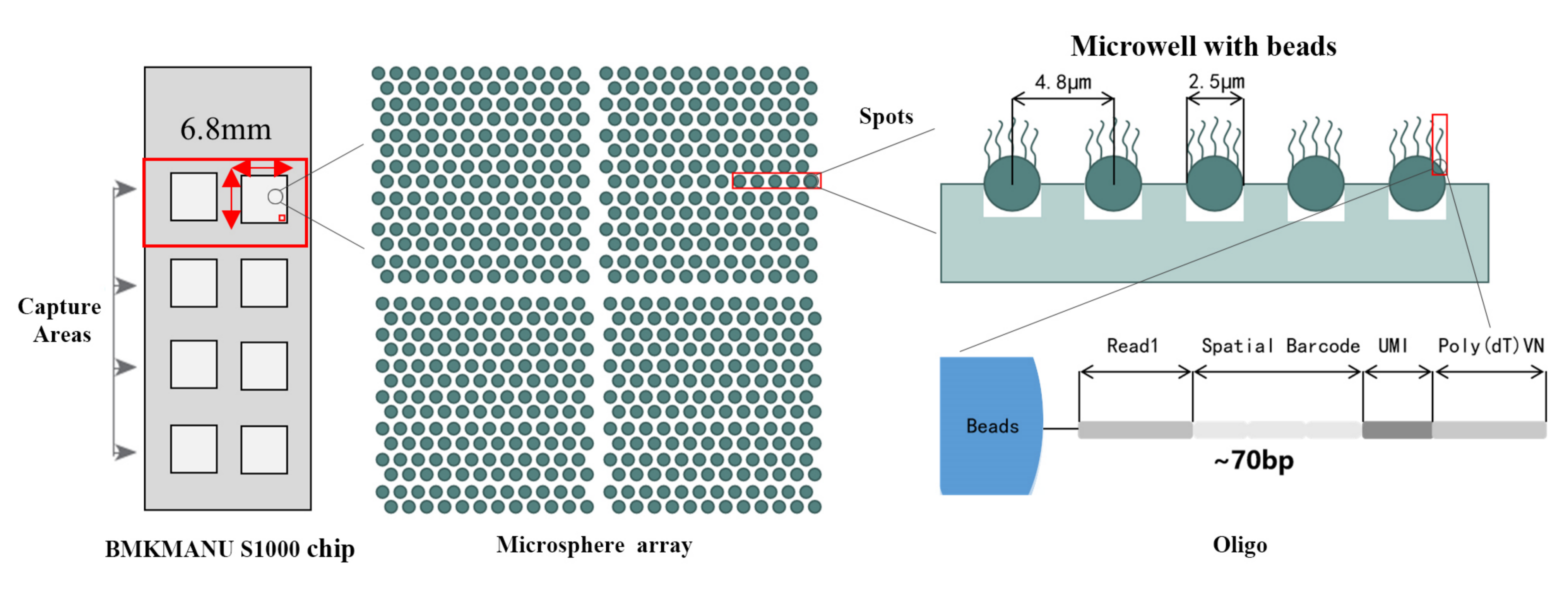
Mga bahin
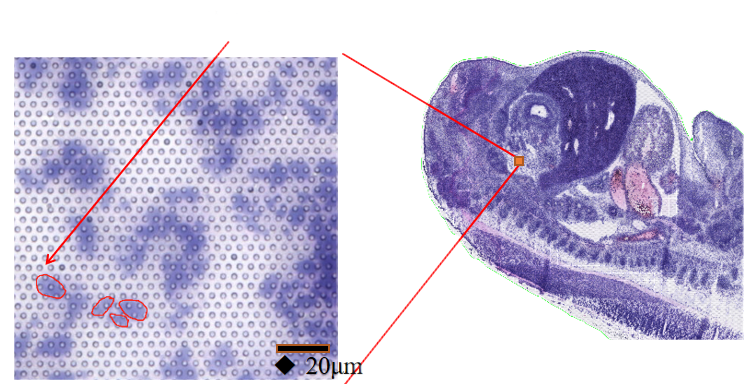
● Resolusyon: 5 µM
● Spot Diameter: 2.5 µM
● Gidaghanon sa mga spots: gibana-bana nga 2 Million
● 3 ka posible nga mga format sa lugar nga makuha: 6.8 mm * 6.8 mm, 11 mm * 11 mm o 15 mm * 20 mm
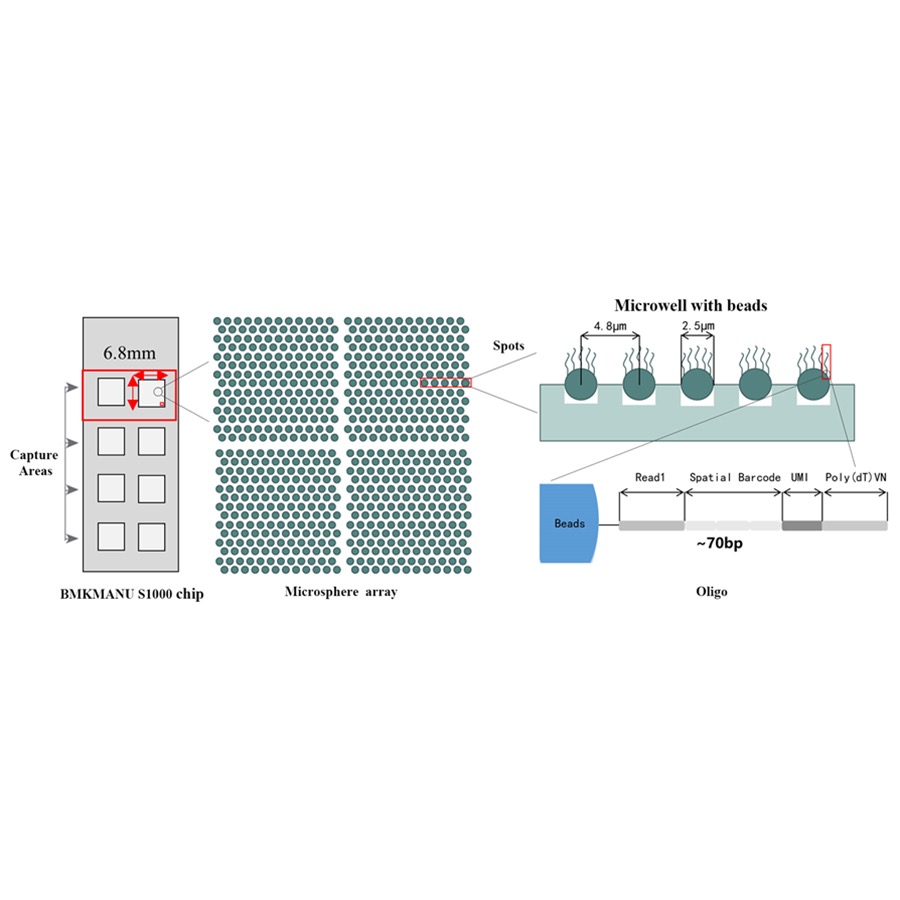
● Ang matag barcoded bead puno sa mga primer nga gilangkoban sa 4 ka seksyon:
poly(dT) tail para sa mRNA priming ug cDNA synthesis
Talagsaon nga Molecular Identifier (UMI) aron matul-id ang bias sa amplification
Spatial nga barcode
Pagbugkos nga han-ay sa partial read 1 sequencing primer
● H&E ug fluorescent staining sa mga seksyon
● Posibilidad sa paggamitteknolohiya sa pagbahinbahin sa selula: integration sa H&E staining, fluorescent staining, ug RNA sequencing aron mahibal-an ang mga utlanan sa matag cell ug hustong i-assign ang gene expression sa matag cell.
Mga bentaha sa BMKMANU S1000
●Sub-cellular nga Resolusyon: Ang matag lugar sa pagdakop adunay> 2 milyon nga spatial Barcoded Spots nga adunay diyametro nga 2.5 µm ug usa ka gilay-on nga 5 µm taliwala sa mga sentro sa lugar, nga makapaarang sa pagtuki sa spatial transcriptome nga adunay sub-cellular nga resolusyon (5 µm).
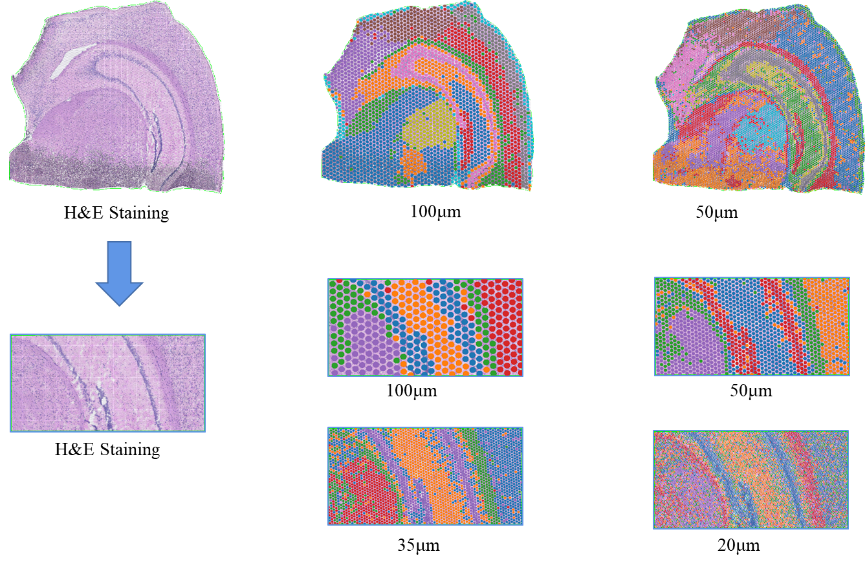
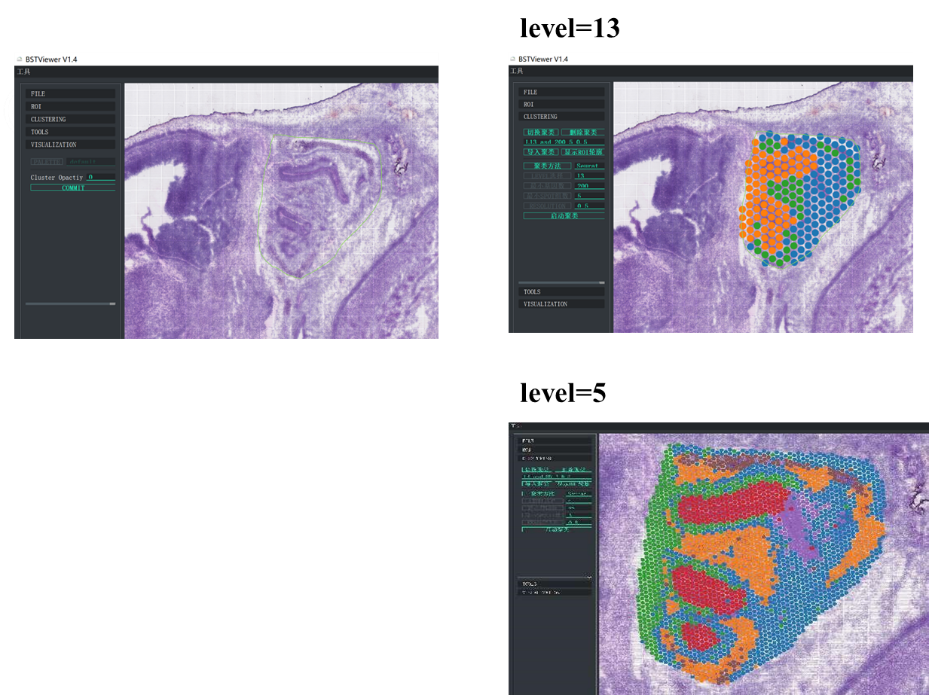
●Multi-level nga Resolution Analysis:Flexible multi-level analysis gikan sa 100 μm ngadto sa 5 μm aron masulbad ang lain-laing mga bahin sa tisyu sa labing maayo nga resolusyon.
●Posibilidad sa Paggamit sa “Three in one slide” Cell Segmentation Technology:Ang paghiusa sa fluorescence staining, H&E staining, ug RNA sequencing sa usa ka slide, ang among "three-in-one" analysis algorithm naghatag gahum sa pag-ila sa mga utlanan sa cell para sa sunod nga cell-based transcriptomics.
●Nahiuyon sa Daghang mga Sequencing Platform: Parehong NGS ug dugay na nga pagbasa nga pagkasunodsunod anaa.
●Flexible nga Disenyo sa 1-8 Active Capture Area: Ang gidak-on sa dapit sa pagdakop kay flexible, nga posible nga gamiton ang 3 ka format (6.8 mm * 6.8 mm., 11 mm * 11 mm ug 15 mm * 20 mm)
●One-stop nga Serbisyo: Gihiusa niini ang tanang kasinatian ug mga lakang nga gibase sa kahanas, lakip ang cryo-sectioning, pagmantsa, pag-optimize sa tissue, spatial barcoding, pag-andam sa librarya, sequencing, ug bioinformatics.
●Comprehensive Bioinformatics ug User-friendly Visualization sa mga Resulta:Ang package naglakip sa 29 ka mga pag-analisa ug 100+ ka taas nga kalidad nga mga numero, inubanan sa paggamit sa inhouse nga naugmad nga software aron mahanduraw ug ipahiangay ang cell splitting ug spot clustering.
●Customized Data Analysis ug Visualization: anaa alang sa lain-laing mga hangyo sa panukiduki
●Highly-skilled Technical Team: nga adunay kasinatian sa kapin sa 250 ka tipo sa tisyu ug 100+ ka espisye lakip ang tawo, ilaga, mammal, isda ug tanom.
●Real-time nga mga Update sa Tibuok Proyekto: nga adunay bug-os nga pagkontrol sa pag-uswag sa eksperimento.
●Opsyonal nga Joint Analysis nga adunay Single-cell mRNA Sequencing
Mga Detalye sa Serbisyo
|
Sampol Mga kinahanglanon
| Library |
Estratehiya sa pagkasunodsunod
| Girekomenda ang datos | Pagkontrol sa Kalidad |
| OCT-embedded cryo samples, 3 ka bloke kada sample | S1000 cDNA librarya | Illumina PE150 (ubang mga plataporma anaa) | 100K PE nga pagbasa kada 100 uM ( 60-150 Gb ) | RIN>7 |
Alang sa dugang nga mga detalye sa giya sa pag-andam sa sample ug dagan sa serbisyo, palihug ayaw pagduhaduha sa pagpakigsulti sa aEksperto sa BMKGENE
Daloy sa Trabaho sa Serbisyo
Sa yugto sa pag-andam sa sample, usa ka inisyal nga daghang pagsulay sa pagkuha sa RNA gihimo aron masiguro nga makuha ang usa ka taas nga kalidad nga RNA. Sa yugto sa pag-optimize sa tisyu ang mga seksyon nabulingan ug gitan-aw ug ang mga kondisyon sa permeabilisasyon alang sa pagpagawas sa mRNA gikan sa tisyu gi-optimize. Ang na-optimize nga protocol dayon gipadapat sa panahon sa pagtukod sa librarya, gisundan sa pagkasunod-sunod ug pagtuki sa datos.
Ang kompleto nga service workflow naglakip sa real-time nga mga update ug mga kumpirmasyon sa kliyente aron mapadayon ang usa ka responsive feedback loop, pagsiguro sa hapsay nga pagpatuman sa proyekto.
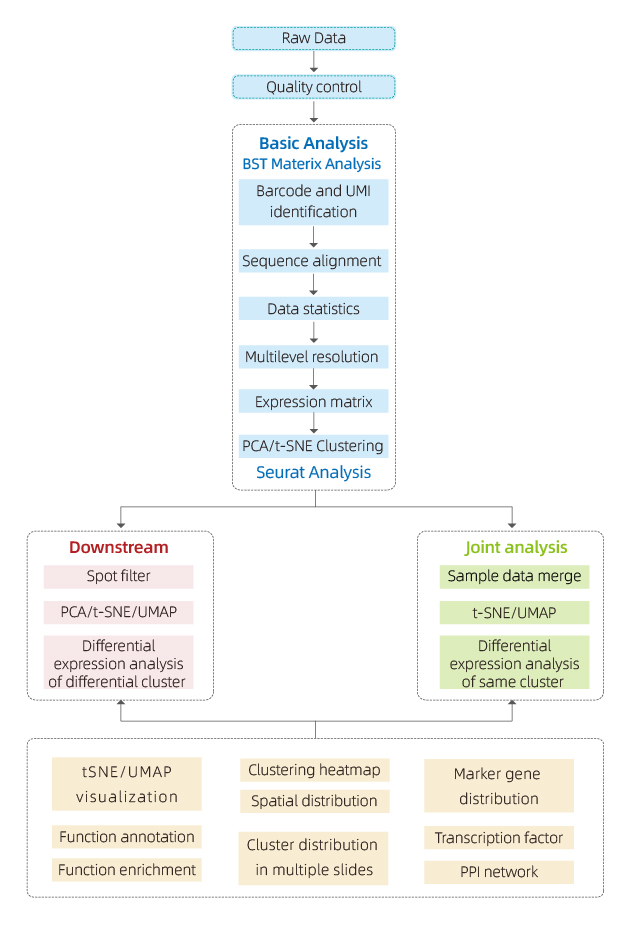
Ang datos nga namugna sa BMKMANU S1000 gisusi gamit ang software nga "BSTMatrix", nga independente nga gidisenyo sa BMKGENE, nga nagmugna og Gene Expression Matrix. Gikan didto, usa ka sumbanan nga taho ang nahimo nga naglakip sa pagkontrol sa kalidad sa datos, pagtuki sa sulud sa sulud ug pagtuki sa inter-grupo.
● Pagkontrol sa Kalidad sa Data:
Output sa datos ug pag-apod-apod sa kalidad nga marka
Ang pagkakita sa gene matag lugar
Pagsakop sa tisyu
● Inner-sample analysis:
Pagkadato sa gene
Spot clustering, lakip ang pagkunhod sa pagtuki sa dimensyon
Differential expression analysis tali sa clusters: pag-ila sa marker genes
Functional nga anotasyon ug pagpalambo sa mga marker gene
● Inter-grupo nga pagtuki:
Re-kombinasyon sa mga spots gikan sa duha ka sample (eg. sakit ug kontrol) ug re-cluster
Pag-ila sa mga marker genes alang sa matag cluster
Functional nga anotasyon ug pagpalambo sa mga marker gene
Differential Expression sa parehas nga cluster tali sa mga grupo
Dugang pa, gimugna sa BMKGENE ang "BSTViewer" usa ka himan nga mahigalaon sa gumagamit nga makapahimo sa tiggamit nga mahanduraw ang ekspresyon sa gene ug pag-cluster sa lugar sa lainlaing mga resolusyon.
Ang BMKGene nakahimo og software alang sa user friendly visualization
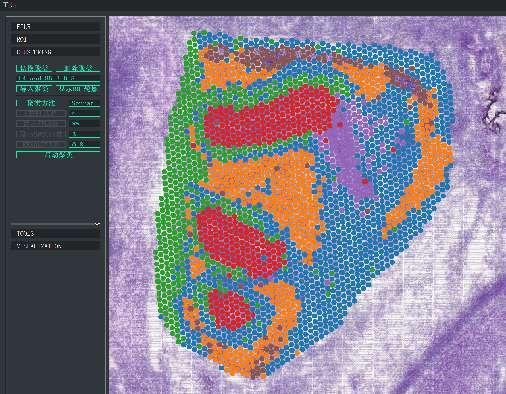
BSTViewer spot clustering sa multi-level nga resolusyon
BSTellViewer: awtomatiko ug manwal nga pagbahin sa cell
Inner-sample nga Pagtuki
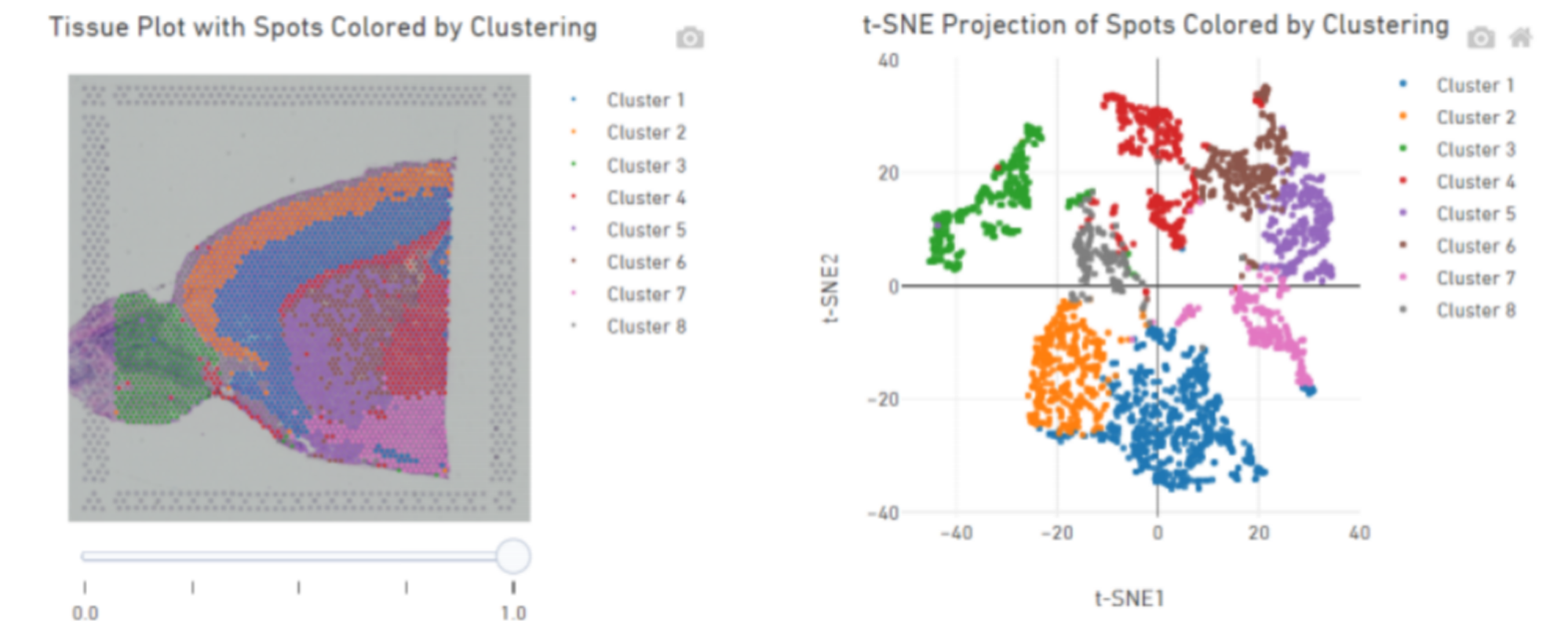
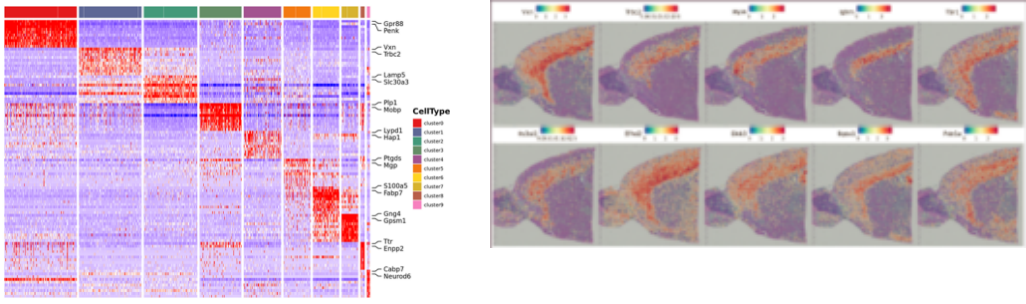
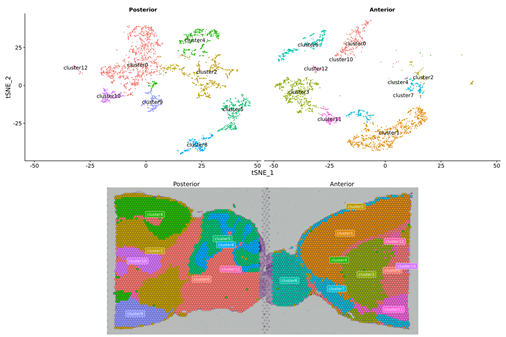
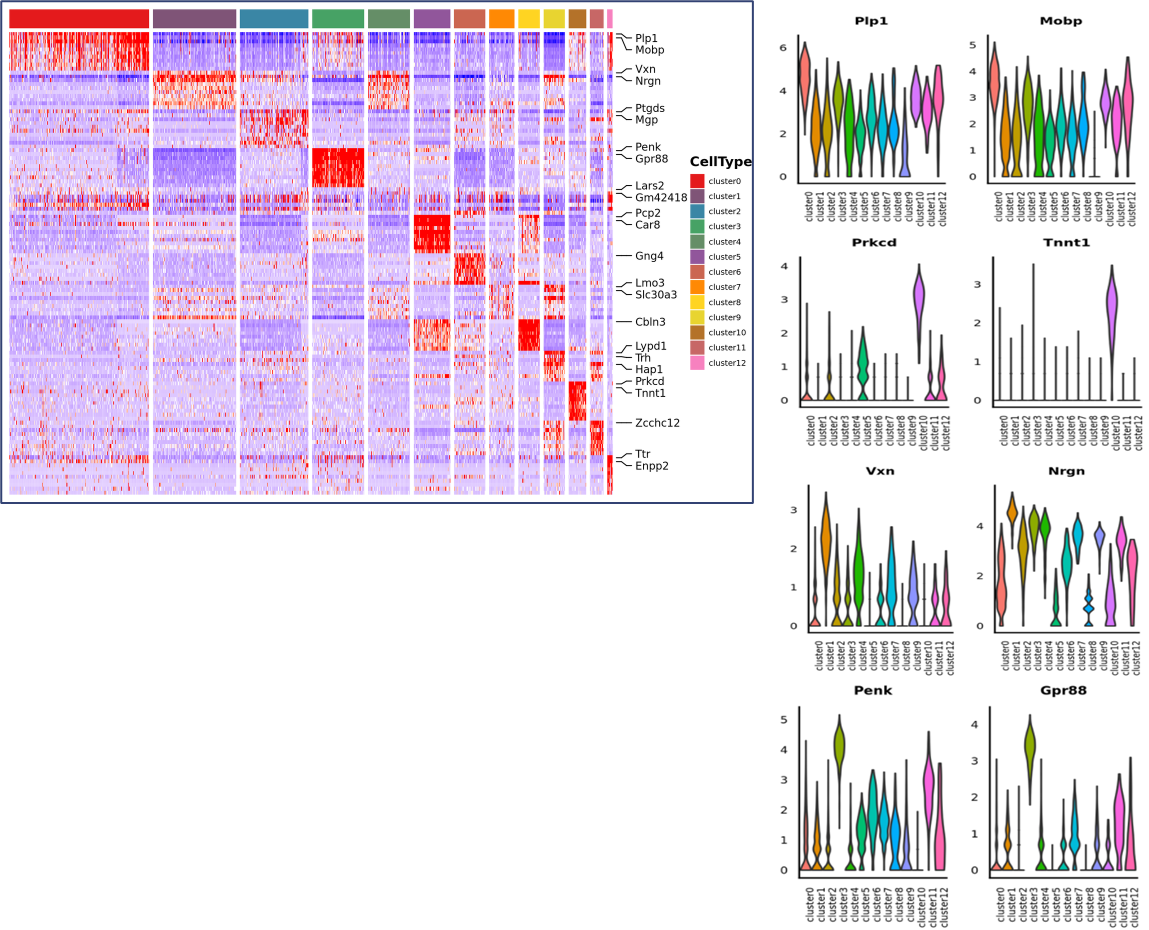
Spot clustering:
Pag-ila sa mga gene sa marker ug spatial distribution:
Pagtuki sa Inter-grupo
Ang kombinasyon sa datos gikan sa duha ka grupo ug re-cluster:
Mga marker nga gene sa bag-ong mga cluster:
Susihon ang mga pag-uswag nga gipadali sa mga serbisyo sa spatial transcriptomics sa BMKGene gamit ang teknolohiya sa BMKManu S1000 sa kini nga gipakita nga publikasyon:
Kanta, X. et al. (2023) 'Ang spatial transcriptomics nagpadayag sa light-induced chlorenchyma cells nga nalambigit sa pagpalambo sa shoot regeneration sa tomato callus',Mga pamaagi sa National Academy of Sciences sa United States of America, 120(38), p. e2310163120. doi: 10.1073/pnas.2310163120
Ikaw, Y. et al. (2023) 'Sistematikong pagtandi sa sequencing-based spatial transcriptomic nga mga pamaagi',bioRxiv, p. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.