Seqüenciació del transcriptoma sencer - Illumina
Funcions
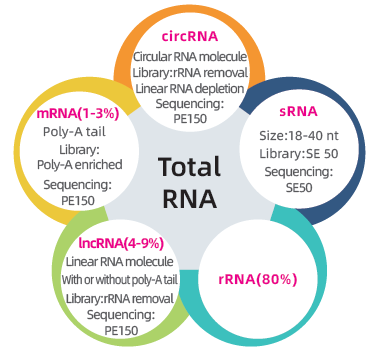
● Biblioteca doble per seqüenciar el transcriptoma complet: esgotament de l'RNA seguit de la preparació de la biblioteca PE150
● Anàlisi de bioinformàtica completa de l'ARNm, lncRNA, circrna i miRNA en informes de bioinformàtica separats
● Anàlisi conjunta de tota l'expressió d'ARN en un informe combinat, inclosa l'anàlisi de Cerna Networks.
Avantatges del servei
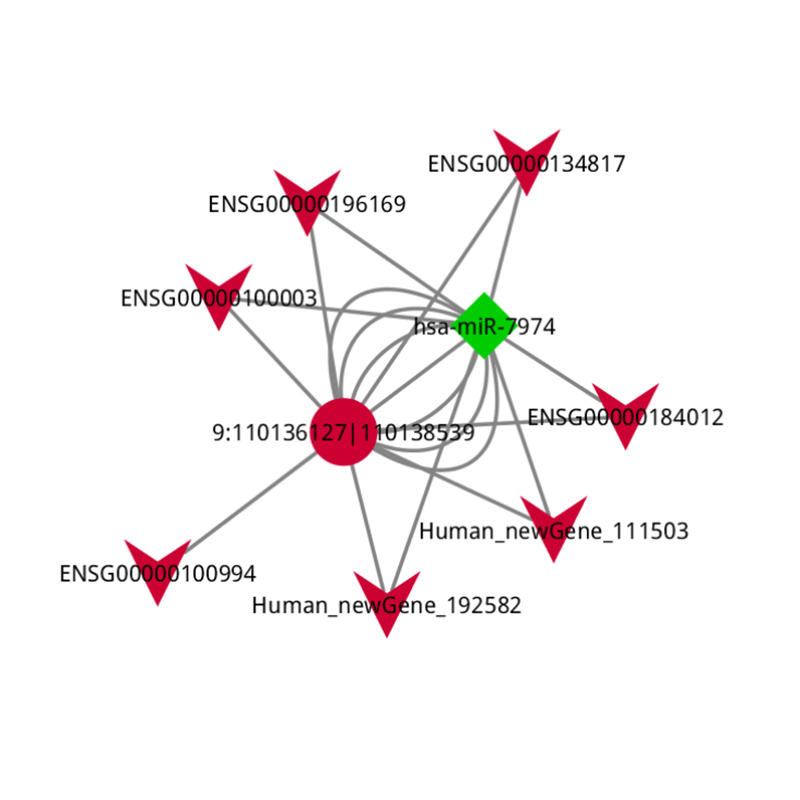
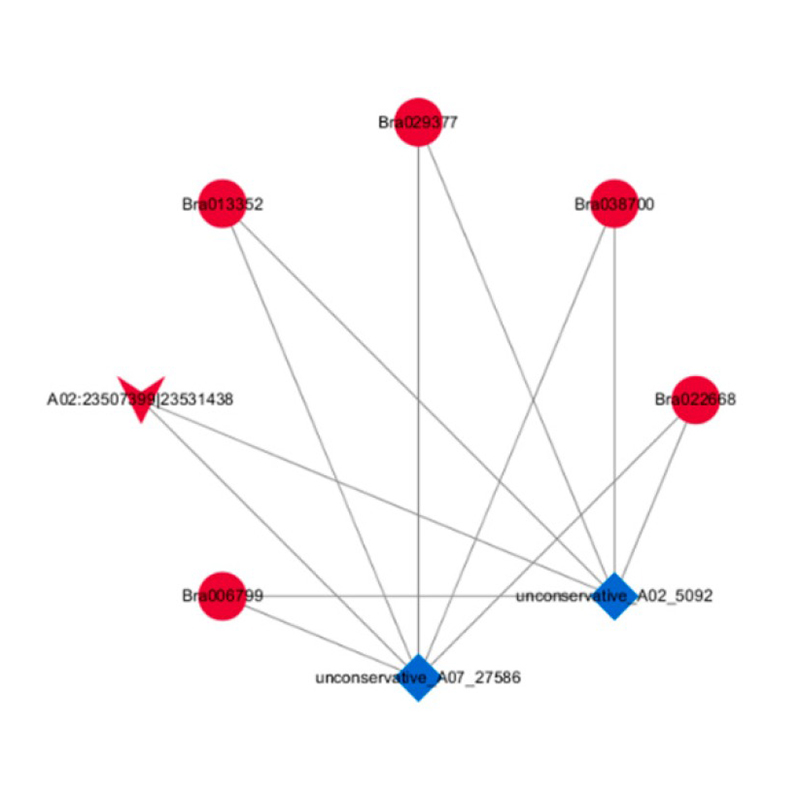
●Anàlisi en profunditat de les xarxes reguladores: L’anàlisi de la xarxa de Cerna està habilitada per la seqüenciació conjunta d’ARNm, lncRNA, circrna i miRNA i per un flux de treball bioinformàtic exhaustiu.
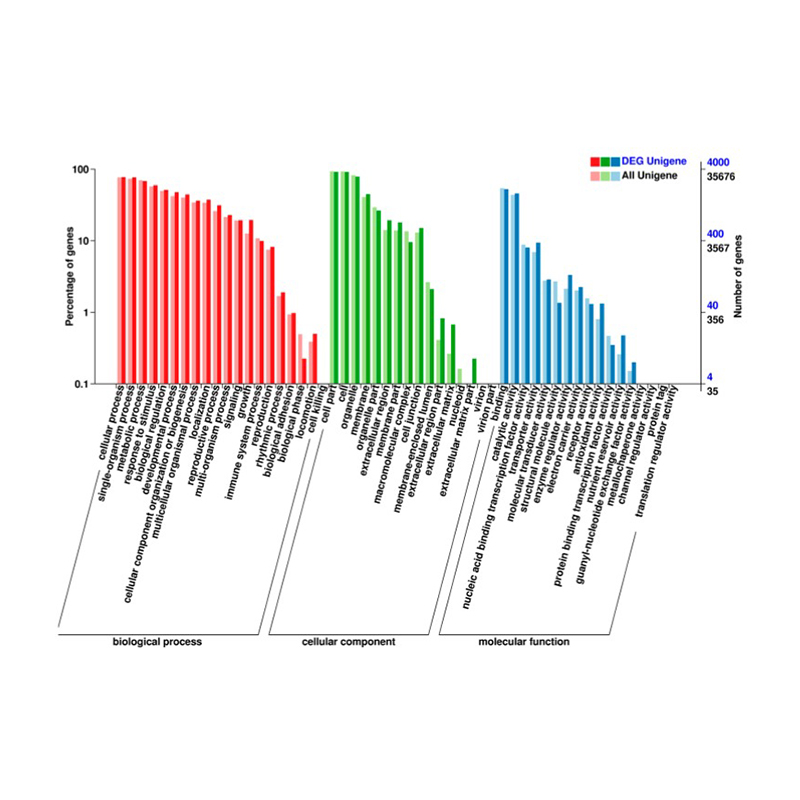
●Anotació completa: Utilitzem diverses bases de dades per anotar funcionalment els gens expressats de manera diferent (DEG) i realitzar l’anàlisi d’enriquiment corresponent, proporcionant informació sobre els processos cel·lulars i moleculars subjacents a la resposta del transcriptoma.
●Àmplia experiència: Amb un historial de tancar amb èxit més de 2100 projectes de transcriptoma sencers en diversos dominis de recerca, el nostre equip aporta una gran experiència a tots els projectes.
●Control de qualitat rigorós: Implementa punts de control bàsics a totes les etapes, des de la preparació de mostres i biblioteques fins a la seqüenciació i la bioinformàtica. Aquest seguiment minuciós garanteix el lliurament de resultats constantment de gran qualitat.
●Suport post-vendes: El nostre compromís s’estén més enllà de la finalització del projecte amb un període de servei posterior a tres mesos. Durant aquest temps, oferim un seguiment del projecte, assistència de resolució de problemes i sessions de Q&A per abordar qualsevol consulta relacionada amb els resultats
Requisits de mostra i lliurament
| Biblioteca | Estratègia de seqüenciació | Dades recomanades | Control de qualitat |
| rRNA esgotat | Illumina PE150 | 16 GB | Q30≥85% |
| Mida seleccionada | Il·lumina se50 | 10-20m de lectures |
Requisits de mostra:
Nucleòtids:
| Conc. (NG/μL) | Quantitat (μg) | Puresa | Integritat |
| ≥ 80 | ≥ 1,6 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Contaminació limitada o nul·la de proteïnes o ADN que es mostra al gel. | Rin≥6.0 5.0≥28s/18S≥1.0; elevació de base limitada o sense base |
Lliurament de mostres recomanat
Contenidor: Tub de centrífuga de 2 ml (no es recomana la làmina de llauna)
Etiquetatge de mostres: grup+replica per exemple A1, A2, A3; B1, B2, B3.
Enviament:
1. Gole sec: cal que les mostres siguin envasades en bosses i enterrades en gel sec.
2. Tubs rnastables: les mostres d’ARN es poden assecar en el tub d’estabilització d’ARN (per exemple, RNastable®) i enviades a temperatura ambient.
Flux de treball del servei

Disseny d'experiments

Lliurament de mostra

Extracció d’ARN

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades

Serveis postvenda
Bioinformàtica
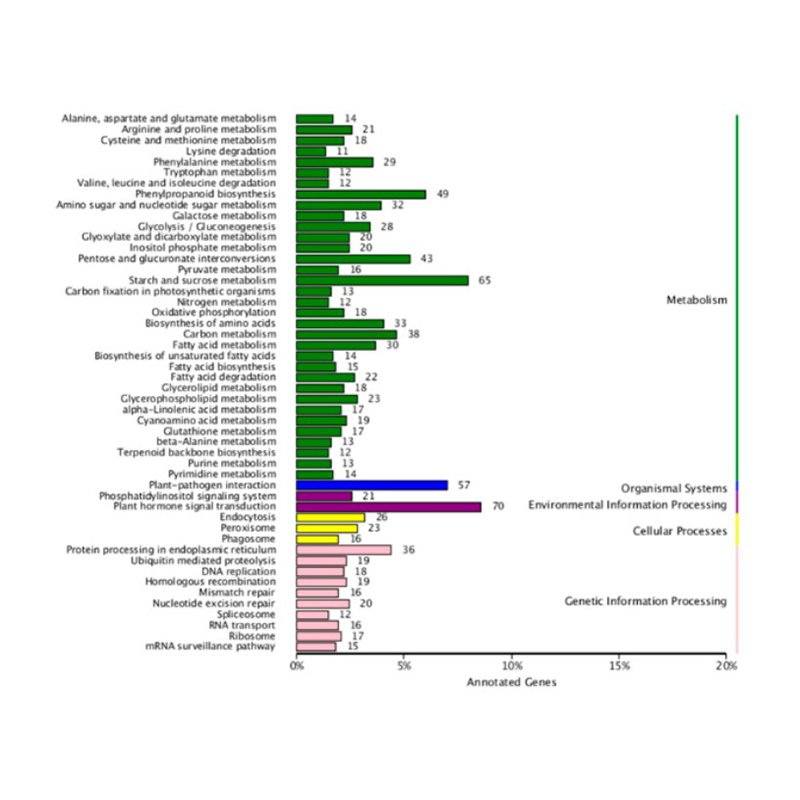
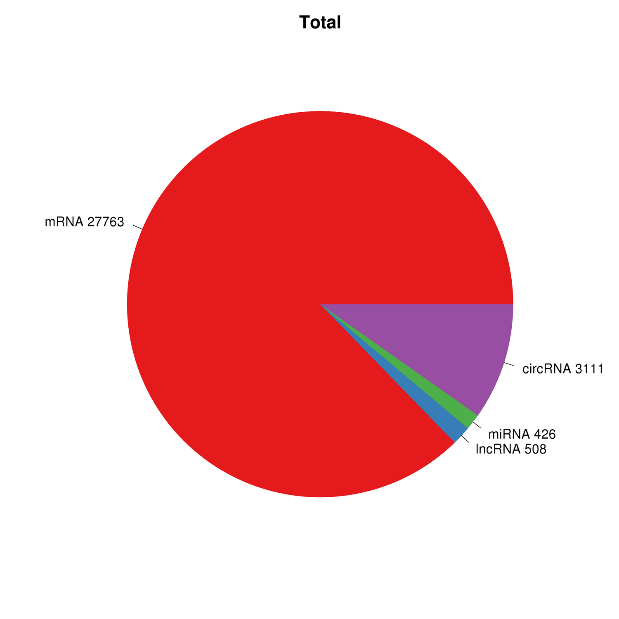
Visió general de l'expressió d'ARN
Gens expressats de manera diferent
Anàlisi de Cerna
Exploreu els avenços de la investigació facilitats pels serveis de seqüenciació de transcriptomes sencers de BMKGENE mitjançant una col·lecció de publicacions curat.
Dai, Y. et al. (2022) "Perfils d'expressió integrals de mRNAs, lncRNAs i miRNAs en la malaltia de Kashin-Beck identificats per la seqüenciació d'ARN", Molecular Omics, 18 (2), pp. 154–166. doi: 10.1039/d1mo00370d.
Liu, N. Nan et al. (2022) "Anàlisi de transcriptomes de longitud completa de la resistència al fred de l'Apis cerana a la muntanya Changbai durant el període de sobreposició.", Gene, 830, pp. 146503–146503. doi: 10.1016/j.gene.2022.146503.
Wang, XJ et al. (2022) "Priorització basada en la integració multi-omica de les xarxes de regulació endògena de RNA competitives en càncer de pulmó de cèl·lules petites: característiques moleculars i candidats a fàrmacs", Frontiers in Oncology, 12, pàg. 904865. Doi: 10.3389/Fonc.2022.904865/Bibtex.
Xu, P. et al. (2022) 'L'anàlisi integrada dels perfils d'expressió de LNCRNA/CIRCRNA-miRNA-MRNA revela visions noves sobre mecanismes potencials en resposta a nematodes de nus arrel en cacauet', BMC Genomics, 23 (1), pp. 1-12. doi: 10.1186/s12864-022-08470-3/figures/7.
Yan, Z. et al. (2022) "La seqüenciació d'ARN del transcriptoma sencer posa de manifest els mecanismes moleculars associats al manteniment de la qualitat de la recol·lecció en el bròquil per irradiació del LED vermell", Biologia i tecnologia de post-collita, 188, pàg. 111878. Doi: 10.1016/j.postharvbio.2022.111878.