Seqüenciació de bisulfits de representació reduïda (RRBS)
Característiques del servei
● Requereix un genoma de referència.
● L'ADN lambda s'utilitza per controlar l'eficiència de conversió de bisulfits.
● També es controla l'eficiència de la digestió MspI.
● Digestió enzimàtica doble per a mostres de plantes.
● Seqüenciació a Illumina NovaSeq.
Avantatges del servei
●Alternativa rendible i eficient a WGBS: permeten que l'anàlisi es dugui a terme a un cost més baix i amb menys requisits de mostra.
●Plataforma completa:proporcionar un servei excel·lent únic, des del processament de mostres, la construcció de biblioteques i la seqüenciació fins a l'anàlisi bioinformàtica.
●Àmplia experiència: amb projectes de seqüenciació RRBS completats amb èxit en una àmplia gamma d'espècies, BMKGENE aporta més d'una dècada d'experiència, un equip d'anàlisi altament qualificat, contingut complet i un excel·lent suport postvenda.
Especificacions del servei
| Biblioteca | Estratègia de seqüenciació | Sortida de dades recomanada | Control de qualitat |
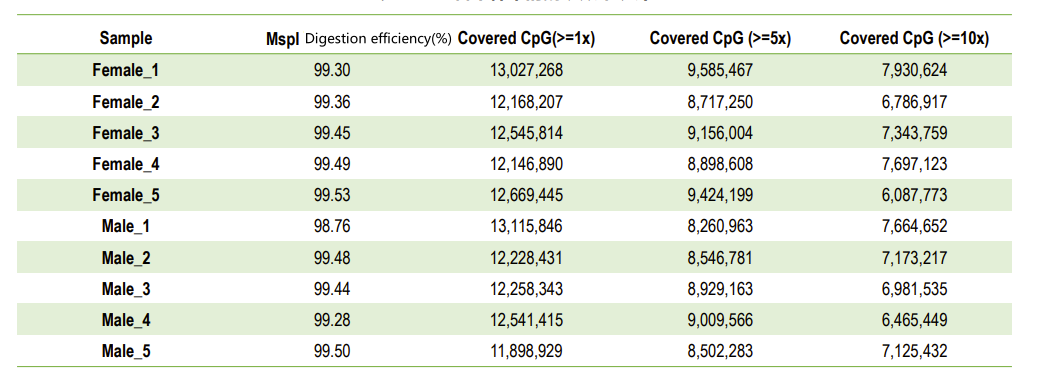
| Biblioteca digerida amb MspI i tractada amb bisulfit | Il·lumina PE150 | 8 Gb | Q30 ≥ 85% Conversió de bisulfit > 99% Eficiència de tall MspI > 95% |
Exemples de requisits
| Concentració (ng/µL) | Quantitat total (µg) |
| |
| ADN genòmic | ≥ 30 | ≥ 1 | Degradació o contaminació limitada |
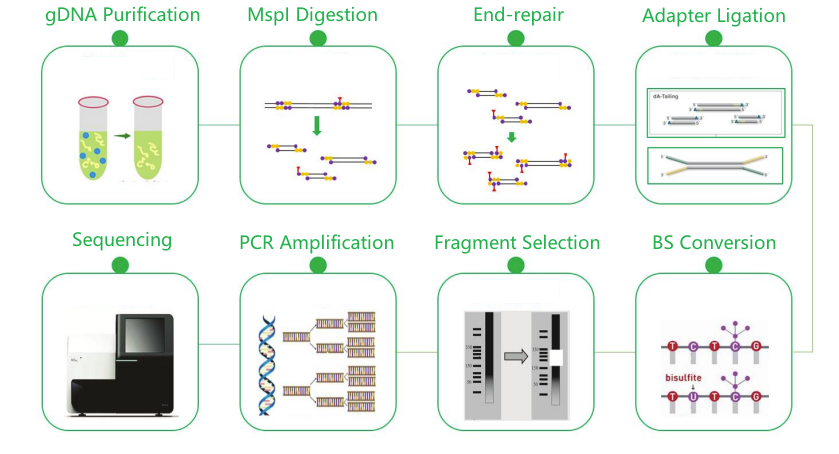
Flux de treball del servei

Lliurament de la mostra

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades
Serveis postvenda
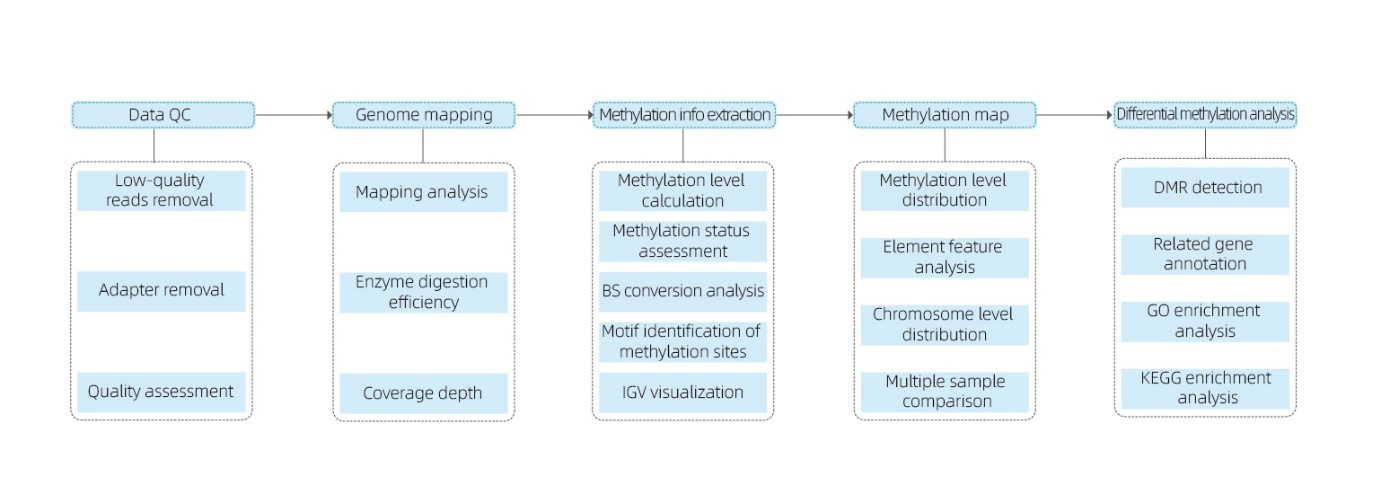
Inclou l'anàlisi següent:
● Control de qualitat de la seqüenciació en brut;
● Cartografia al genoma de referència;
● Detecció de bases metilades 5mC i identificació de motius;
● Anàlisi de la distribució de la metilació i comparació de mostres;
● Anàlisi de regions diferencialment metilades (DMR);
● Anotació funcional de gens associats a DMR.
Control de qualitat: eficiència de la digestió (en mapes del genoma)
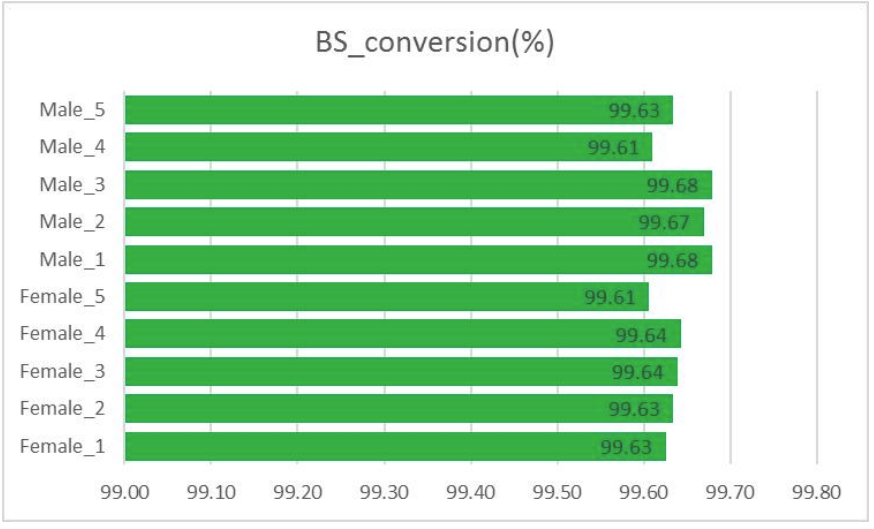
Control de qualitat: conversió de bisulfit (en extracció d'informació de metilació)
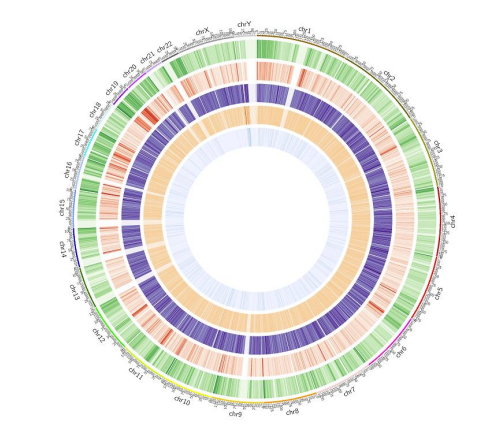
Mapa de metilació: distribució a tot el genoma de metilació de 5 mC
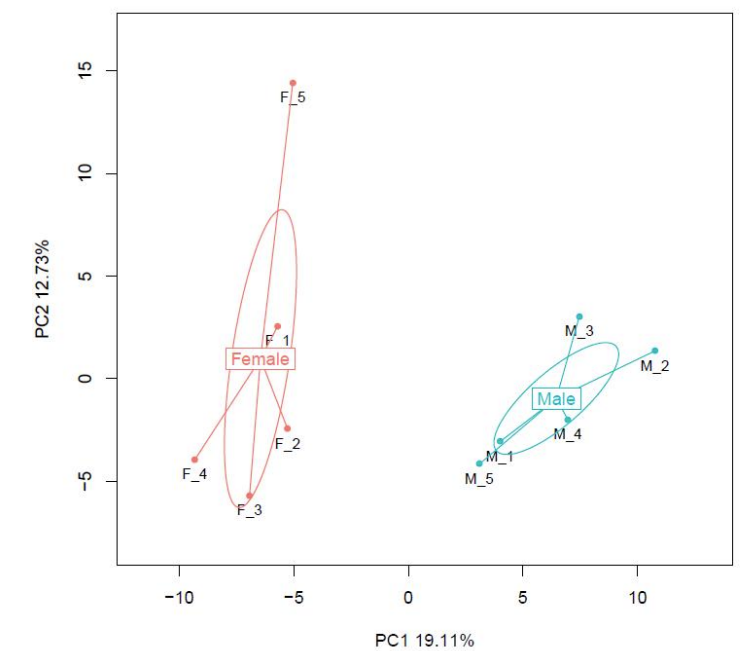
Comparació de mostres: Anàlisi de components principals
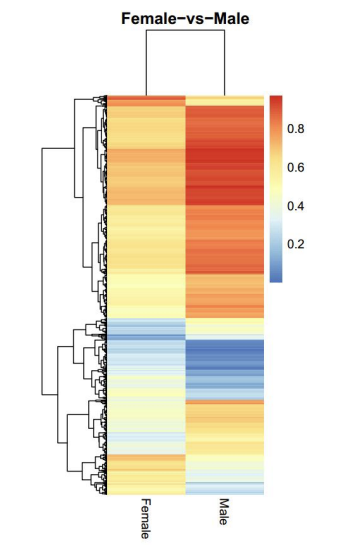
Anàlisi de regions diferencialment metilades (DMRs): mapa de calor
Exploreu els avenços de la investigació facilitats pels serveis de seqüenciació de bisulfits del genoma sencer de BMKGene mitjançant una col·lecció de publicacions curada.
Li, Z. et al. (2022) "Reprogramació d'alta fidelitat en cèl·lules semblants a Leydig per activació de CRISPR i factors paracrins",PNAS Nexus, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. et al. (2023) "Anàlisi de metilació de l'ADN a tot el genoma de la composició corporal en bessons monozigòtics xinesos",Revista Europea d'Investigació Clínica, 53(11), pàg. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) "Metilació de l'ADN i relació cintura-maluc: un estudi d'associació a tot l'epigenoma en bessons monozigòtics xinesos",Revista d'Investigació Endocrinològica, 45(12), pàg. 2365–2376. doi: 10.1007/S40618-022-01878-4.