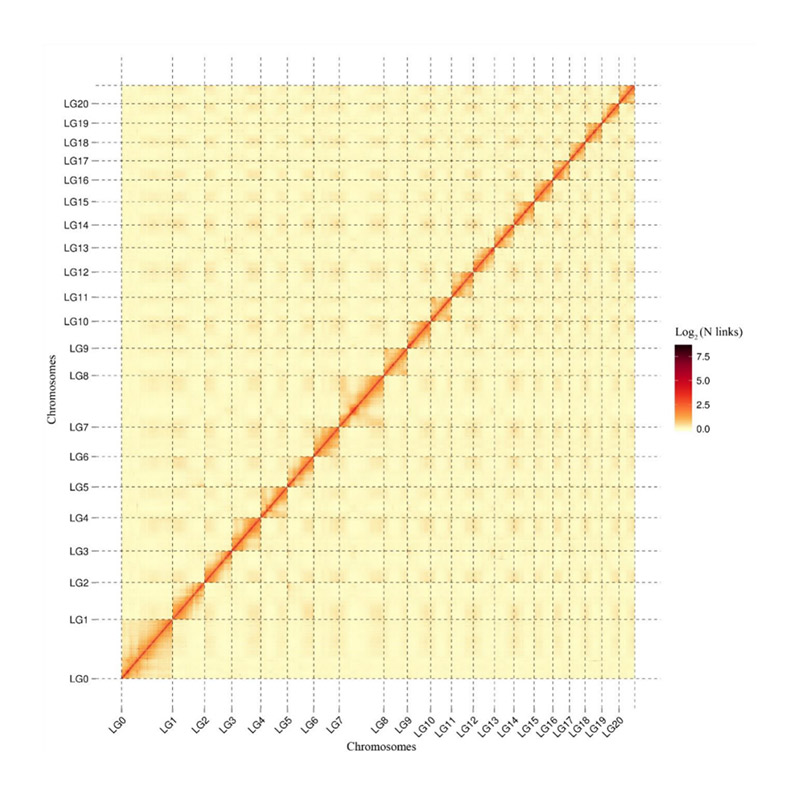
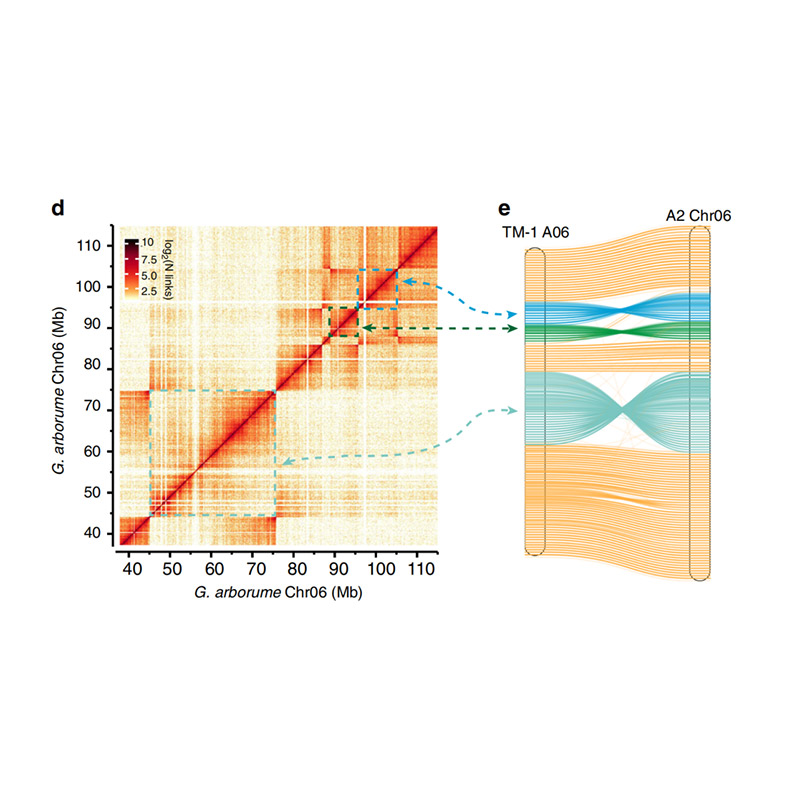
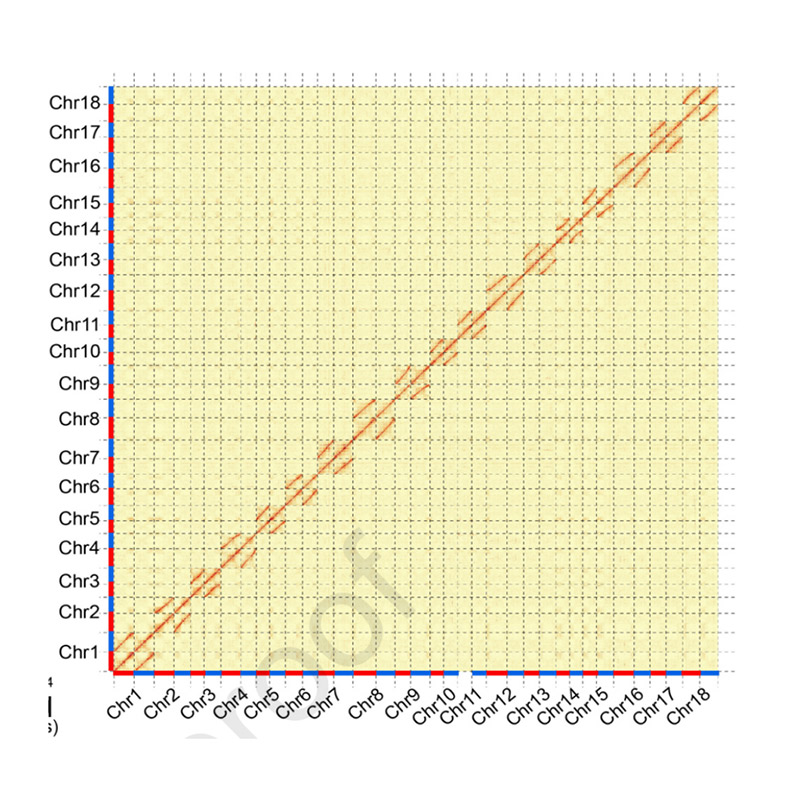
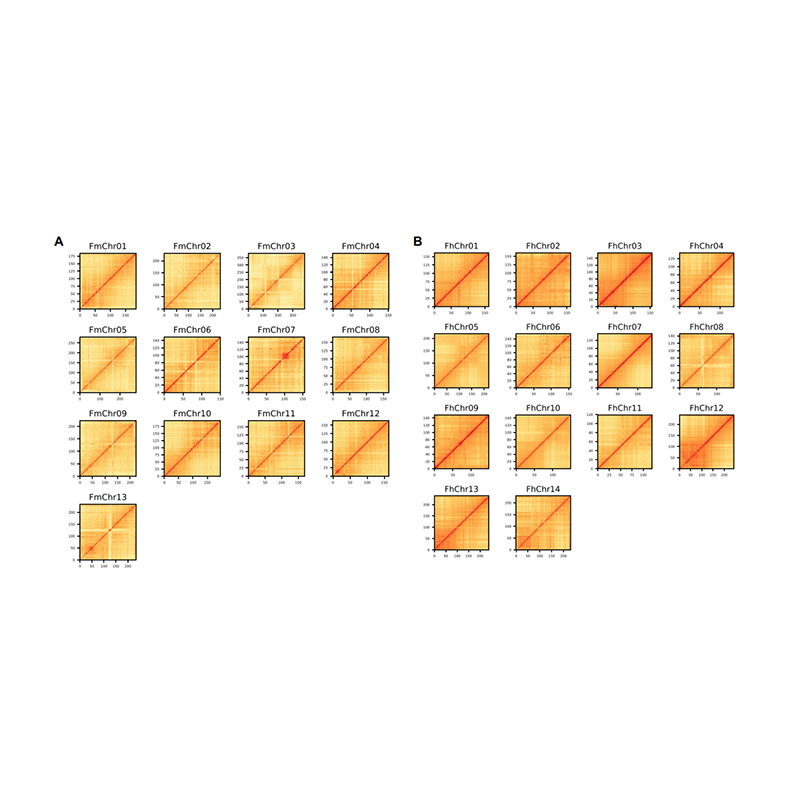
Muntatge del genoma basat en Hi-C
Característiques del servei
● Seqüenciació a Illumina NovaSeq amb PE150.
● El servei requereix que mostres de teixit, en lloc d'àcids nucleics extrets, s'entretinguin amb formaldehid i conservin les interaccions ADN-proteïna.
● L'experiment Hi-C implica la restricció i la reparació final dels extrems enganxosos amb biotina, seguida de la circularització dels extrems roms resultants tot preservant les interaccions. A continuació, l'ADN es tira cap avall amb perles d'estreptavidina i es purifica per a la posterior preparació de la biblioteca.
Avantatges del servei

Visió general de Hi-C
(Lieberman-Aiden E et al.,Ciència, 2009)
●Eliminació de la necessitat de dades genètiques de població:Hi-C substitueix la informació essencial necessària per a l'ancoratge contig.
●Alta densitat de marcadors:donant lloc a una alta relació d'ancoratge contig per sobre del 90%.
●Àmplia experiència i registres de publicacions:BMKGene té una àmplia experiència amb més de 2000 casos de muntatge del genoma Hi-C de 1000 espècies diferents i diverses patents. Més de 200 casos publicats tenen un factor d'impacte acumulat superior a 2000.
●Equip de bioinformàtica altament qualificat:amb patents internes i drets d'autor de programari per a experiments Hi-C i anàlisi de dades, el programari de dades de visualització de desenvolupament propi permet moure, invertir, revocar i refer manualment blocs.
●Suport postvenda:El nostre compromís s'estén més enllà de la finalització del projecte amb un període de servei postvenda de 3 mesos. Durant aquest temps, oferim seguiment del projecte, assistència per a la resolució de problemes i sessions de preguntes i respostes per resoldre qualsevol consulta relacionada amb els resultats.
●Anotació integral: fem servir diverses bases de dades per anotar funcionalment els gens amb variacions identificades i realitzar l'anàlisi d'enriquiment corresponent, proporcionant informació sobre diversos projectes de recerca.
Especificacions del servei
| Preparació de la biblioteca | Estratègia de seqüenciació | Sortida de dades recomanada | Control de qualitat |
| Biblioteca Hi-C | Illumina NovaSeq PE150 | 100x | Q30 ≥ 85% |
Exemples de requisits
| Teixit | Import requerit |
| Vísceres d'animals | ≥ 2 g |
| Múscul animal | |
| Sang de mamífers | ≥ 2 ml |
| Sang d'au/Peix | |
| Planta- Fulla fresca | ≥ 3 g |
| Cèl·lules cultivades | ≥ 1x107 |
| Insecte | ≥ 2 g |
Flux de treball del servei

Disseny d'experiments

Lliurament de la mostra

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades

Serveis postvenda
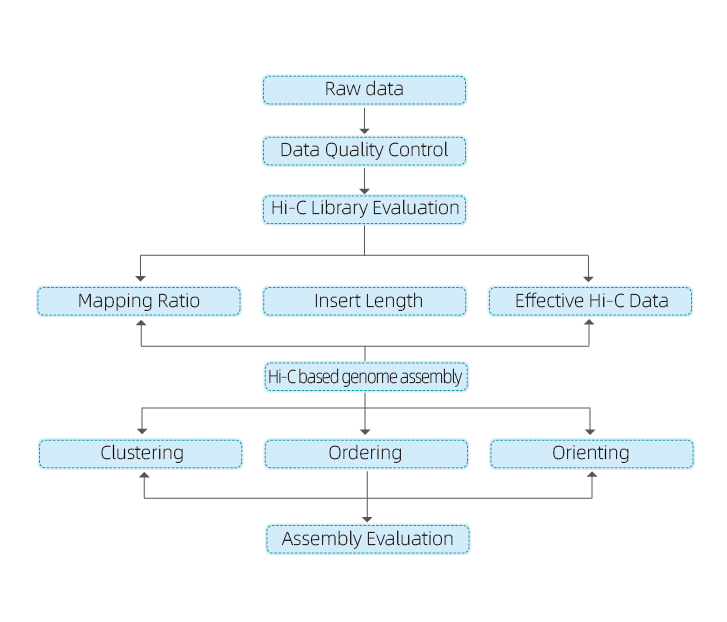
1) QC de dades en brut
2) QC de la biblioteca Hi-C: estimació d'interaccions Hi-C vàlides
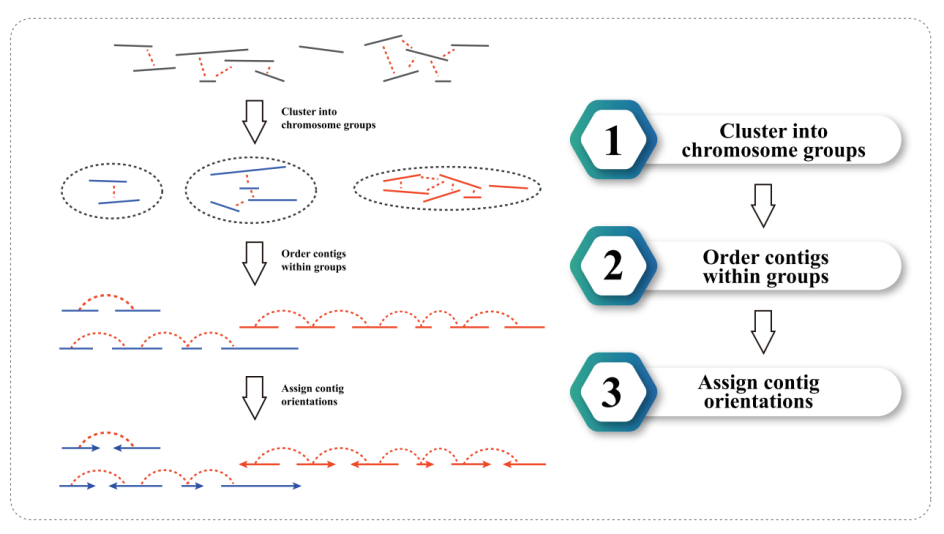
3) Muntatge Hi-C: agrupació de contigs en grups, seguit d'ordenació contig dins de cada grup i assignació d'orientació contig
4) Avaluació Hi-C
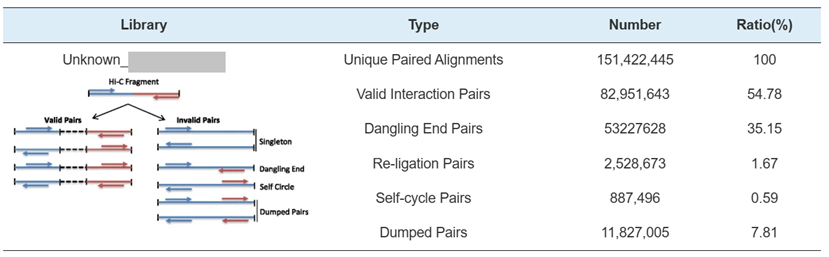
Hi-C Library QC: estimació de parells d'interacció vàlids Hi-C
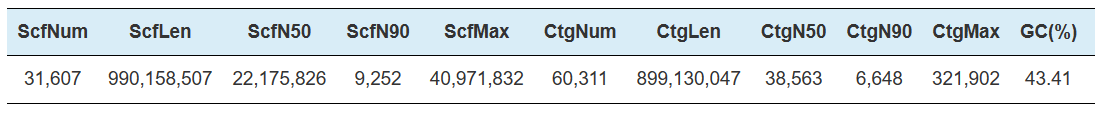
Hi-C Assembly - estadístiques
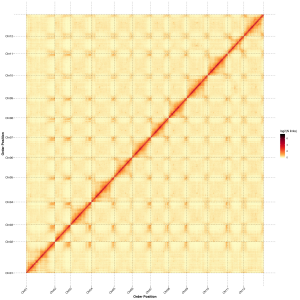
Avaluació posterior al muntatge: mapa de calor de la intensitat del senyal entre contenidors
Exploreu els avenços facilitats pels serveis de muntatge Hi-C de BMKGene mitjançant una col·lecció de publicacions seleccionada.
Tian, T. et al. (2023) "Conjunt del genoma i dissecció genètica d'un germoplasma de blat de moro resistent a la sequera prominent", Nature Genetics 2023 55:3, 55(3), pàgs. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) "Una assemblea a escala cromosòmica del genoma de l'abella asiàtica Apis cerana", Frontiers in Genetics, 11, pàg. 524140. doi: 10.3389/FGENE.2020.00279/BIBTEX.
Zhang, F. et al. (2023) "Revelar l'evolució de la biosíntesi d'alcaloides de tropà mitjançant l'anàlisi de dos genomes de la família de les Solanàcies", Nature Communications 2023 14:1, 14(1), pàgs. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) 'Genomes of the Banyan Tree and Pollinator Wasp Provide Insights into Fig-Wasp Coevolution', Cell, 183(4), pàg. 875-889.e17. doi: 10.1016/J.CELL.2020.09.043