Seqüenciació d'ARNm de longitud completa-Nanopore
Característiques
● Captura d'ARNm poli-A seguida de síntesi d'ADNc i preparació de biblioteques
● Seqüenciació de les transcripcions integrals
● Anàlisi bioinformàtica basada en l'alineació amb un genoma de referència
● L'anàlisi bioinformàtica inclou no només l'expressió a nivell de gens i isoformes, sinó també l'anàlisi de lncRNA, fusions de gens, poliadenilació i estructura gènica.
Avantatges del servei
●Quantificació de l'expressió a nivell d'isoformes: permet una anàlisi detallada i precisa de l'expressió, revelant el canvi que es pot emmascarar quan s'analitza tota l'expressió gènica
●Demandes de dades reduïdes:En comparació amb la seqüenciació de nova generació (NGS), la seqüenciació de Nanopore presenta requisits de dades més baixos, permetent nivells equivalents de saturació de quantificació de l'expressió gènica amb dades més petites.
●Major precisió de la quantificació de l'expressió: tant a nivell de gens com d'isoformes
●Identificació d'informació transcriptòmica addicional: poliadenilació alternativa, gens de fusió i lcnRNA i els seus gens diana
●Àmplia experiència: El nostre equip aporta una gran experiència a cada projecte, havent completat més de 850 projectes de transcriptoma de llarga durada Nanopore i processat més de 8.000 mostres.
●Suport postvenda: el nostre compromís s'estén més enllà de la finalització del projecte amb un període de servei postvenda de 3 mesos. Durant aquest temps, oferim seguiment del projecte, assistència per a la resolució de problemes i sessions de preguntes i respostes per resoldre qualsevol consulta relacionada amb els resultats.
Requisits de mostra i lliurament
| Biblioteca | Estratègia de seqüenciació | Dades recomanades | Control de qualitat |
| Poly A enriquit | Il·lumina PE150 | 6/12 Gb | Puntuació de qualitat mitjana: Q10 |
Requisits de mostra:
Nucleòtids:
| Conc. (ng/μl) | Quantitat (μg) | Puresa | Integritat |
| ≥ 100 | ≥ 1,0 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Es mostra al gel una contaminació limitada o nul·la de proteïnes o ADN. | Per a plantes: RIN≥7,0; Per a animals: RIN≥7,5; 5,0≥28S/18S≥1,0; elevació de la línia de base limitada o nul·la |
● Plantes:
Arrel, tija o pètal: 450 mg
Fulla o llavor: 300 mg
Fruita: 1,2 g
● Animal:
Cor o intestí: 300 mg
Vísceres o cervell: 240 mg
Múscul: 450 mg
Ossos, cabell o pell: 1 g
● Artròpodes:
Insectes: 6g
Crustacis: 300 mg
● Sang sencera: 1 tub
● Cèl·lules: 106 cèl·lules
Lliurament de mostres recomanat
Envàs: tub de centrífuga de 2 ml (no es recomana paper d'alumini)
Etiquetatge de la mostra: grup+rèplica, p. ex. A1, A2, A3; B1, B2, B3.
Enviament:
1. Gel sec: les mostres s'han d'envasar en bosses i enterrar-les en gel sec.
2. Tubs RNAstable: les mostres d'ARN es poden assecar en un tub d'estabilització d'ARN (per exemple, RNAstable®) i enviar-se a temperatura ambient.
Flux de treball del servei
Nucleòtids:

Lliurament de la mostra

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades

Serveis postvenda
Flux de treball del servei
teixit:

Disseny d'experiments

Lliurament de la mostra

Extracció d'ARN

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades

Serveis postvenda
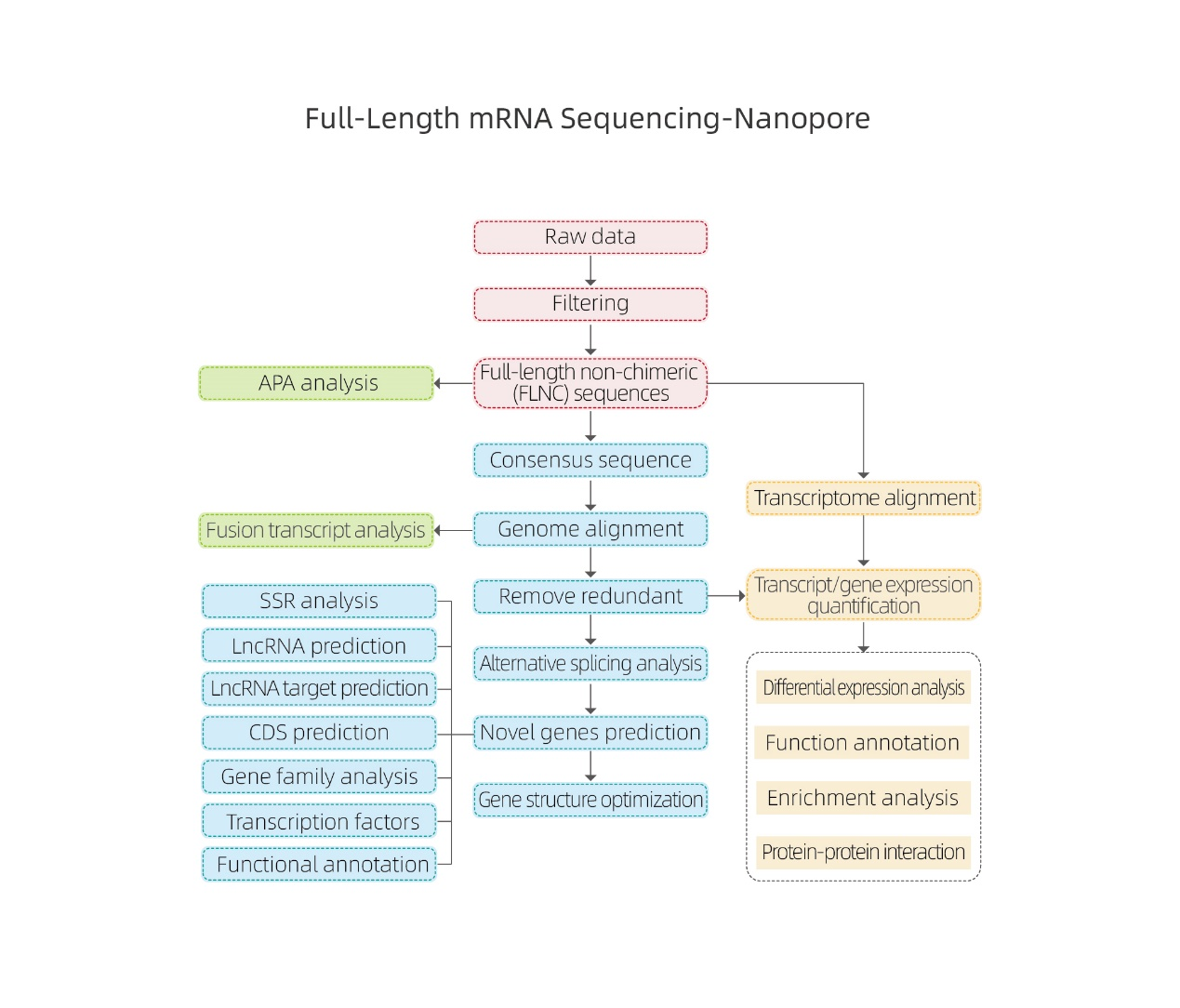
● Tractament de dades en brut
● Identificació de la transcripció
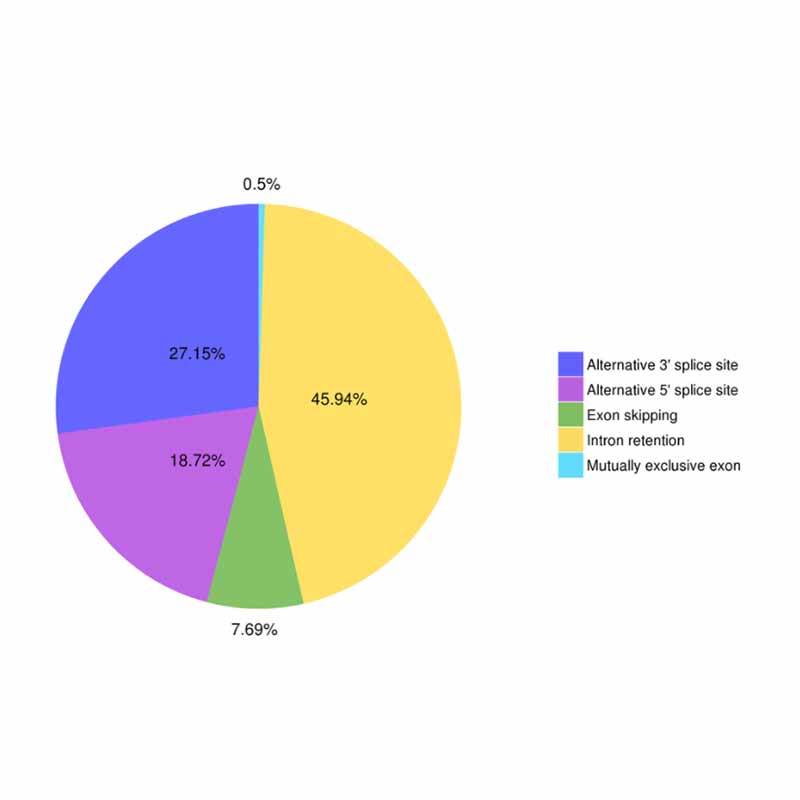
● Empalmament alternatiu
● Quantificació de l'expressió a nivell de gens i isoformes
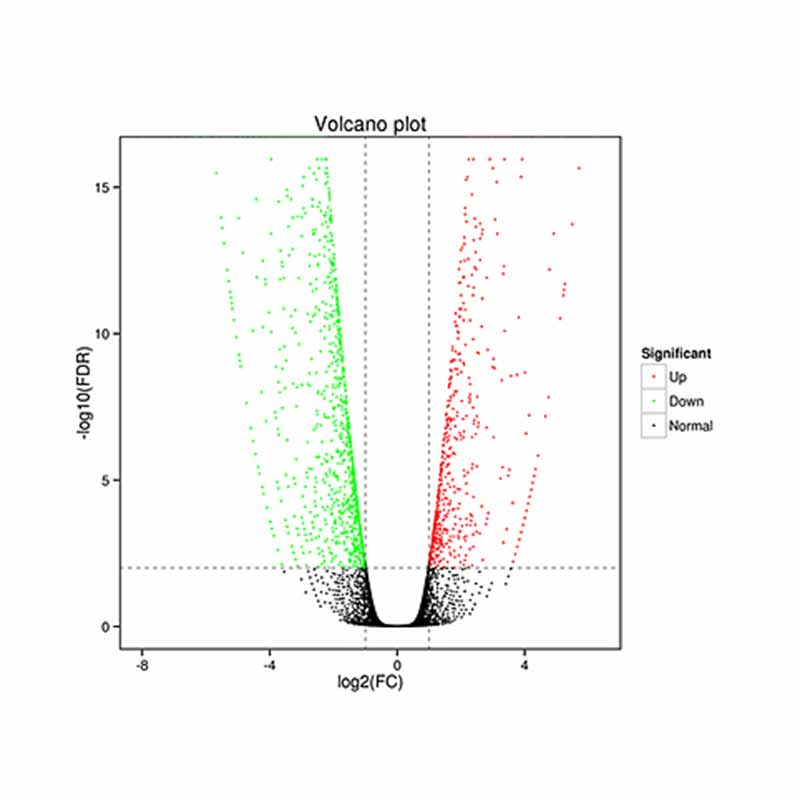
● Anàlisi d'expressió diferencial
● Annotació i enriquiment de funcions (DEG i DET)
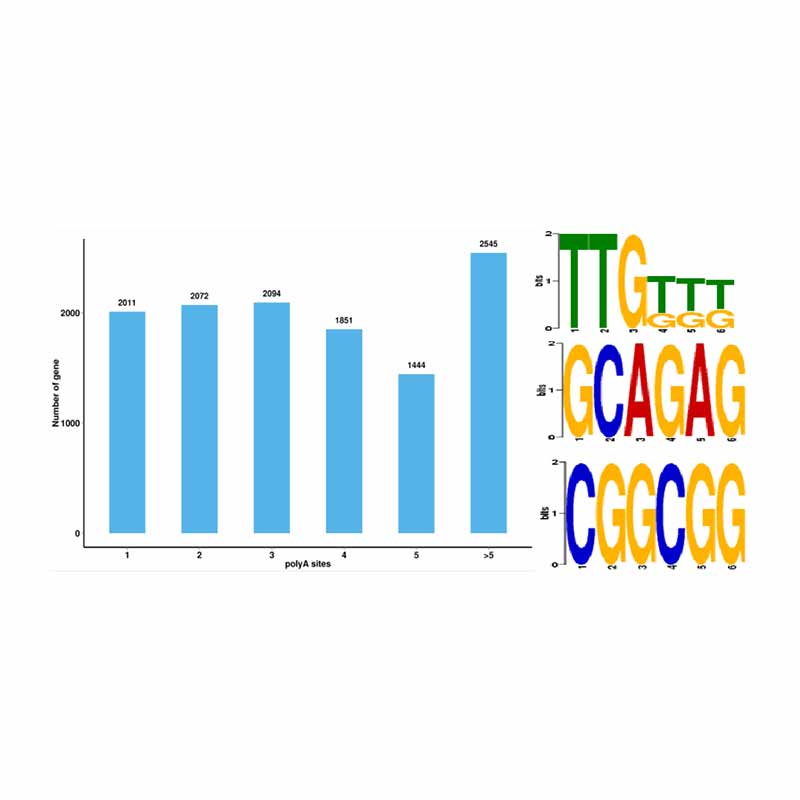
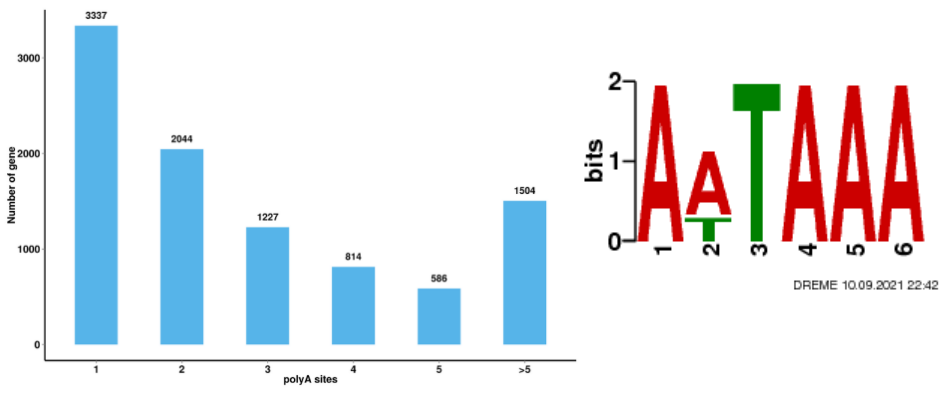
Anàlisi d'empalmament alternatiu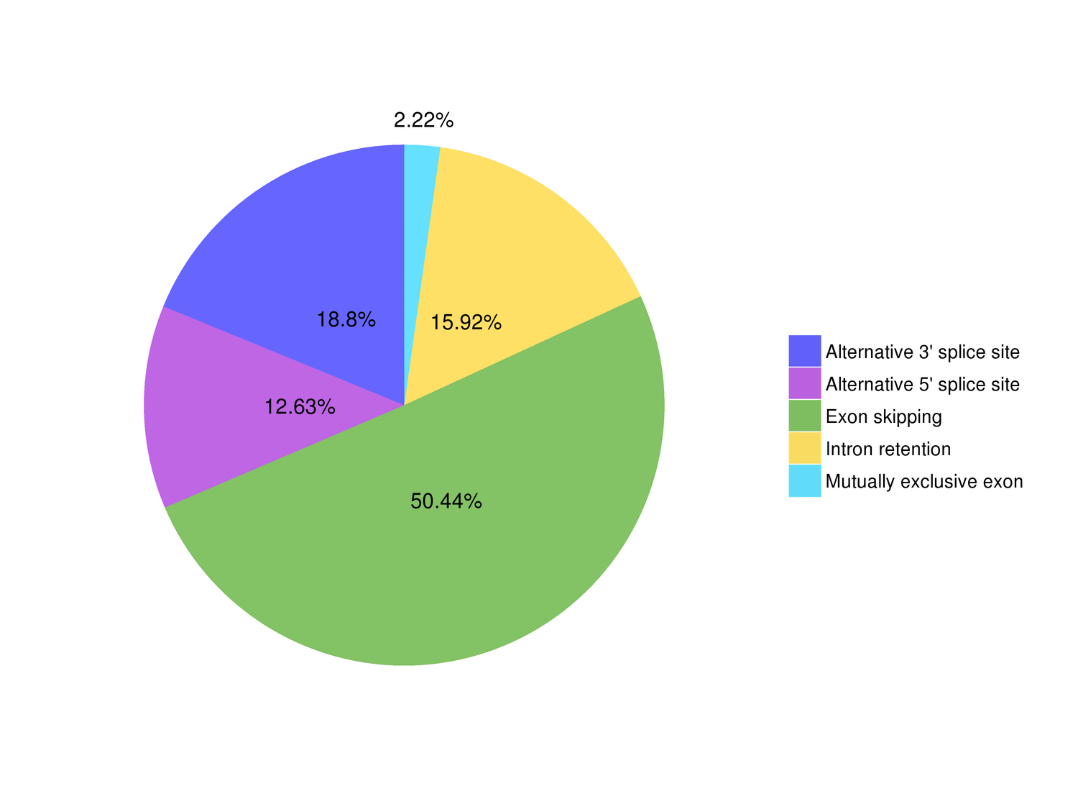 Anàlisi de poliadenilació alternativa (APA)
Anàlisi de poliadenilació alternativa (APA)
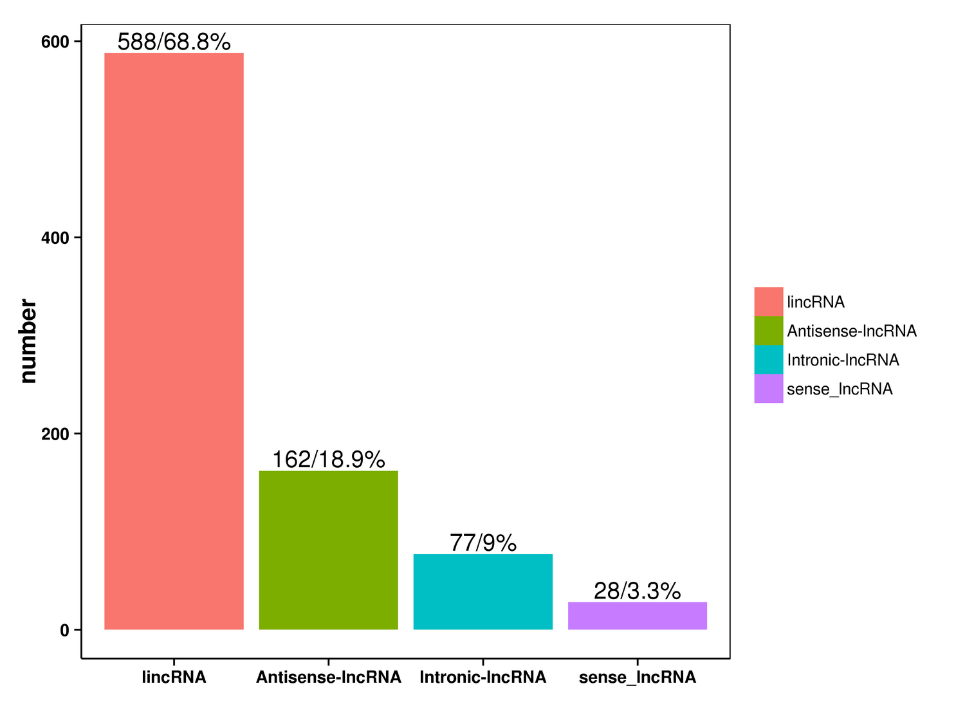
Predicció de lncRNA
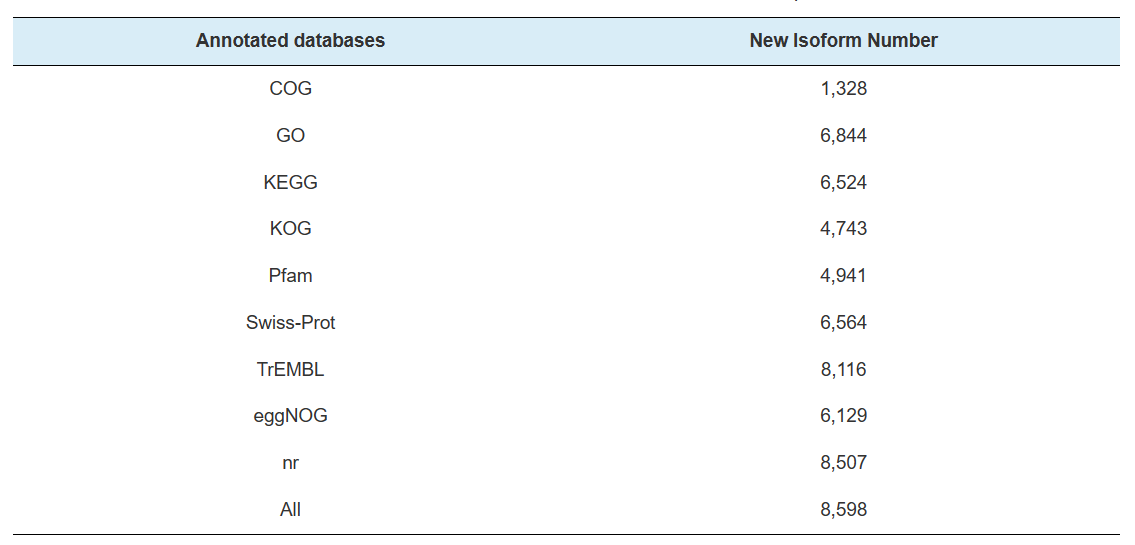
Anotació de gens nous
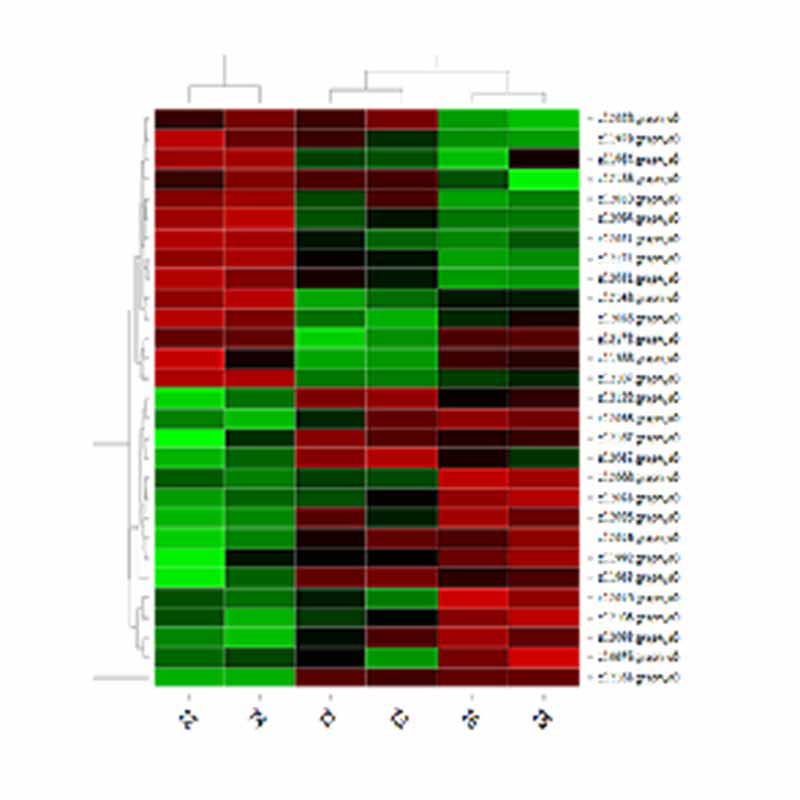
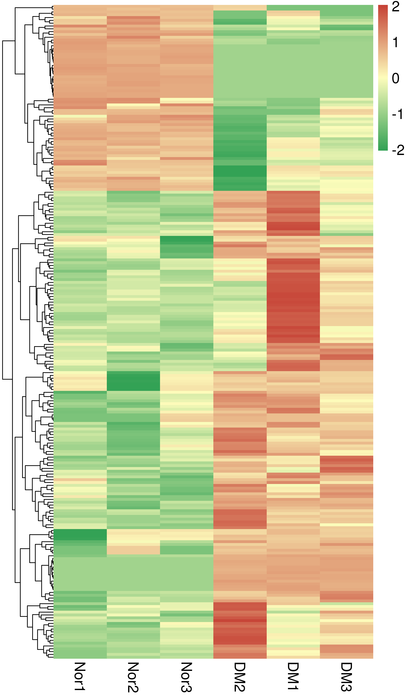
Agrupació de DET
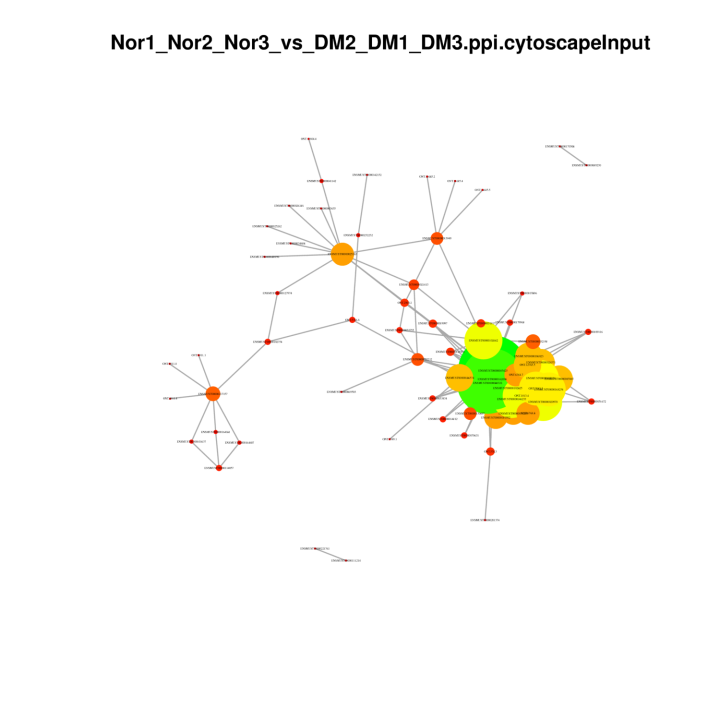
Xarxes proteïnes-proteïnes en DEG
Exploreu els avenços facilitats pels serveis de seqüenciació d'ARNm de llarga durada Nanopore de BMKGene mitjançant una col·lecció de publicacions curada.
Gong, B. et al. (2023) "Activació epigenètica i transcripcional de la quinasa secretora FAM20C com a oncogen en glioma", Journal of Genetics and Genomics, 50 (6), pàgs. 422–433. doi: 10.1016/J.JGG.2023.01.008.
Ell, Z. et al. (2023) "La seqüenciació de transcriptoma de longitud completa dels limfòcits responen a l'IFN-γ revela una resposta immune esbiaixada Th1 a la platassa (Paralichthys olivaceus)", Fish & Shellfish Immunology, 134, pàg. 108636. doi: 10.1016/J.FSI.2023.108636.
Ma, Y. et al. (2023) "Anàlisi comparada dels mètodes de seqüenciació d'ARN PacBio i ONT per a la identificació de verí de Nemopilema Nomurai", Genomics, 115 (6), pàg. 110709. doi: 10.1016/J.YGENO.2023.110709.
Yu, D. et al. (2023) "L'anàlisi nano-seq revela una tendència funcional diferent entre exosomes i microvesícules derivades de hUMSC", Stem Cell Research and Therapy, 14(1), pàgs. 1–13. doi: 10.1186/S13287-023-03491-5/TAULAS/6.