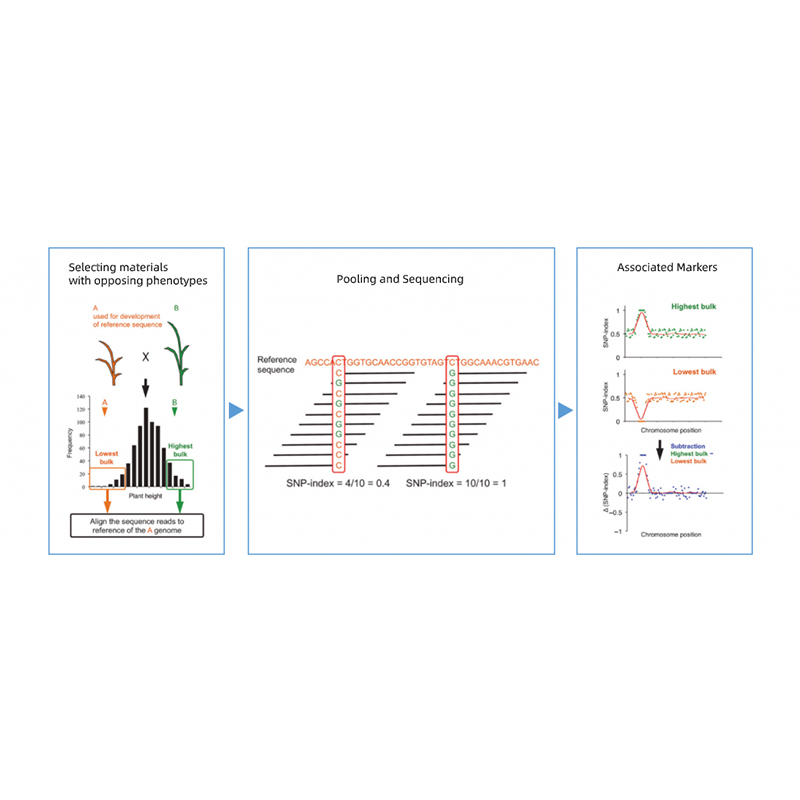
বাল্কড সেগ্রিগ্যান্ট বিশ্লেষণ
পরিষেবার সুবিধা

তাকাগি এট আল।, প্ল্যান্ট জার্নাল, 2013
● সঠিক স্থানীয়করণ: ব্যাকগ্রাউন্ডের শব্দ কমাতে 30+30 থেকে 200+200 ব্যক্তির সাথে বাল্ক মিশ্রিত করা; অ-সমার্থক মিউট্যাট্যান্ট-ভিত্তিক প্রার্থী অঞ্চলের পূর্বাভাস।
● ব্যাপক বিশ্লেষণ: NR, SwissProt, GO, KEGG, COG, KOG, ইত্যাদি সহ গভীরভাবে প্রার্থী জিন ফাংশন টীকা।
● দ্রুত টার্নরাউন্ড সময়: 45 কার্যদিবসের মধ্যে দ্রুত জিন স্থানীয়করণ।
● ব্যাপক অভিজ্ঞতা: শস্য, জলজ পণ্য, বন, ফুল, ফল ইত্যাদির মতো বৈচিত্র্যময় প্রজাতিকে কভার করে হাজার হাজার বৈশিষ্ট্য স্থানীয়করণে বিএমকে অবদান রেখেছে।
পরিষেবা নির্দিষ্টকরণ
জনসংখ্যা:
বিরোধী ফেনোটাইপগুলির সাথে পিতামাতার বংশধরদের আলাদা করা।
যেমন F2 বংশধর, ব্যাকক্রসিং (BC), রিকম্বিন্যান্ট ইনব্রিড লাইন (RIL)
মেশানো পুল
গুণগত বৈশিষ্ট্যের জন্য: 30 থেকে 50 জন (সর্বনিম্ন 20)/বাল্ক
পরিমাণগত ট্রাটিসের জন্য: সমগ্র জনসংখ্যার মধ্যে চরম ফেনোটাইপ সহ শীর্ষ 5% থেকে 10% ব্যক্তি (ন্যূনতম 30+30)।
প্রস্তাবিত সিকোয়েন্সিং গভীরতা
কমপক্ষে 20X/পিতামাতা এবং 1X/সন্তান ব্যক্তি (যেমন 30+30 পৃথক পৃথক সন্তানের মিক্সিং পুলের জন্য, সিকোয়েন্সিং গভীরতা প্রতি বাল্ক 30X হবে)
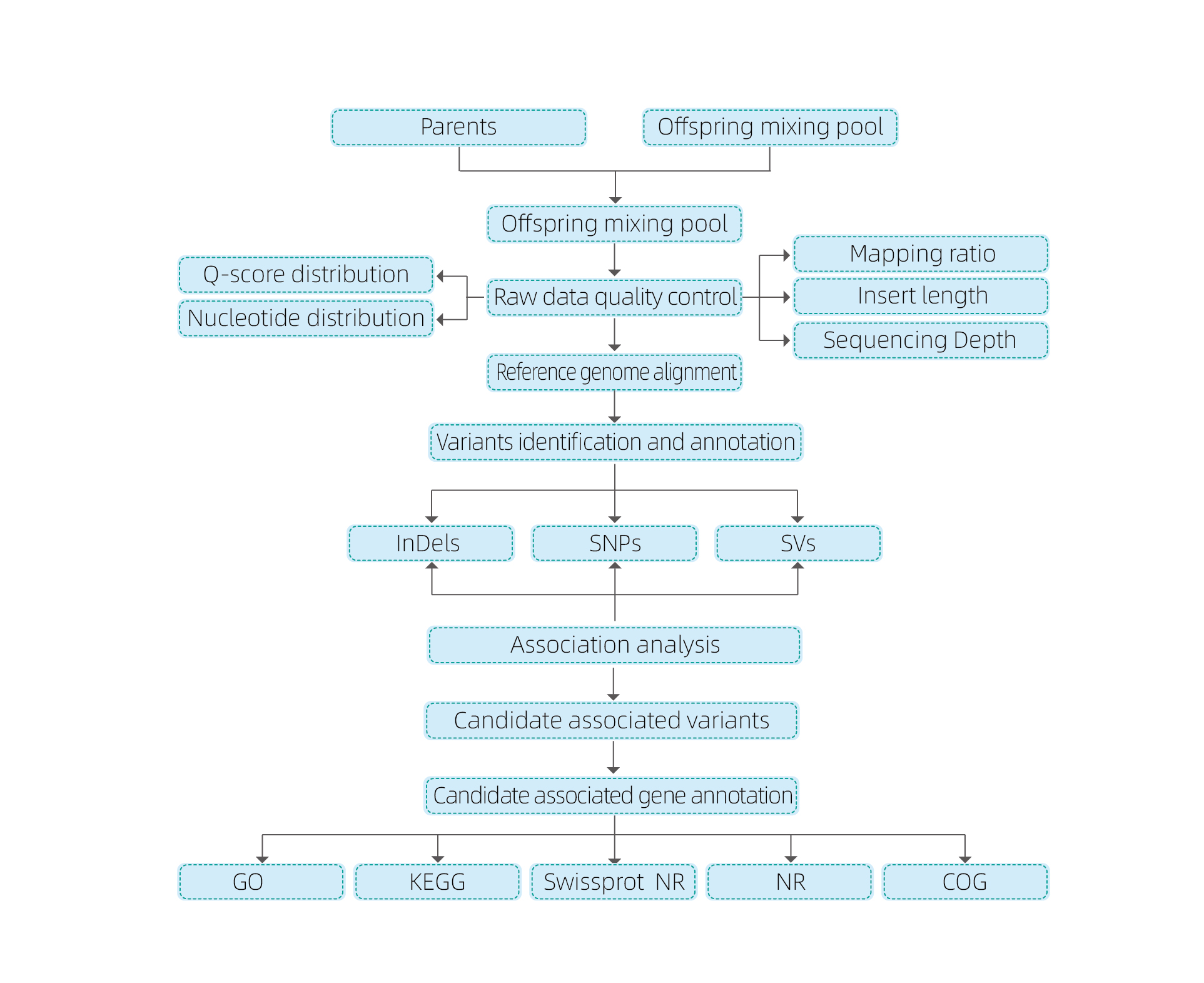
বায়োইনফরমেটিক্স বিশ্লেষণ
● সম্পূর্ণ জিনোম রিকোয়েন্সিং
● ডেটা প্রক্রিয়াকরণ
● SNP/Indel কলিং
● প্রার্থীর অঞ্চল স্ক্রীনিং
● প্রার্থীর জিন ফাংশন টীকা

নমুনা প্রয়োজনীয়তা এবং ডেলিভারি
নমুনা প্রয়োজনীয়তা:
নিউক্লিওটাইডস:
| জিডিএনএ নমুনা | টিস্যু নমুনা |
| ঘনত্ব: ≥30 ng/μl | গাছপালা: 1-2 গ্রাম |
| পরিমাণ: ≥2 μg (ভলিউম ≥15 μl) | প্রাণী: 0.5-1 গ্রাম |
| বিশুদ্ধতা: OD260/280= 1.6-2.5 | সম্পূর্ণ রক্ত: 1.5 মিলি |
সার্ভিস ওয়ার্ক ফ্লো

পরীক্ষামূলক নকশা

নমুনা বিতরণ

আরএনএ নিষ্কাশন

গ্রন্থাগার নির্মাণ

সিকোয়েন্সিং

ডেটা বিশ্লেষণ

বিক্রয়োত্তর সেবা
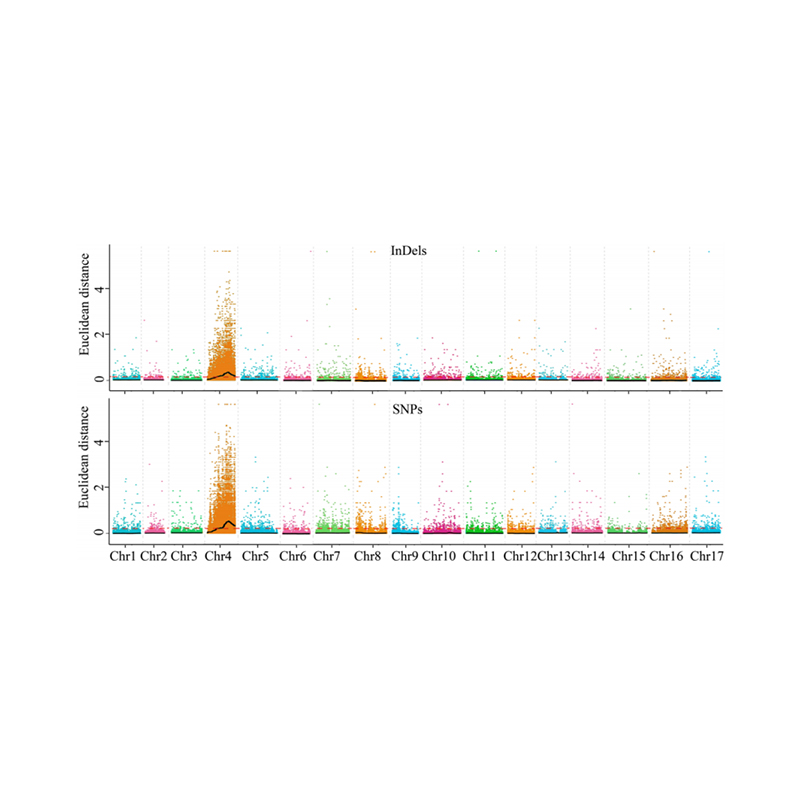
1. প্রার্থী অঞ্চল সনাক্ত করতে ইউক্লিডীয় দূরত্ব (ED) এর উপর অ্যাসোসিয়েশন বিশ্লেষণ বেস। নিম্নলিখিত চিত্রে
X-অক্ষ: ক্রোমোজোম সংখ্যা; প্রতিটি বিন্দু একটি SNP এর একটি ED মান উপস্থাপন করে। কালো লাইন লাগানো ED মানের সাথে মিলে যায়। একটি উচ্চতর ED মান সাইট এবং ফেনোটাইপের মধ্যে আরও উল্লেখযোগ্য সম্পর্ক নির্দেশ করে। লাল ড্যাশ লাইন উল্লেখযোগ্য সংযোগের প্রান্তিক প্রতিনিধিত্ব করে।

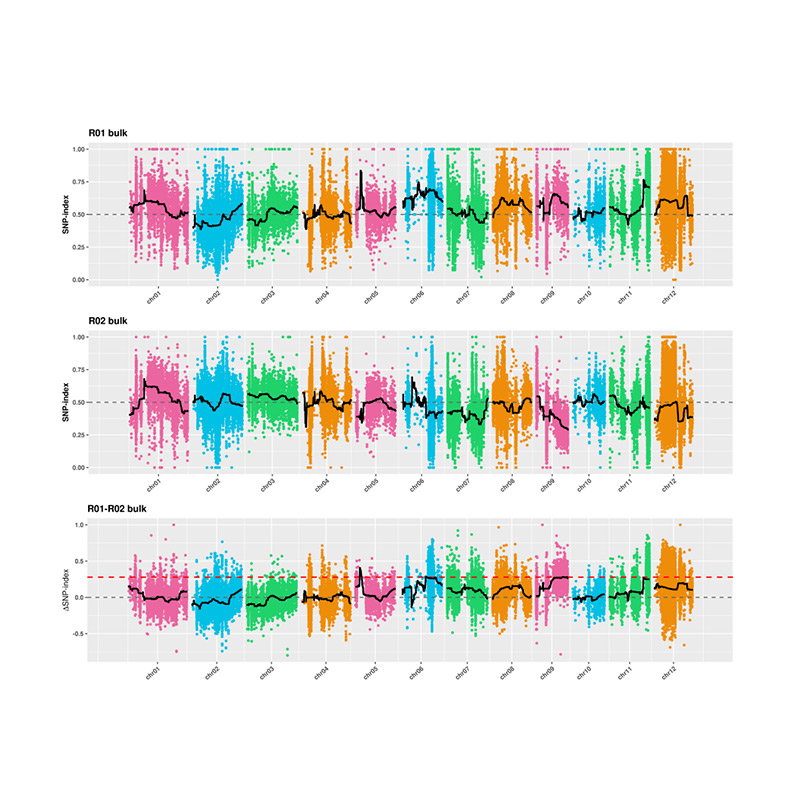
2. কোন SNP-সূচক ভিত্তিক অ্যাসোসিয়েশন বিশ্লেষণ
X-অক্ষ: ক্রোমোজোম সংখ্যা; প্রতিটি বিন্দু SNP-সূচক মান উপস্থাপন করে। কালো রেখাটি ফিট করা SNP-সূচক মান বোঝায়। মান যত বড়, সমিতি তত বেশি তাৎপর্যপূর্ণ।

বিএমকে কেস
প্রধান-প্রভাব পরিমাণগত বৈশিষ্ট্য লোকাস Fnl7.1 শসায় ফলের ঘাড় দৈর্ঘ্যের সাথে যুক্ত একটি দেরী ভ্রূণজনিত প্রচুর প্রোটিন এনকোড করে
প্রকাশিত: উদ্ভিদ বায়োটেকনোলজি জার্নাল, 2020
সিকোয়েন্সিং কৌশল:
পিতামাতা (Jin5-508, YN): 34× এবং 20× এর জন্য সম্পূর্ণ জিনোম রেকোয়েন্সিং।
ডিএনএ পুল (50 লম্বা গলা এবং 50 ছোট গলা): 61× এবং 52× এর জন্য রেসকুয়েন্সিং
মূল ফলাফল
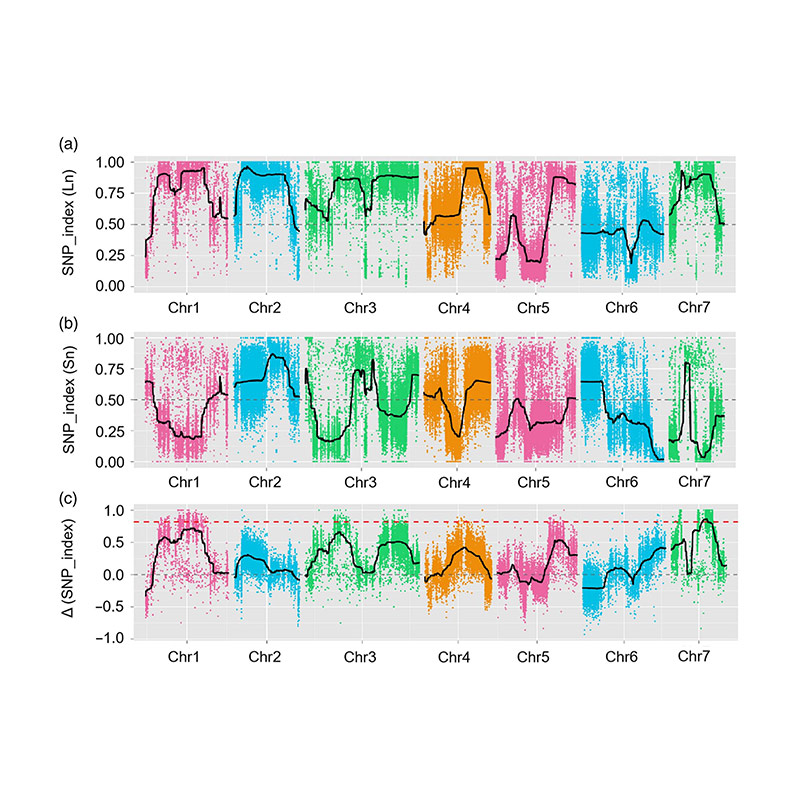
এই সমীক্ষায়, লং-নেক শসার লাইন জিন5-508 এবং ছোট-ঘাড়ের ওয়াইএন অতিক্রম করার মাধ্যমে পৃথকীকরণ জনসংখ্যা (F2 এবং F2:3) তৈরি করা হয়েছিল। দুটি ডিএনএ পুল 50 জন চরম লম্বা গলা ব্যক্তি এবং 50 জন চরম ছোট গলা ব্যক্তি দ্বারা নির্মিত হয়েছিল। BSA বিশ্লেষণ এবং ঐতিহ্যগত QTL ম্যাপিং দ্বারা Chr07-এ প্রধান-প্রভাব QTL চিহ্নিত করা হয়েছিল। সূক্ষ্ম-ম্যাপিং, জিন এক্সপ্রেশন কোয়ান্টিফিকেশন এবং ট্রান্সজেনিক পরীক্ষা-নিরীক্ষার মাধ্যমে প্রার্থী অঞ্চলটিকে আরও সংকীর্ণ করা হয়েছিল, যা ঘাড়-দৈর্ঘ্য নিয়ন্ত্রণে মূল জিন প্রকাশ করে, CsFnl7.1। উপরন্তু, CsFnl7.1 প্রবর্তক অঞ্চলে পলিমরফিজম সংশ্লিষ্ট অভিব্যক্তির সাথে যুক্ত পাওয়া গেছে। আরও ফিলোজেনেটিক বিশ্লেষণে পরামর্শ দেওয়া হয়েছে যে Fnl7.1 লোকাস ভারত থেকে উদ্ভূত হওয়ার সম্ভাবনা রয়েছে।
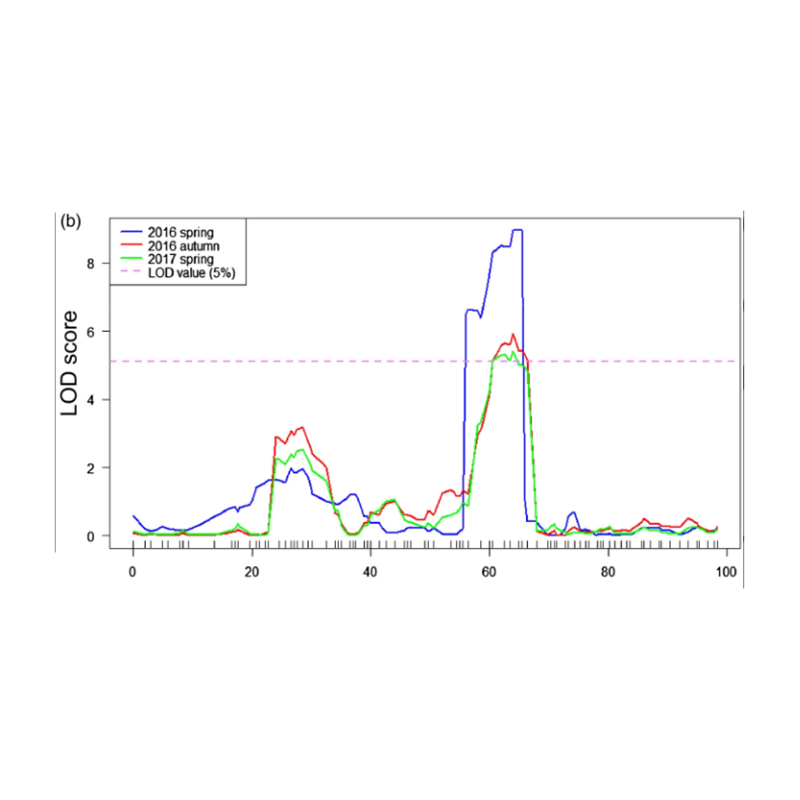
 বিএসএ বিশ্লেষণে QTL-ম্যাপিং শসার ঘাড়ের দৈর্ঘ্যের সাথে যুক্ত প্রার্থী অঞ্চল সনাক্ত করতে |  শসার ঘাড়-দৈর্ঘ্যের QTL-এর LOD প্রোফাইল Chr07-এ শনাক্ত করা হয়েছে |
Xu, X. , et al. "প্রধান-প্রভাব পরিমাণগত বৈশিষ্ট্য লোকাস Fnl7.1 শসার ফলের ঘাড়ের দৈর্ঘ্যের সাথে যুক্ত একটি দেরী ভ্রূণজনিত প্রচুর প্রোটিন এনকোড করে।" প্ল্যান্ট বায়োটেকনোলজি জার্নাল 18.7(2020)।