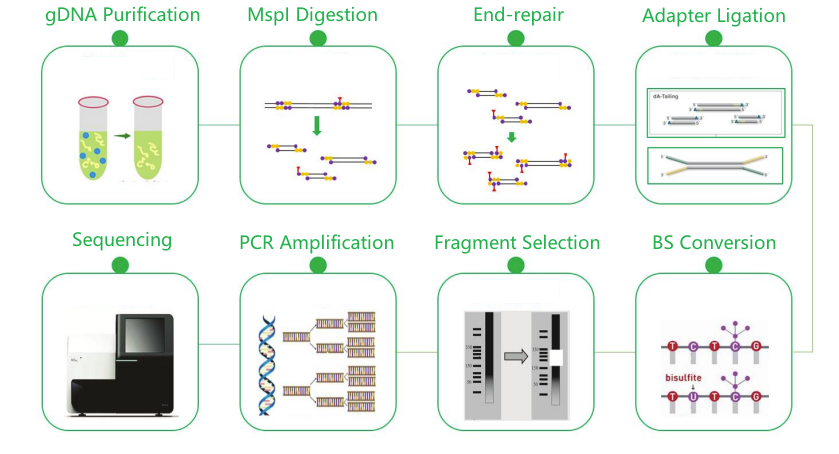
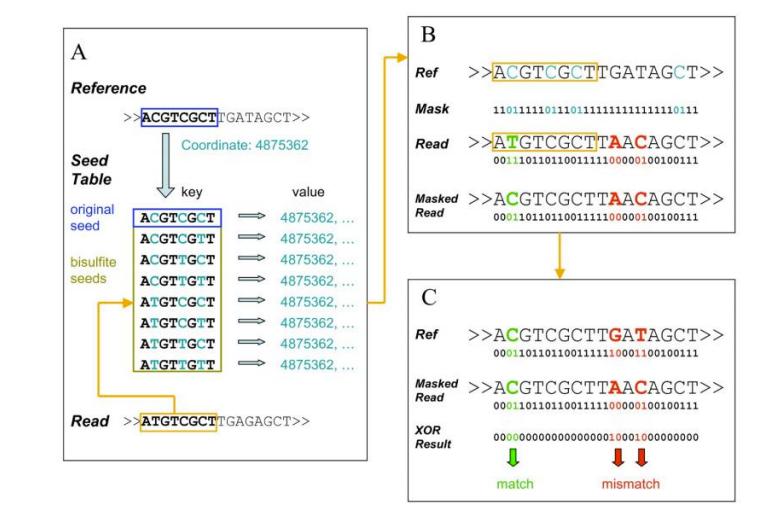
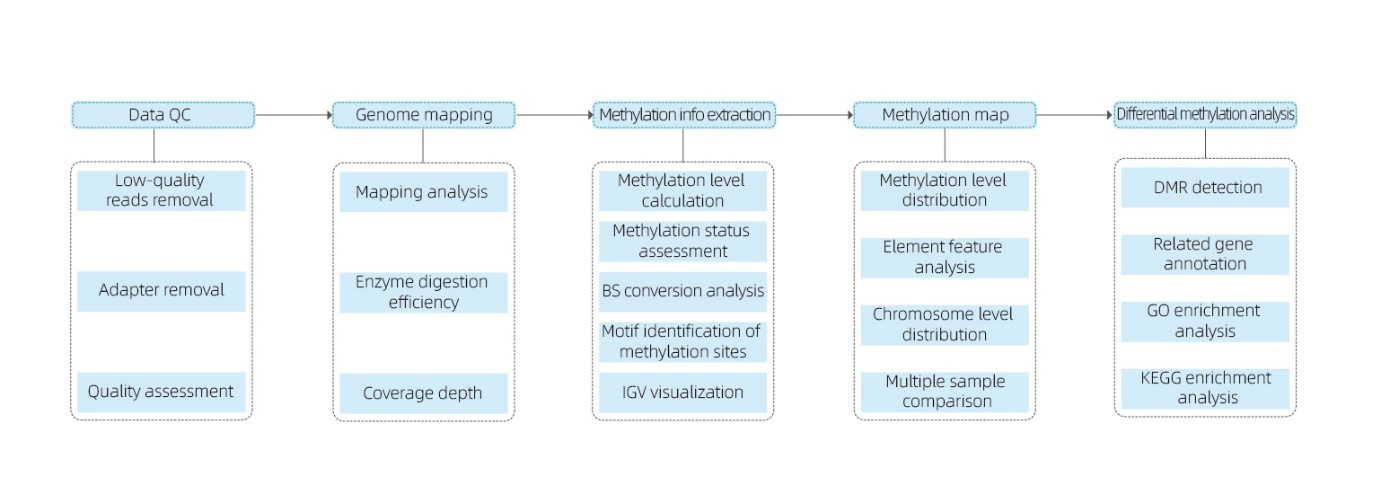
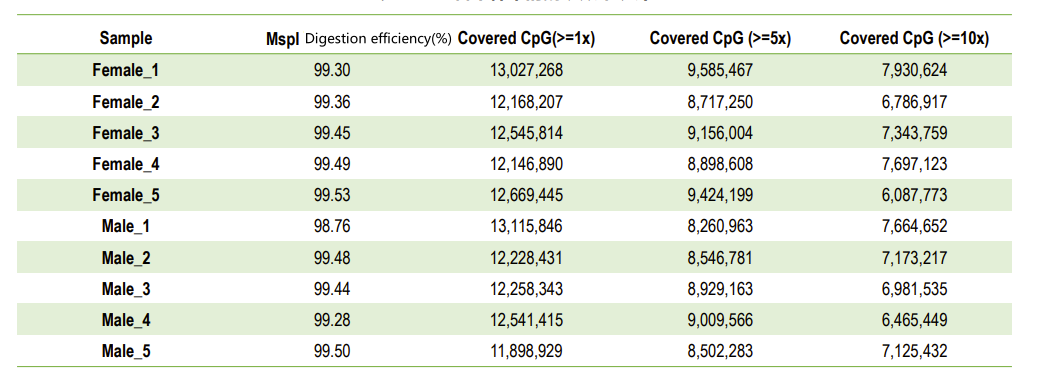
Бисулфитно секвениране с намалено представяне (RRBS)
Характеристики на услугата
● Изисква референтен геном.
● Ламбда ДНК се използва за наблюдение на ефективността на преобразуване на бисулфит.
● Ефективността на храносмилането на MspI също се наблюдава.
● Двойно ензимно смилане за растителни проби.
● Секвениране на Illumina NovaSeq.
Предимства на услугата
●Рентабилна и ефикасна алтернатива на WGBS: позволява извършването на анализа на по-ниска цена и с по-ниски изисквания за проби.
●Пълна платформа:предоставят отлично обслужване на едно гише от обработка на проби, изграждане на библиотека и секвениране до биоинформатичен анализ.
●Обширен експертен опит: с проекти за секвениране на RRBS, успешно завършени в разнообразна гама от видове, BMKGENE носи над десетилетие опит, висококвалифициран екип за анализ, цялостно съдържание и отлична поддръжка след продажбата.
Сервизни спецификации
| Библиотека | Стратегия за последователност | Препоръчителен изход на данни | Контрол на качеството |
| MspI разградена и третирана с бисулфит библиотека | Ilumina PE150 | 8 Gb | Q30 ≥ 85% Конверсия на бисулфит > 99% MspI ефективност на рязане > 95% |
Примерни изисквания
| Концентрация (ng/µL) | Общо количество (µg) |
| |
| Геномна ДНК | ≥ 30 | ≥ 1 | Ограничено разграждане или замърсяване |
Работен поток на услугата

Доставка на мостри

Изграждане на библиотека

Секвениране

Анализ на данни
Следпродажбени услуги
Включва следния анализ:
● Контрол на качеството на суровото секвениране;
● Картиране към референтен геном;
● Откриване на 5mC метилирани бази и идентификация на мотиви;
● Анализ на разпределението на метилирането и сравнение на пробите;
● Анализ на диференциално метилирани региони (DMRs);
● Функционална анотация на гени, свързани с DMR.
Контрол на качеството: ефективност на храносмилането (при картографиране на генома)
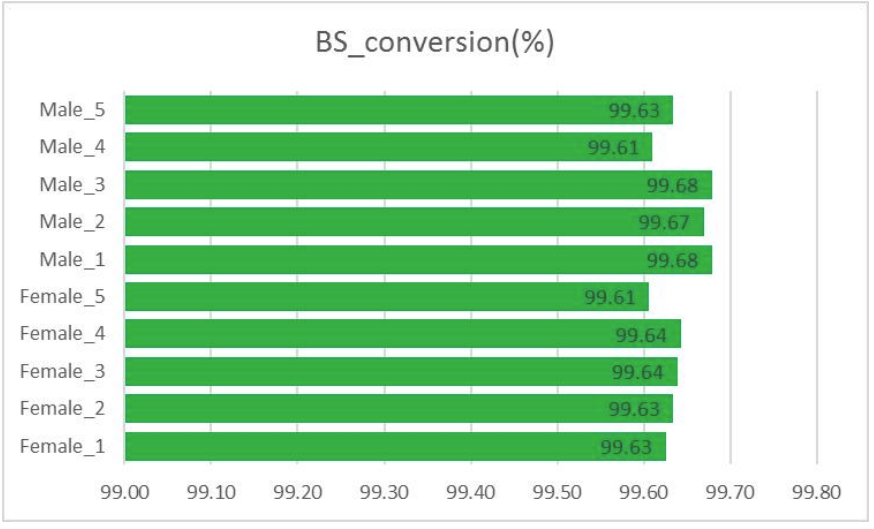
Контрол на качеството: преобразуване на бисулфит (при извличане на информация за метилиране)
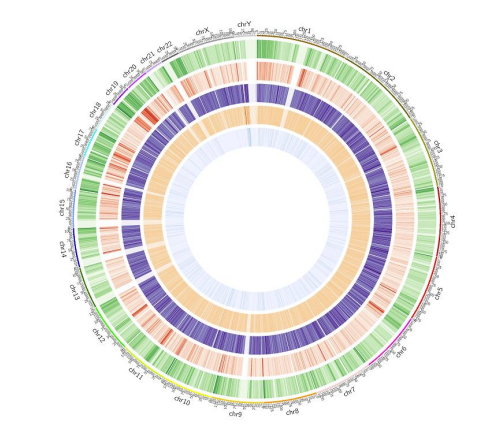
Карта на метилиране: 5mC метилиране в целия геном
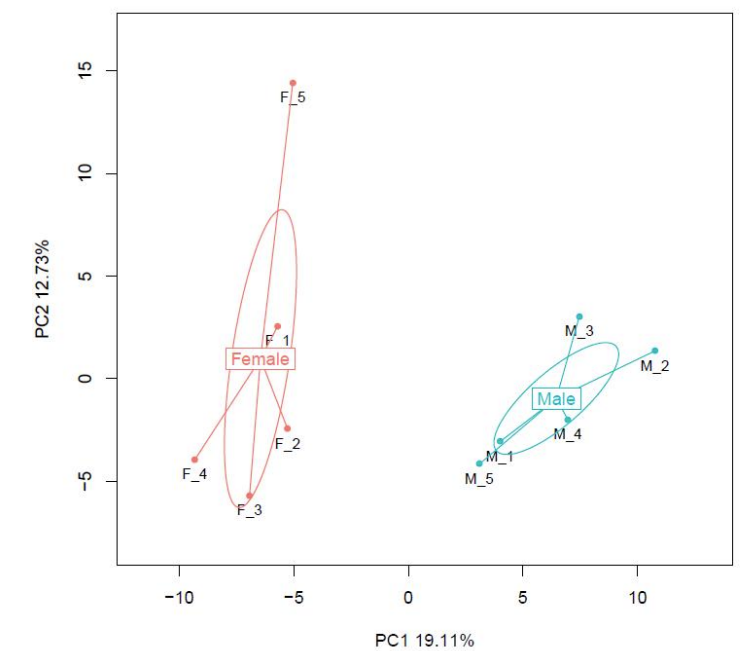
Примерно сравнение: Анализ на основните компоненти
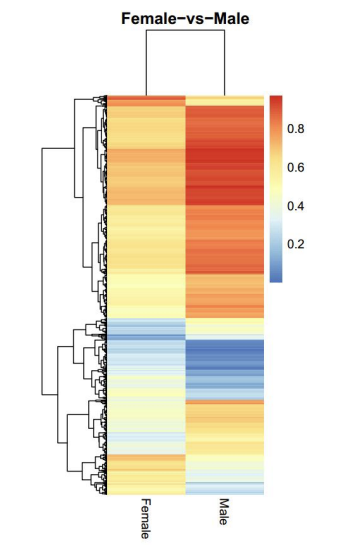
Анализ на диференциално метилирани региони (DMRs): топлинна карта
Разгледайте изследователския напредък, улеснен от услугите на BMKGene за бисулфитно секвениране на целия геном чрез подбрана колекция от публикации.
Li, Z. et al. (2022) „Препрограмиране с висока точност в подобни на Лайдиг клетки чрез активиране на CRISPR и паракринни фактори“,PNAS Nexus, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. et al. (2023) „Анализ на метилиране на ДНК в целия геном на състава на тялото при китайски монозиготни близнаци“,Европейски вестник за клинични изследвания, 53(11), стр. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) „ДНК метилиране и съотношение талия към ханш: проучване за асоцииране в епигенома при китайски монозиготни близнаци“,Журнал за ендокринологични изследвания, 45 (12), стр. 2365–2376. doi: 10.1007/S40618-022-01878-4.