DNBSEQ предварително създадени библиотеки
Характеристики
●платформа:MGI-DNBSEQ-T7
●Режими на последователност:PE150
●Прехвърляне на библиотеки на Illumina къмMGI:позволяващи последователност на големи обеми данни на ниска цена.
●Контрол на качеството на библиотеките преди секвениране.
●QC и доставка на данни за последователност:доставка на QC отчет и необработени данни във формат fastq след демултиплексиране и филтриране на Q30 четения.
Предимства
●Гъвкавост на услугите за секвениране:клиентът може да избере да подреди по лента или количество данни.
●Висок изход на данни:1500 Gb/лента
●Доставка на QC доклад за секвениране:с показатели за качество, точност на данните и цялостно изпълнение на проекта за секвениране.
●Зрял процес на секвениране:с кратко време за изпълнение.
●Строг контрол на качеството: прилагаме строги изисквания за контрол на качеството, за да гарантираме предоставянето на постоянно висококачествени резултати.
Примерни изисквания
| Количество данни (X) | Концентрация (qPCR/nM) | Обем | |
| Частична лента
| X ≤ 10 Gb | ≥ 1nM | ≥ 25 μl |
| 10 Gb < X ≤ 50 Gb | ≥ 2 nM | ≥ 25 μl | |
| 50 Gb < X ≤ 100 Gb | ≥ 3 nM | ≥ 25 μl | |
| X > 100 Gb | ≥ 4 nM | ||
| Единична лента | На Лейн | ≥ 1,5 nM / Библиотечен пул | ≥ 25 μl / Библиотечен басейн |
В допълнение към концентрацията и общото количество е необходим и подходящ пиков модел.
Забележка: Последователността на лентите на библиотеки с ниско разнообразие изисква PhiX spike-in, за да осигури стабилно базово повикване.
Препоръчваме да изпратите предварително обединени библиотеки като проби. Ако искате BMKGENE да извърши обединяване на библиотеки, моля, вижте
библиотечни изисквания за частична последователност на лентите.
Размер на библиотеката (карта на пика)
Основният пик трябва да бъде в рамките на 300-450 bp.
Библиотеките трябва да имат единичен основен пик, без замърсяване на адаптера и без димери на праймера.
Работен процес на услугата




Доклад за QC на библиотеката
Предоставя се отчет за качеството на библиотеката преди секвениране, оценка на количеството на библиотеката и фрагментиране.
QC доклад за секвениране
Таблица 1. Статистически данни за секвениране на данни.
| Примерен идентификатор | BMKID | Сурови четения | Сурови данни (bp) | Чисто четене (%) | Q20 (%) | Q30(%) | GC (%) |
| C_01 | BMK_01 | 22,870,120 | 6 861 036 000 | 96,48 | 99.14 | 94,85 | 36,67 |
| C_02 | BMK_02 | 14,717,867 | 4,415,360,100 | 96,00 | 98,95 | 93,89 | 37.08 |
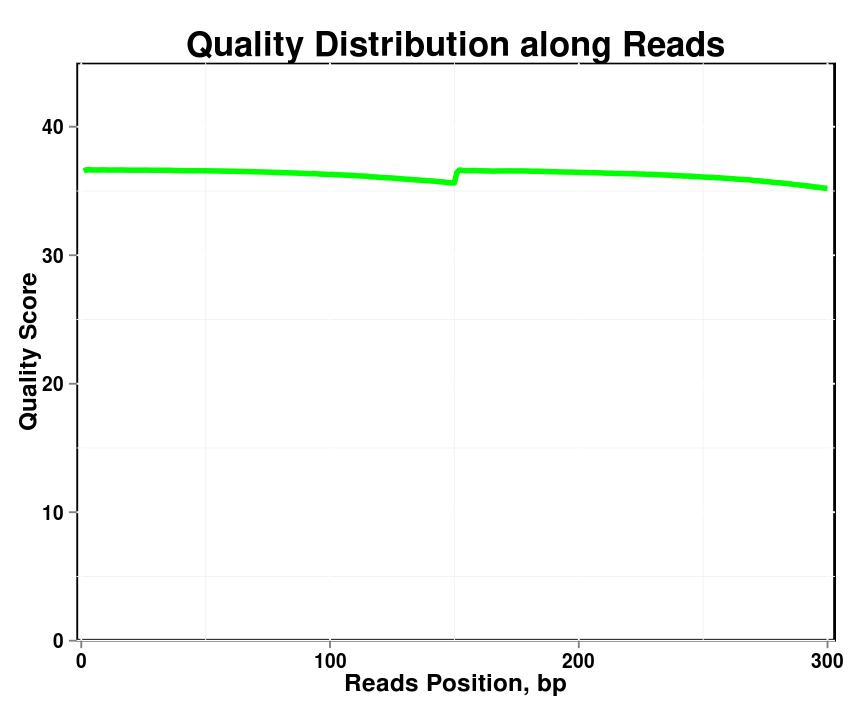
Фигура 1. Разпределение на качеството по отчитанията във всяка проба
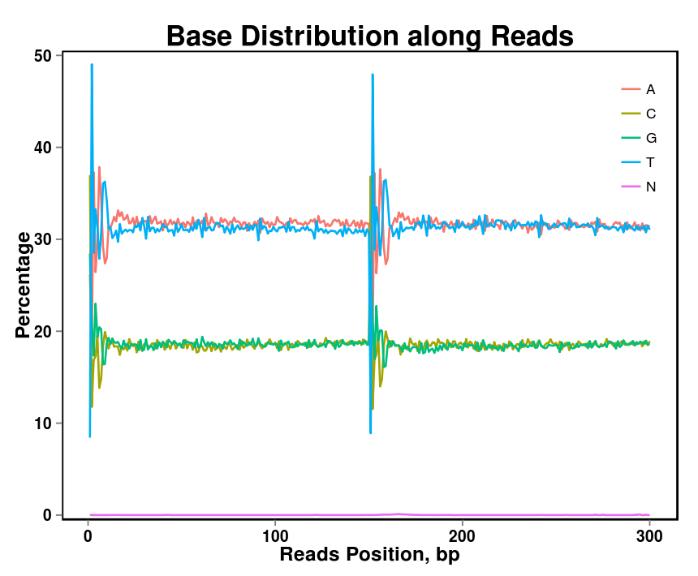
Фигура 2. Базово разпределение на съдържанието
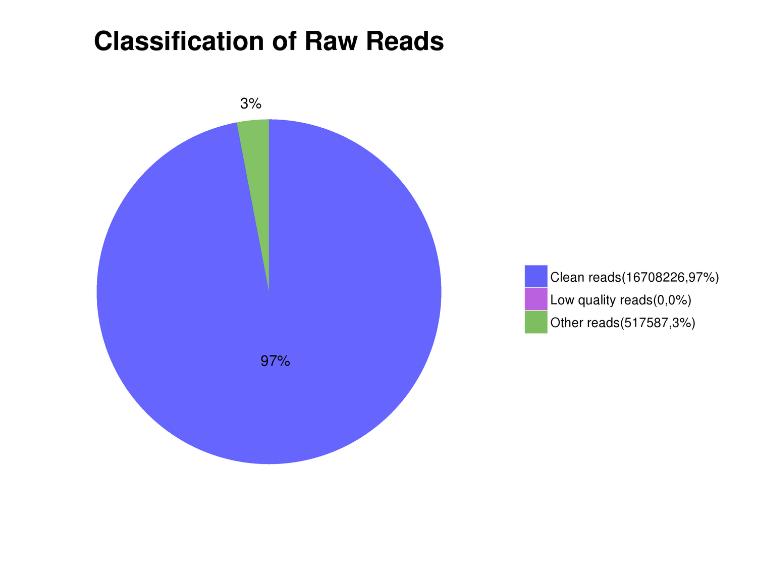
Фигура 3. Разпределение на прочетеното съдържание в данните за последователност