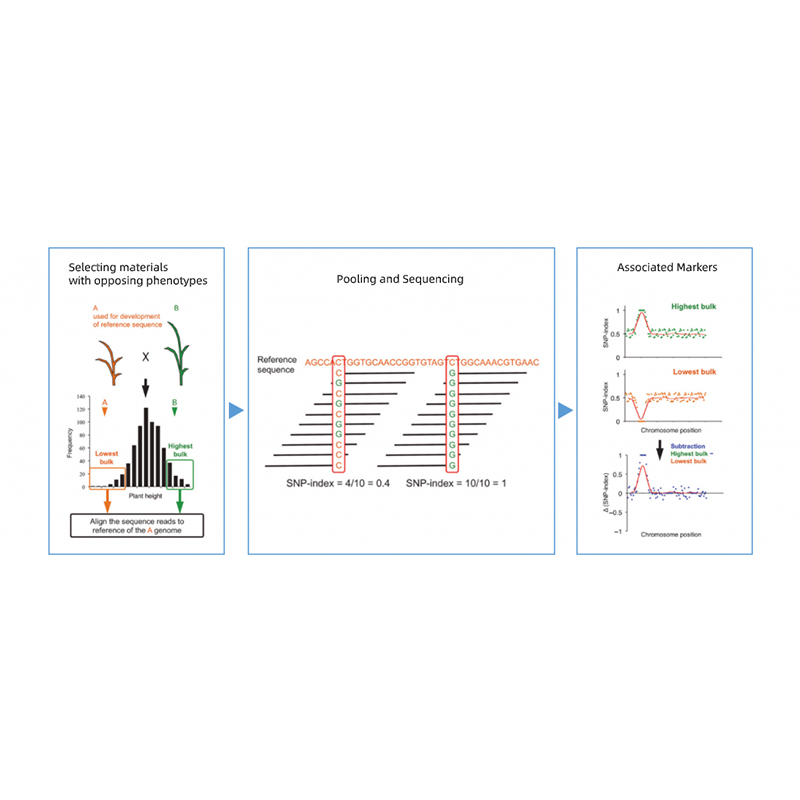
Натрупан сегрегант анализ
Предимства на услугата

Takagi et al., The Plant Journal, 2013
● Точна локализация: смесване на насипни с 30+30 до 200+200 индивида, за да се сведе до минимум фоновия шум; Несинонимично прогнозиране на региона, базиран на мутанти.
● Изчерпателен анализ: Анотация на функцията за задълбочена кандидат-ген, включително NR, Swissprot, GO, KEGG, COG, KOG и др.
● По -бързо време за обороти: Бърза локализация на ген в рамките на 45 работни дни.
● Обширен опит: BMK е допринесъл в хиляди локализация на черти, обхващайки различни видове като култури, водни продукти, гора, цветя, плодове и др.
Спецификации на услугата
Население:
Разделяне на потомството на родители с противоположни фенотипове.
напр. F2 потомство, обратно кръстосване (BC), рекомбинантна инбредна линия (RIL)
Смесване на басейн
За качествени черти: 30 до 50 индивида (минимум 20)/насипно състояние
За количествени трати: Най -добрите 5% до 10% индивиди с или екстремни фенотипове в цялата популация (минимум 30+30).
Препоръчителна дълбочина на секвениране
Най -малко 20x/родител и 1x/Offspring индивид (напр. За смесване на басейн от 30+30 индивидуални, дълбочината на секвениране ще бъде 30 пъти на насипно състояние)
Анализи на биоинформатика
● Резюме на целия геном
● Обработка на данни
● SNP/Indel Обаждане
● Проверка на кандидата за регион
● Анотация на кандидат -генната функция

Примерни изисквания и доставка
Изисквания за извадка:
Нуклеотиди:
| GDNA проба | Тъканна проба |
| Концентрация: ≥30 ng/μl | Растения: 1-2 g |
| Сума: ≥2 μg (Volumn ≥15 μl) | Животни: 0,5-1 g |
| Чистота: OD260/280 = 1.6-2.5 | Пълна кръв: 1,5 ml |
Работен поток на сервиз

Дизайн на експеримента

Доставка на проба

Екстракция на РНК

Библиотечно строителство

Секвениране

Анализ на данните

Услуги след продажба
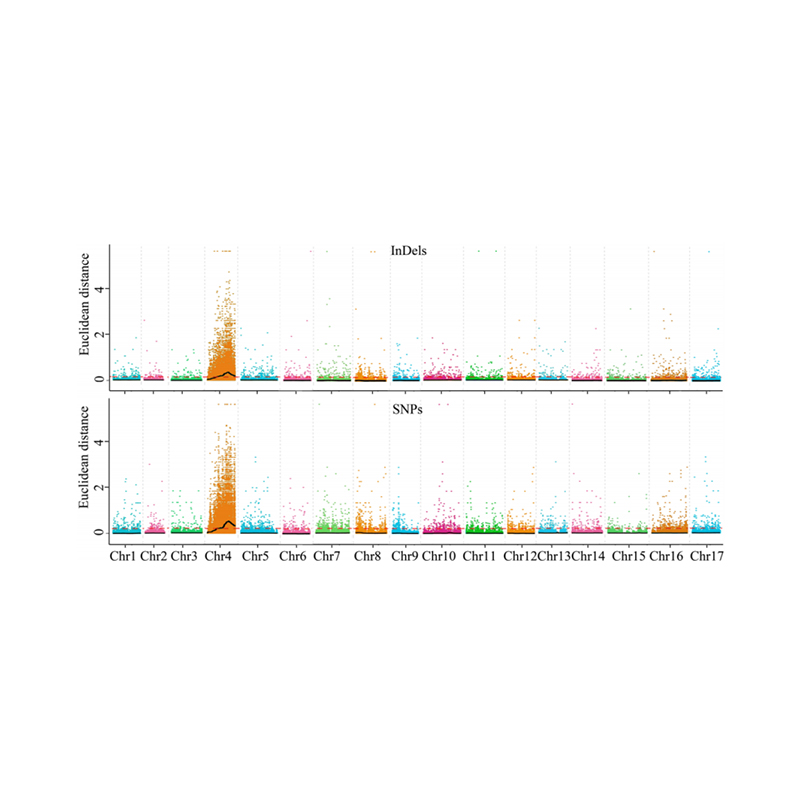
1. Асоциация Анализ на база на евклидовото разстояние (ED) за идентифициране на кандидат -регион. В следната фигура
X-ос: номер на хромозома; Всяка точка представлява ED стойност на SNP. Черната линия съответства на монтираната ED стойност. По -високата стойност на ЕД показва по -значителна връзка между сайта и фенотипа. Red Dash Line представлява праг на значителна асоциация.

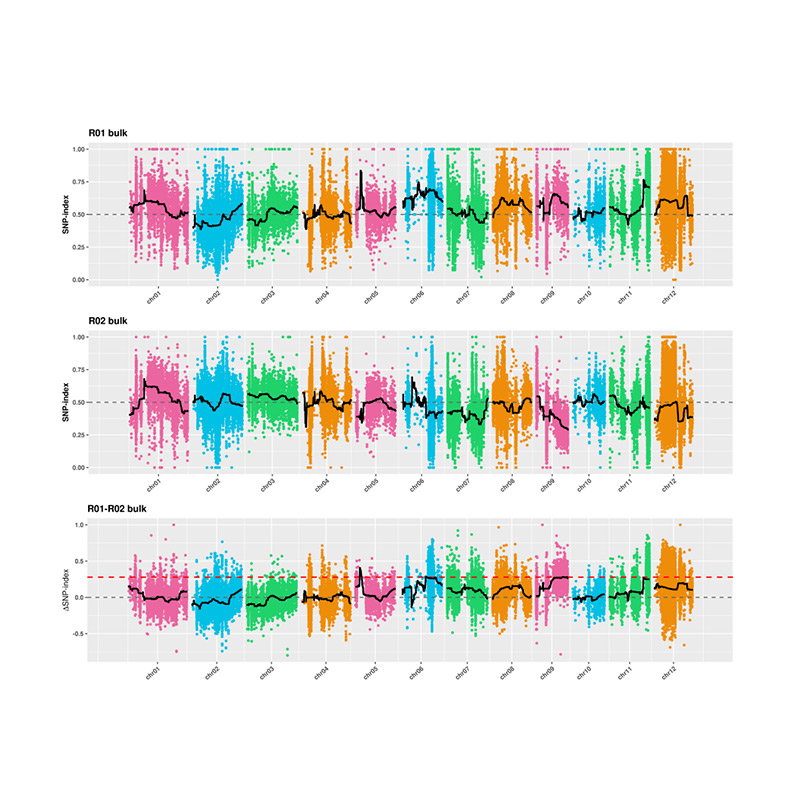
2. Асоциация Анализ, базиран на SNP-индекс
X-ос: номер на хромозома; Всяка точка представлява стойност на SNP-индекс. Черната линия означава монтирана стойност на SNP-индекс. Колкото по -голяма е стойността, толкова по -значима е асоциацията.

Случай на BMK
Количественият локус на количествената черта FNL7.1 кодира късен ембриогенеза изобилен протеин, свързан с дължината на плодовата шия в краставицата
Публикувано: Списание за биотехнология на растенията, 2020
Стратегия за секвениране:
Родители (JIN5-508, YN): Целият геном се радва за 34 × и 20 ×.
ДНК басейни (50 дълги деколтета и 50 къси деколте): преразглеждане за 61 × и 52 ×
Основни резултати
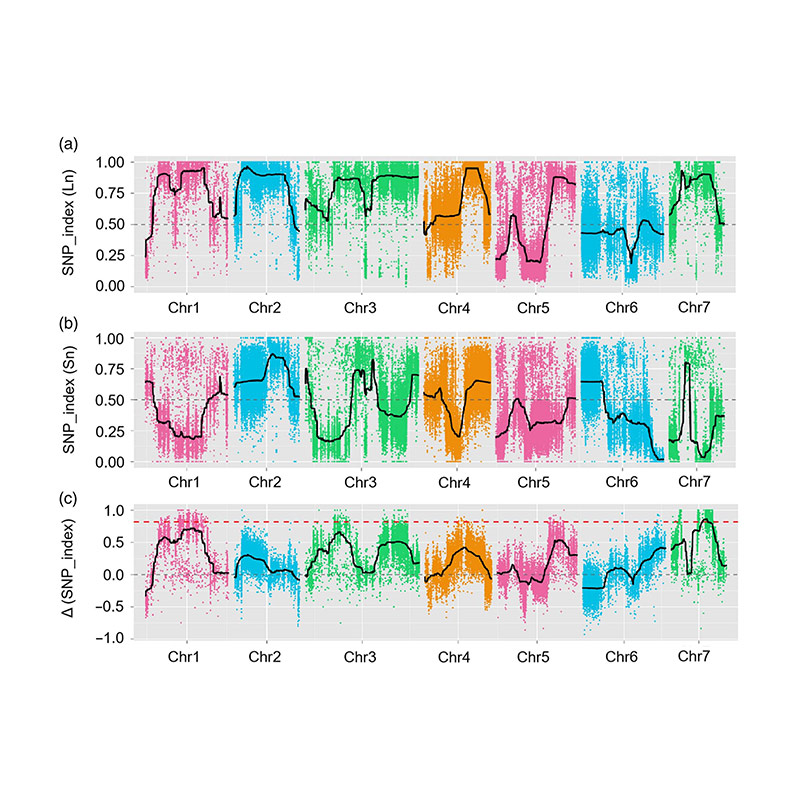
В това проучване сегрегиращата популация (F2 и F2: 3) се генерира чрез пресичане на линията на краставицата с дълги деколте JIN5-508 и YN с късо деколте. Два ДНК басейна са конструирани от 50 екстремни индивиди с дълги деколтове и 50 екстремни индивида с къси деколте. Основен ефект QTL беше идентифициран на CHR07 чрез BSA анализ и традиционно QTL картографиране. Кандидатският регион беше допълнително стеснен от фино картографиране, количествено определяне на генната експресия и трансгенни експерименти, които разкриват ключов ген при контролиране на дължината на шията, CSFNL7.1. В допълнение, полиморфизмът в CSFNL7.1 промоторния регион е свързан със съответната експресия. По -нататъшният филогенетичен анализ предполага, че локусът на FNL7.1 е много вероятно да произхожда от Индия.
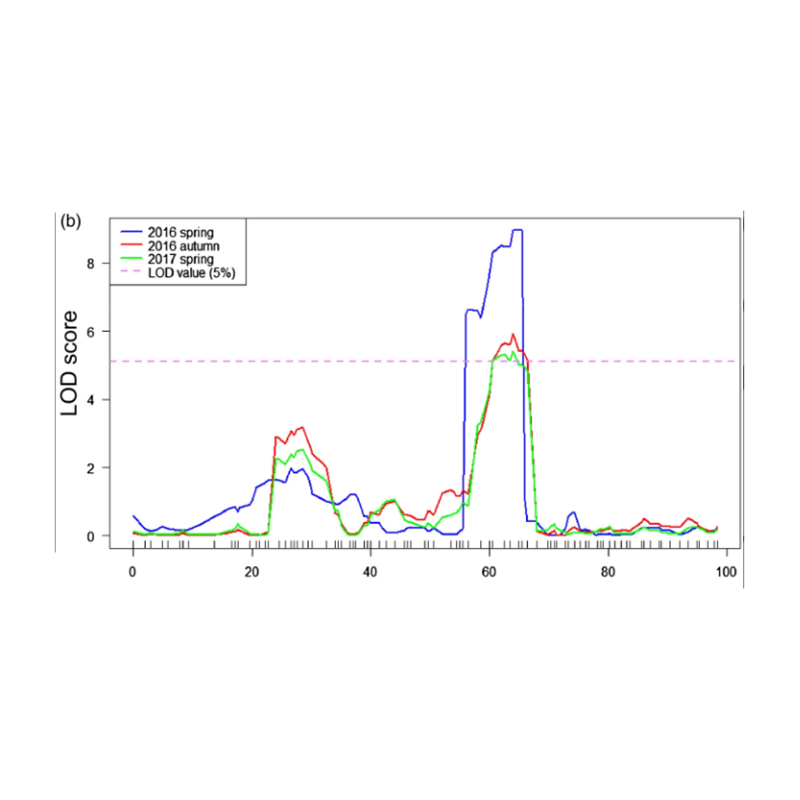
 QTL-картографиране в BSA анализ за идентифициране на кандидат-регион, свързан с дължината на шията на краставицата |  LOD профили на QTL с дължина на шията на краставицата, идентифицирани на CHR07 |
Xu, X., et al. „Количественият локус на количествената черта FNL7.1 кодира късен ембриогенеза изобилен протеин, свързан с дължината на плодовата шия в краставицата.“ Списание за биотехнология на растенията 18.7 (2020).