16S/18S/ITS ампликонно секвениране-PacBio
Характеристики на услугата
● Платформа за секвениране: PacBio Revio
● Режим на последователност: CCS (HiFi чете)
● Амплификация на целевата област, последвана от тандемно свързване на ампликони преди подготовката на HiFi SMRT bell библиотека
Предимства на услугата
●По-висока таксономична разделителна способност: Tхан късо ампликонно секвениране,позволявайки по-високи нива на класификация на OTU на ниво вид.
●Много точно базово повикване: PacBio CCS режим секвениране (HiFi четене).
●Без изолация: Бърза идентификация на микробния състав в проби от околната среда.
●Широко приложим: Различни проучвания на микробната общност.
●Цялостен биоинформационен анализ: Най-новият пакет QIIME2 (количествен поглед върху микробната екология) с различни анализи по отношение на база данни, анотация, OTU/ASV.
●Обширен експертен опит: С хиляди проекти за секвениране на ампликони, провеждани годишно, BMKGENE носи над десетилетие опит, висококвалифициран екип за анализи, изчерпателно съдържание и отлична поддръжка след продажбата.
Сервизни спецификации
| Библиотека | Стратегия за последователност | Препоръчителни данни |
| Ампликон | PacBio Revio | 10/30/50 K тагове (CCS) |
Примерни изисквания
| Концентрация (ng/µL) | Общо количество (µg) | Обем (µL) |
| ≥5 | ≥0,3 | ≥20 |
Препоръчителна доставка на мостри
Замразете пробите в течен азот за 3-4 часа и ги съхранявайте в течен азот или -80 градуса за дългосрочно съхранение. Изисква се мостра за доставка със сух лед.
Работен поток на услугата

Доставка на мостри

Изграждане на библиотека

Секвениране

Анализ на данни

Следпродажбени услуги
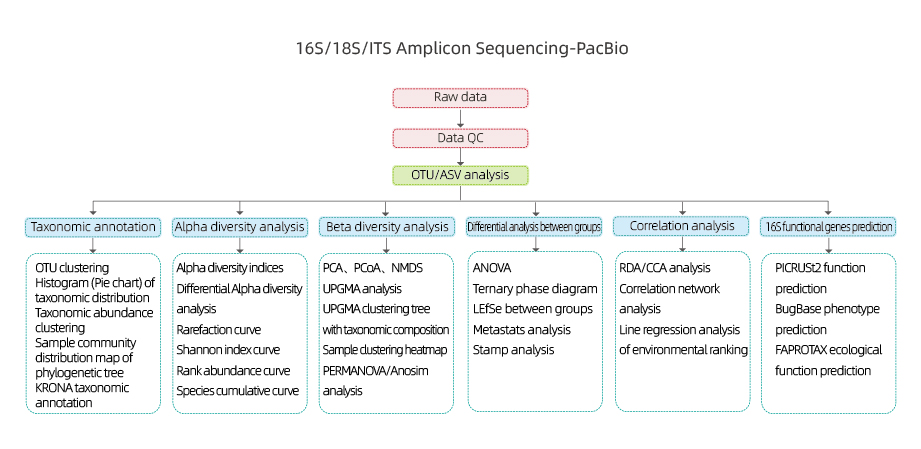
Включва следния анализ:
●Контрол на качеството на необработените данни
●OTU групиране/де-шум (ASV)
●OTU анотация
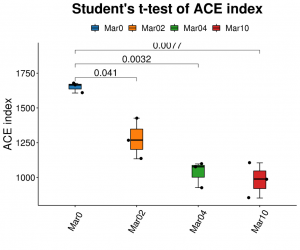
● Алфа анализ на разнообразието: множество индекси, включително Shannon, Simpson и ACE.
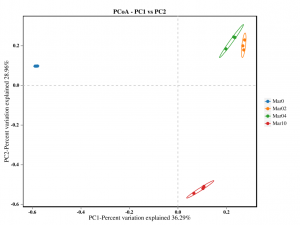
● Бета анализ на разнообразието
●Междугрупов анализ
●Корелационен анализ: между факторите на околната среда и състава и разнообразието на OUT
● 16S функционално прогнозиране на ген
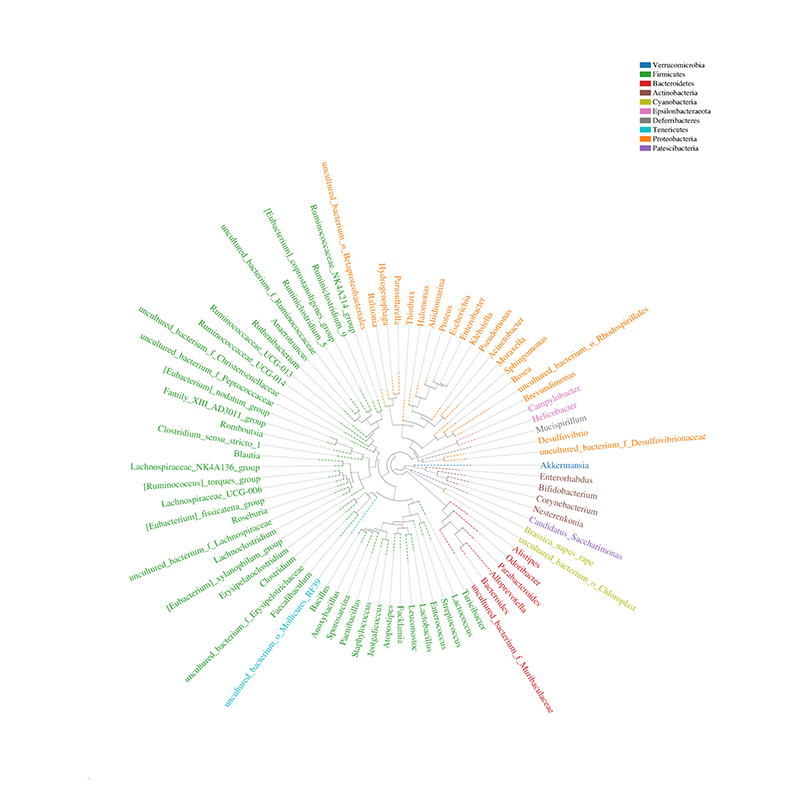
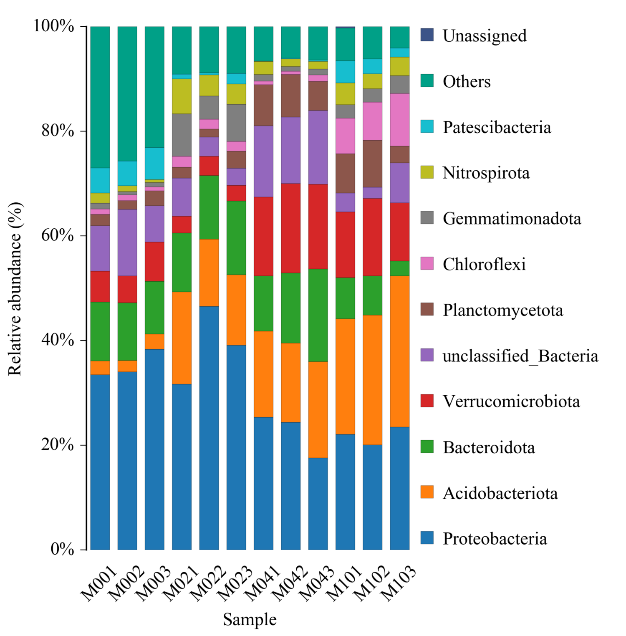
Хистограма на таксономично разпределение
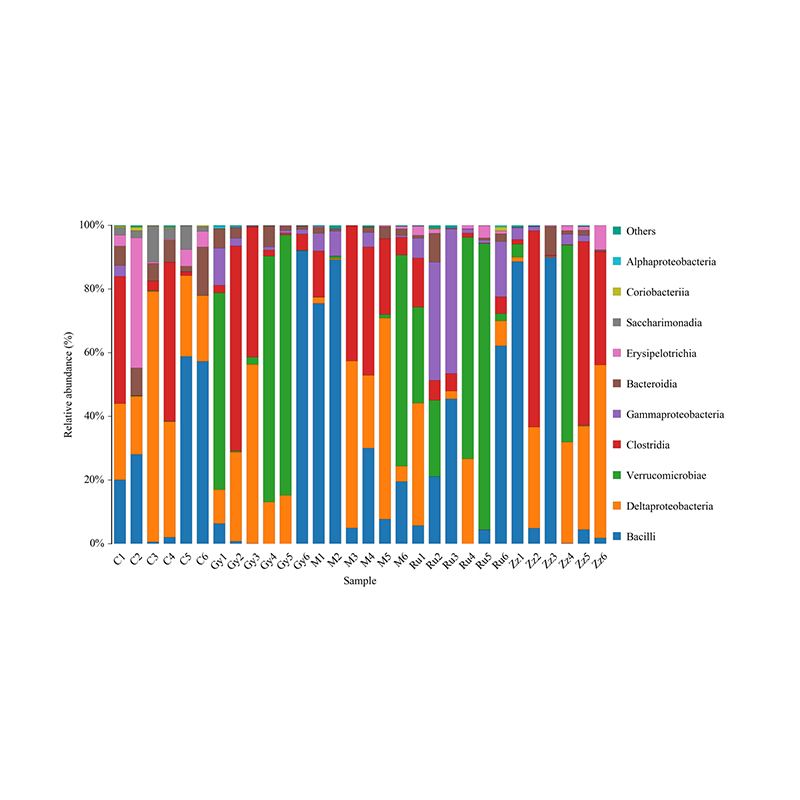
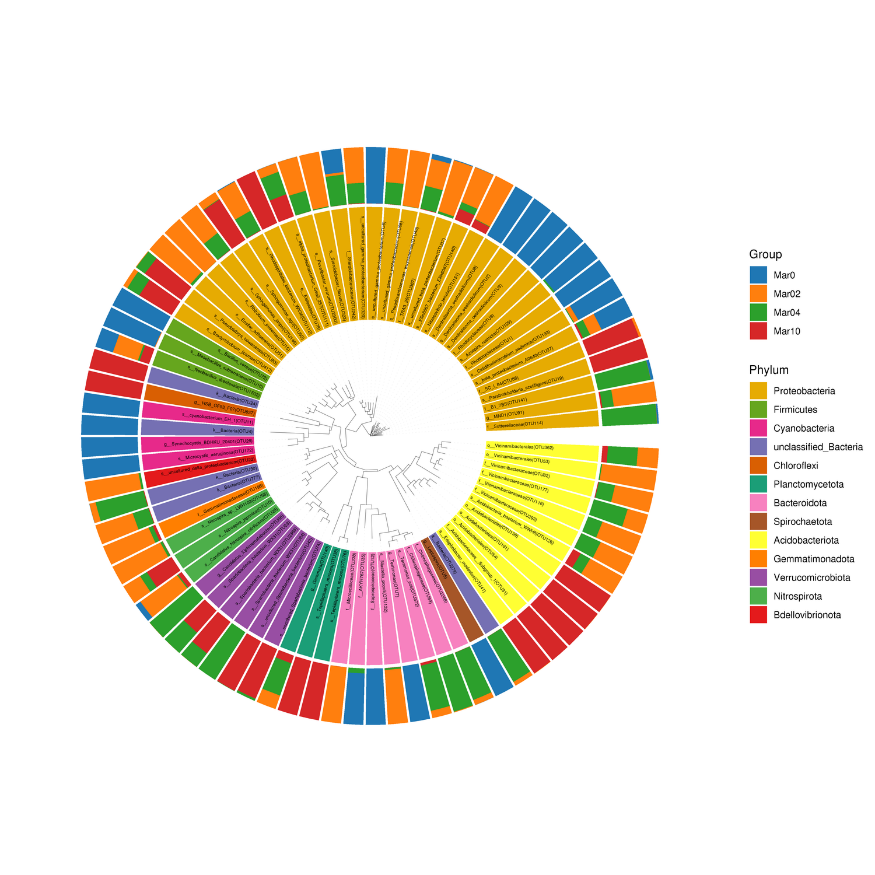
Филогенетично дърво на разпространение на общността
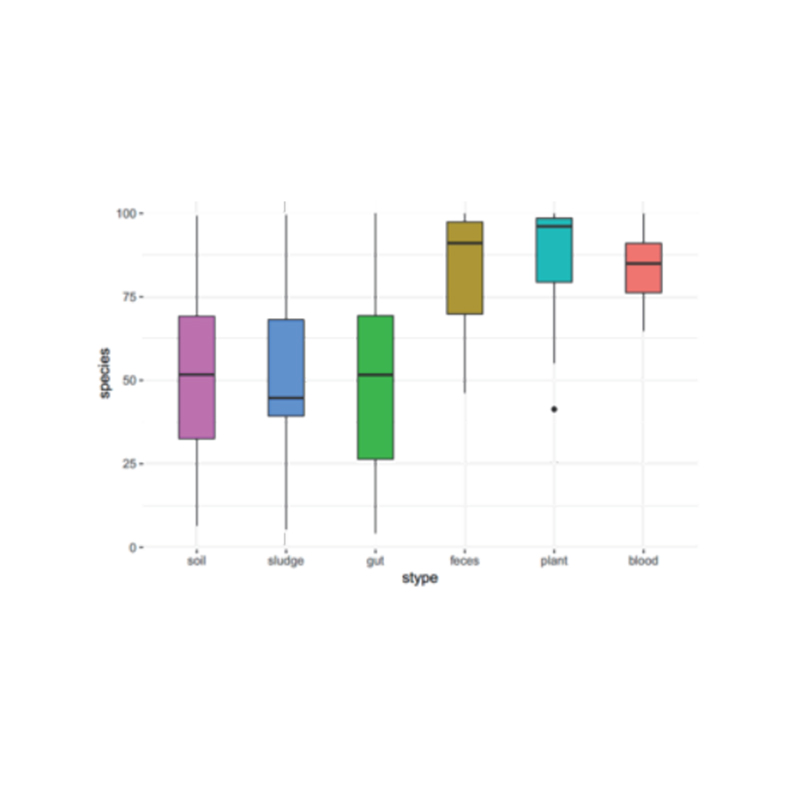
Алфа анализ на разнообразието: ACE
Бета анализ на разнообразието: PCoA
Междугрупов анализ: ANOVA
Разгледайте напредъка, улеснен от услугите за секвениране на ампликони на BMKGene с PacBio чрез подбрана колекция от публикации.
Гао, X. и Уанг, Х. (2023) „Сравнителен анализ на бактериални профили и функции на румен по време на адаптация към различна фенология (червенозелени срещу тревисти) при алпийски мериносови овце с два етапа на растеж на алпийска поляна“, Ферментация, 9( 1), стр. 16. doi: 10.3390/ФЕРМЕНТАЦИЯ9010016/S1.
Li, S. et al. (2023) „Улавяне на микробната тъмна материя в пустинни почви с помощта на базирана на културомика метагеномика и анализ с висока разделителна способност“, npj Biofilms and Microbiomes 2023 9:1, 9(1), стр. 1–14. doi: 10.1038/s41522-023-00439-8.
Mu, L. et al. (2022) „Ефекти на солите на мастни киселини върху ферментационните характеристики, бактериалното разнообразие и аеробната стабилност на смесения силаж, приготвен с люцерна, оризова слама и пшенични трици“, Journal of the Science of Food and Agriculture, 102(4), стр. 1475– 1487. doi: 10.1002/JSFA.11482.
Yang, J. et al. (2023) „Взаимодействието между биомаркери на оксидативен стрес и чревна микробиота в антиоксидантните ефекти на екстракти от Sonchus brachyotus DC. в индуциран от оксазолон чревен оксидативен стрес при възрастни рибки данио“, Антиоксиданти 2023 г., том. 12, страница 192, 12(1), стр. 192. doi: 10.3390/ANTIOX12010192.