10x пространствен транскриптом на Genomics Visium
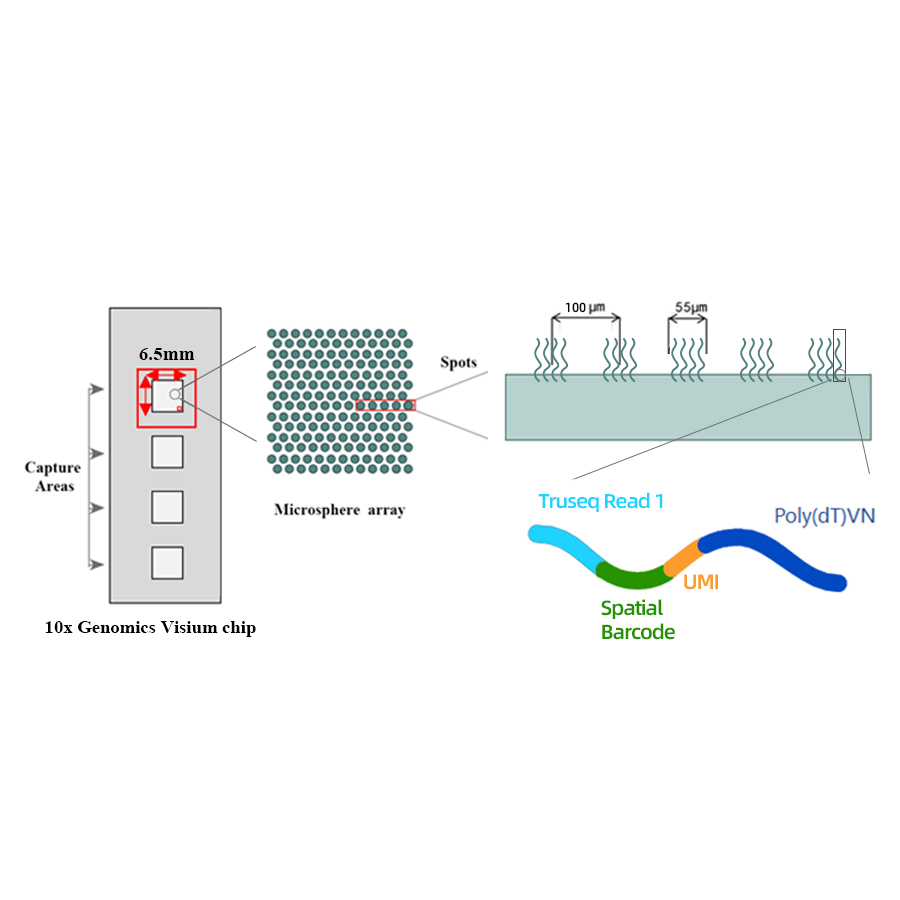
Техническа схема

Характеристики
● Разделителна способност: 100 µM
● Диаметър на петното: 55 µM
● Брой места: 4992
● Зона на заснемане: 6.5 x 6.5 mm
● Всяко място с баркод е заредено с праймери, съставени от 4 секции:
- поли(dT) опашка за праймиране на иРНК и синтез на кДНК
- Уникален молекулярен идентификатор (UMI) за коригиране на отклонението при усилване
- Пространствен баркод
- Свързваща последователност на праймер за секвениране на частично четене 1
● H&E оцветяване на срезове
Предимства
●Обслужване на едно гише: интегрира всички стъпки, базирани на опит и умения, включително крио-срезове, оцветяване, оптимизиране на тъканите, пространствено баркодиране, подготовка на библиотека, секвениране и биоинформатика.
● Висококвалифициран технически екип: с опит в над 250 вида тъкани и 100+ вида, включително хора, мишки, бозайници, риби и растения.
●Актуализация в реално време на целия проект: с пълен контрол върху експерименталния прогрес.
●Изчерпателна стандартна биоинформатика:пакетът включва 29 анализа и 100+ висококачествени фигури.
●Персонализиран анализ на данни и визуализация: достъпно за различни заявки за изследване.
●Допълнителен съвместен анализ с едноклетъчно иРНК секвениране
Спецификации
| Примерни изисквания | Библиотека | Стратегия за последователност | Препоръчителни данни | Контрол на качеството |
| OCT-вградени криопроби (Оптимален диаметър: прибл. 6x6x6 mm³) 2 блока на проба | 10X Visium cDNA библиотека | Ilumina PE150 | 50K PE четения на място (60Gb) | RIN > 7 |
За повече подробности относно насоките за подготовка на проби и работния процес на обслужване, моля, не се колебайте да говорите с aBMKGENE експерт
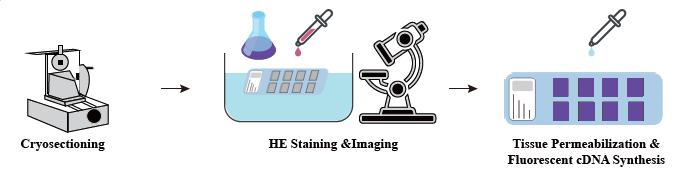
Работен процес на услугата
Във фазата на подготовка на пробата се извършва първоначален опит за екстракция на масова РНК, за да се гарантира, че може да се получи висококачествена РНК. В етапа на тъканна оптимизация срезовете се оцветяват и визуализират и условията на пермеабилизация за освобождаване на иРНК от тъканта се оптимизират. След това оптимизираният протокол се прилага по време на изграждането на библиотеката, последвано от последователност и анализ на данните.
Пълният работен процес на услугата включва актуализации в реално време и клиентски потвърждения за поддържане на отзивчива верига за обратна връзка, гарантираща гладко изпълнение на проекта.

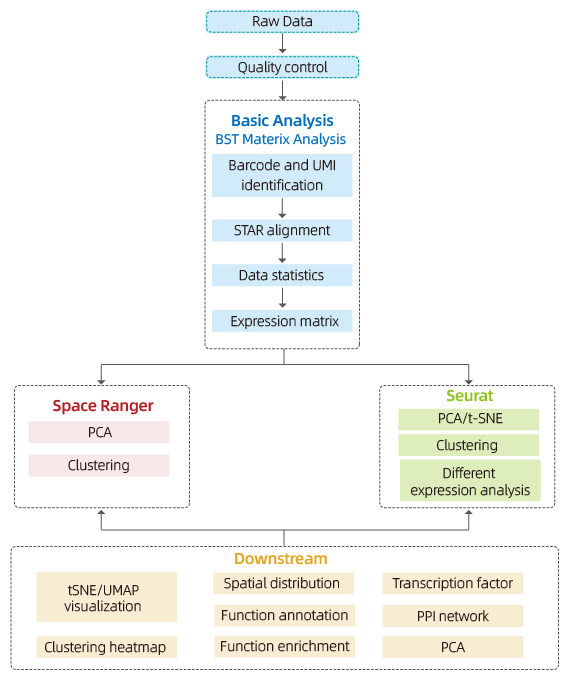
Включва следния анализ:
Контрол на качеството на данните:
o Извеждане на данни и разпределение на резултата за качество
o Откриване на ген на място
o Покритие на тъканите
Анализ на вътрешната проба:
o Генно богатство
o Групиране на място, включително анализ на намалени измерения
o Анализ на диференциална експресия между клъстери: идентифициране на маркерни гени
o Функционална анотация и обогатяване на маркерни гени
Междугрупов анализ
o Повторно комбиниране на петна от двете проби (напр. болни и контролни) и повторно групиране
o Идентифициране на маркерни гени за всеки клъстер
o Функционална анотация и обогатяване на маркерни гени
o Диференциално изразяване на един и същ клъстер между групите
Анализ на вътрешна извадка
Групиране на място

Идентификация на маркерни гени и пространствено разпределение


Междугрупов анализ
Комбинация от данни от двете групи и повторно групиране
Маркерни гени на нови клъстери
Разгледайте напредъка, улеснен от услугата за пространствена транскриптомия на BMKGene от 10X Visium В тези представени публикации:
Chen, D. et al. (2023) „mthl1, потенциален хомолог на Drosophila на адхезионни GPCRs на бозайници, участва в антитуморни реакции към инжектирани онкогенни клетки в мухи“,Сборник на Националната академия на науките на Съединените американски щати, 120(30), стр. e2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) „STEEL позволява очертаване с висока разделителна способност на пространствено-времеви транскриптомни данни“,Брифинги по биоинформатика, 24(2), стр. 1–10. doi: 10.1093/BIB/BBAD068.
Liu, C. et al. (2022) „Пространствено-времеви атлас на органогенезата в развитието на цветята на орхидеята“,Изследване на нуклеинови киселини, 50 (17), стр. 9724–9737. doi: 10.1093/NAR/GKAC773.
Wang, J. et al. (2023) „Интегрирането на пространствената транскриптомика и едноядреното РНК секвениране разкрива потенциалните терапевтични стратегии за лейомиома на матката“,Международен журнал за биологични науки, 19 (8), стр. 2515–2530. doi: 10.7150/IJBS.83510.