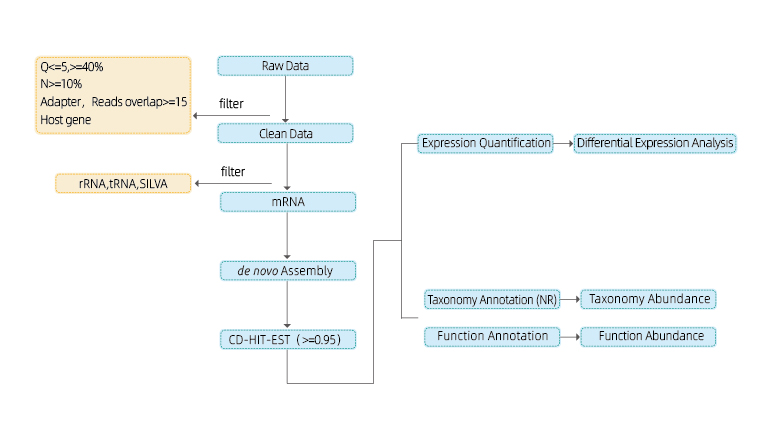
Паслядоўнасць Metatranscriptome
Асаблівасці абслугоўвання
● Знішчэнне рРНК з наступнай падрыхтоўкай бібліятэкі мРНК.
● Паслядоўнасць на Illumina Novaseq.
Перавагі абслугоўвання
●Вывучыце змены складаных мікробных супольнасцей:Гэта адбываецца на ўзроўні транскрыпцыі і вывучае новыя патэнцыйныя гены.
●Тлумачэнне ўзаемадзеяння мікробнай супольнасці з гаспадаром або навакольным асяроддзем.
●Комплексны біяінфарматычны аналіз: Гэта дае ўяўленне аб таксанамічных і функцыянальных кампазіцыях грамадства, а таксама дыферэнцыяльнага аналізу экспрэсіі генаў.
●Шырокая анатацыя генаў:Выкарыстанне сучасных баз дадзеных функцый генаў для інфарматыўнай інфармацыі пра экспрэсію генаў мікробных супольнасцей.
●Падтрымка пасля продажаў:Наша абавязацельства выходзіць за рамкі завяршэння праекта з 3-месячным перыядам пасля продажу. За гэты час мы прапануем наступныя дзеянні праекта, дапамогу па ліквідацыі непаладак і сесіі па пытаннях і пытаннях для вырашэння любых запытаў, звязаных з вынікамі.
Тэхнічныя характарыстыкі службы
| Платформа паслядоўнасці | Стратэгія паслядоўнасці | Рэкамендаваныя дадзеныя | Кантроль якасці дадзеных |
| Illumina novaseq | PE150 | 12 ГБ | Q30≥85% |
Патрабаванні да ўзору
| Канцэнтрацыя (нг/мкл) | Агульная колькасць (мкг) | Аб'ём (µL) | OD260/280 | OD260/230 | Рын |
| ≥50 | ≥1,0 | ≥20 | 1.8-2.0 | 1,0-2,5 | ≥6,5 |
Працоўны паток абслугоўвання

Дастаўка ўзору

Будаўніцтва бібліятэкі

Паслядоўнасць

Аналіз дадзеных

Паслугі пасля продажу
Уключае ў сябе наступны аналіз:
● Секвенсія кантролю якасці дадзеных
● Асамблея стэнаграмы
● Таксанамічная анатацыя і багацце
● Функцыянальная анатацыя і багацце
● колькаснае вызначэнне выразаў і дыферэнцыяльны аналіз
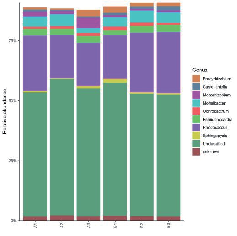
Таксанамічнае размеркаванне кожнага ўзору:
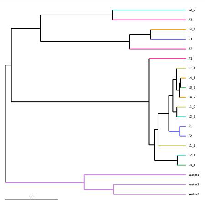
Аналіз бэта -разнастайнасці: UPGMA
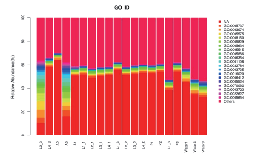
Функцыянальная анатацыя - ідзіце багаццем
Багацце дыферэнцыяльнай таксанаміі - Lefse
Вывучыце дасягненні, якія садзейнічалі паслугам паслядоўнасці мета -транскрыптыкі BMKGene праз кураваную калекцыю публікацый.
Лу, З. і інш. (2023) "Кіслата талерантнасць да лактата, які выкарыстоўвае бактэрыі парадку бактэроідалаў, спрыяе прафілактыцы румінальнага ацыдозу ў коз, адаптаваных да дыеты з высокай канцэнтрацыяй", ",",Харчаванне жывёл, 14, с. 130–140. doi: 10.1016/j.aninu.2023.05.006.
Песня, З. і інш. (2017) "Разгадванне асноўнай функцыянальнай мікрабіёты ў традыцыйнай цвёрдацельнай закісанні з дапамогай ампліконаў з высокай прапускной здольнасцю і паслядоўнасці метатранскрыптымікі",Мяжы ў мікрабіялогіі, 8 (ліпень). doi: 10.3389/fmicb.2017.01294/поўная.
Ван, У. і інш. (2022) "Раман мікавірусы, выяўленыя з метатранскрыптымікі, фітапатагеннага грыба", ",", ",", ",", ",",Вірусы, 14 (11), с. 2552. Doi: 10.3390/v14112552/s1.
Вэй, Дж. І інш. (2022) "Паралельны аналіз метатранскрыпту паказвае дэградацыю другасных метабалітаў раслін жукамі і іх сімбіёнтамі кішачніка", ",Малекулярны Экалогія, 31 (15), стар. 3999–4016. doi: 10.1111/mec.16557.