Menslike hele eksoom-volgordebepaling
Dienskenmerke
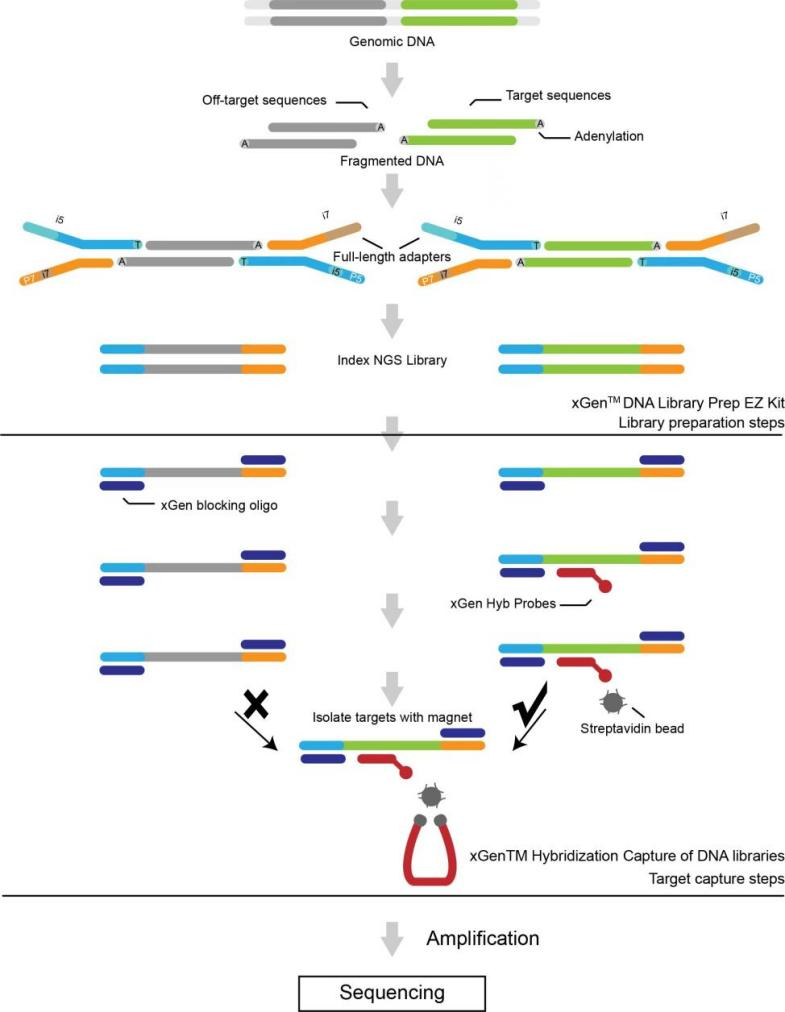
● Twee eksoompanele beskikbaar gebaseer op teikenverryking met probes: Sure Select Human All Exon v6 (Agilent) en xGen Exome Hibridization Panel v2 (IDT).
● Opeenvolging op Illumina NovaSeq.
● Bioinformatiese pyplyn gerig op siekte-analise of tumor-analise.
Diensvoordele
●Teiken Proteïenkoderingstreek: Deur die vaslegging en volgorde van proteïenkoderende streke, word hWES gebruik om variante wat verband hou met proteïenstruktuur te openbaar.
●Koste-effektief:hWES lewer ongeveer 85% van die menslike siekte-geassosieerde mutasies uit 1% van die menslike genoom.
●Hoë akkuraatheid: Met 'n hoë volgorde-diepte, fasiliteer hWES die opsporing van beide algemene variante en skaars variante met frekwensies laer as 1%.
●Streng kwaliteitbeheer: Ons implementeer vyf kernbeheerpunte oor alle stadiums, van monster- en biblioteekvoorbereiding tot volgordebepaling en bioinformatika. Hierdie noukeurige monitering verseker die lewering van konsekwent hoë kwaliteit resultate.
●Omvattende bioinformatika-analise: ons pyplyn gaan verder as die identifisering van variasies aan die verwysingsgenoom, aangesien dit gevorderde analise insluit wat ontwerp is om spesifiek navorsingsvrae wat verband hou met genetiese aspekte van siektes of tumoranalise aan te spreek.
●Ondersteuning na verkope:Ons verbintenis strek verder as projekvoltooiing met 'n 3-maande na-verkope dienstydperk. Gedurende hierdie tyd bied ons projekopvolging, probleemoplossingshulp en V&A-sessies aan om enige navrae wat met die resultate verband hou, aan te spreek.
Voorbeeld spesifikasies
| Exon Capture Strategie | Opeenvolgingstrategie | Aanbevole data-uitvoer |
| Seker kies Human All Exon v6 (Agilent) of xGen Exome Hibridization Panel v2 (IDT)
| Illumina NovaSeq PE150 | 5 -10 Gb Vir mendeliese afwykings/skaars siektes: > 50x Vir tumormonsters: ≥ 100x |
Voorbeeldvereistes
| Voorbeeld tipe
| Bedrag(Qubit®)
| Konsentrasie | Volume
| Suiwerheid (NanoDrop™) |
|
Genomiese DNA
| ≥ 50 ng | ≥ 6 ng/μL | ≥ 15 μL | OD260/280=1.8-2.0 geen agteruitgang, geen kontaminasie
|
Bioinformatika

Bioinformatiese ontleding van hWES-siektemonsters sluit in:
● Opeenvolging van data QC
● Verwysingsgenoombelyning
● Identifikasie van SNP's en InDels
● Funksionele annotasie van SNP's en InDels

Bioinformatiese ontleding van tumormonsters sluit in:
● Opeenvolging van data QC
● Verwysingsgenoombelyning
● Identifikasie van SNP's, InDels en somatiese variasies
● Identifikasie van kiemlynvariante
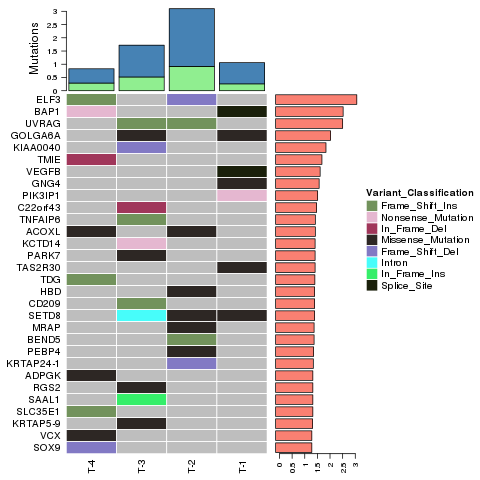
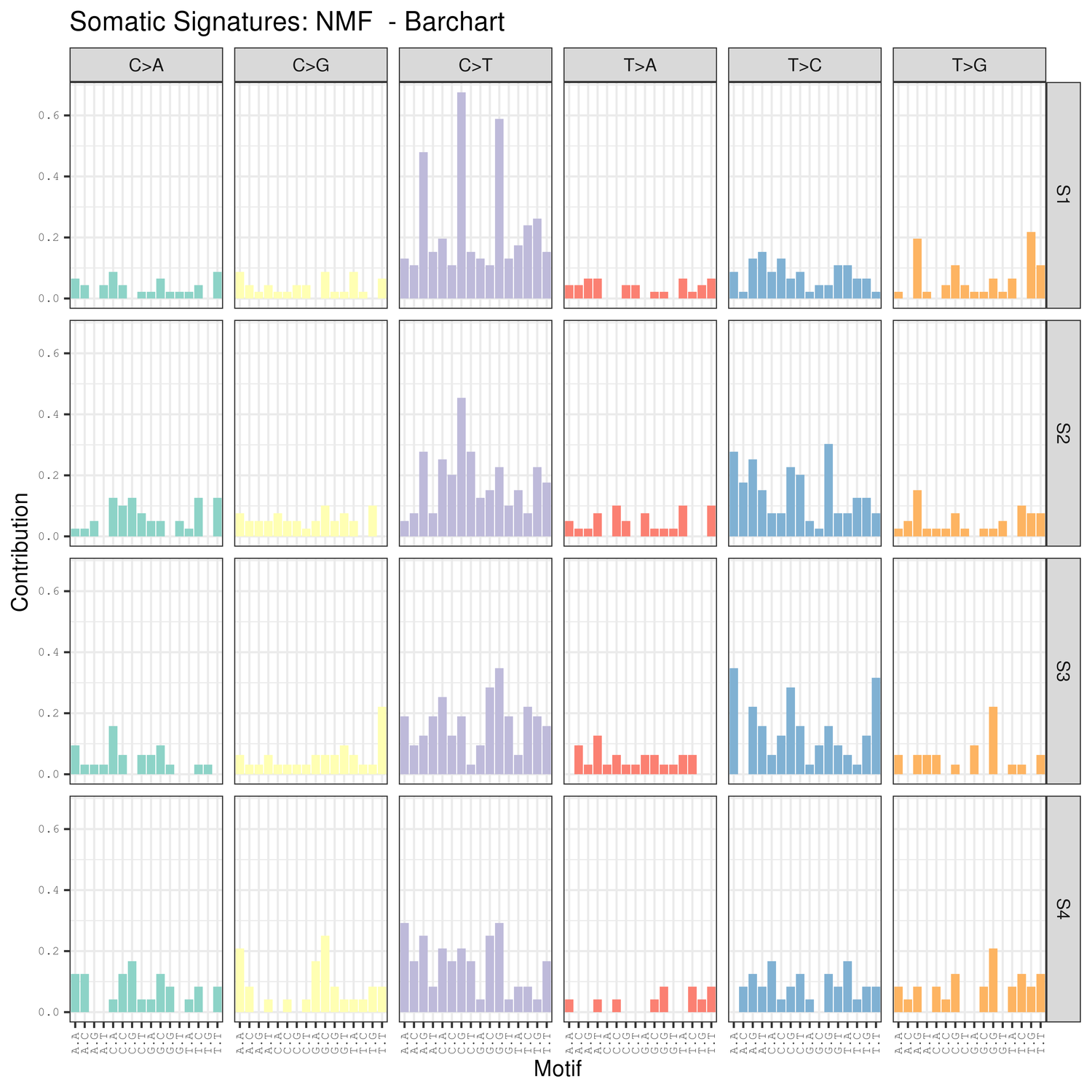
● Mutasie-handtekeninge-analise
● Identifikasie van dryfgene gebaseer op wins-van-funksie mutasies
● Mutasie-annotasie op die vlak van geneesmiddelgevoeligheid
● Heterogeniteitsanalise – berekening van suiwerheid en ploïdie
Dienswerkvloei

Voorbeeld aflewering

DNA onttrekking

Biblioteek konstruksie

Opeenvolging

Data-analise

Data aflewering

Na-verkope dienste
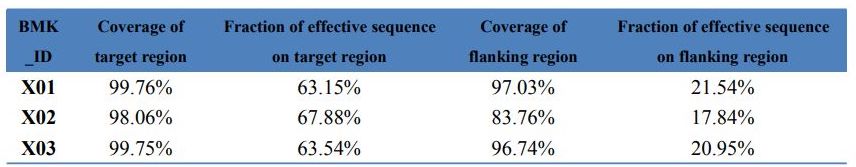
Data QC – Statistiek van Exome-vaslegging
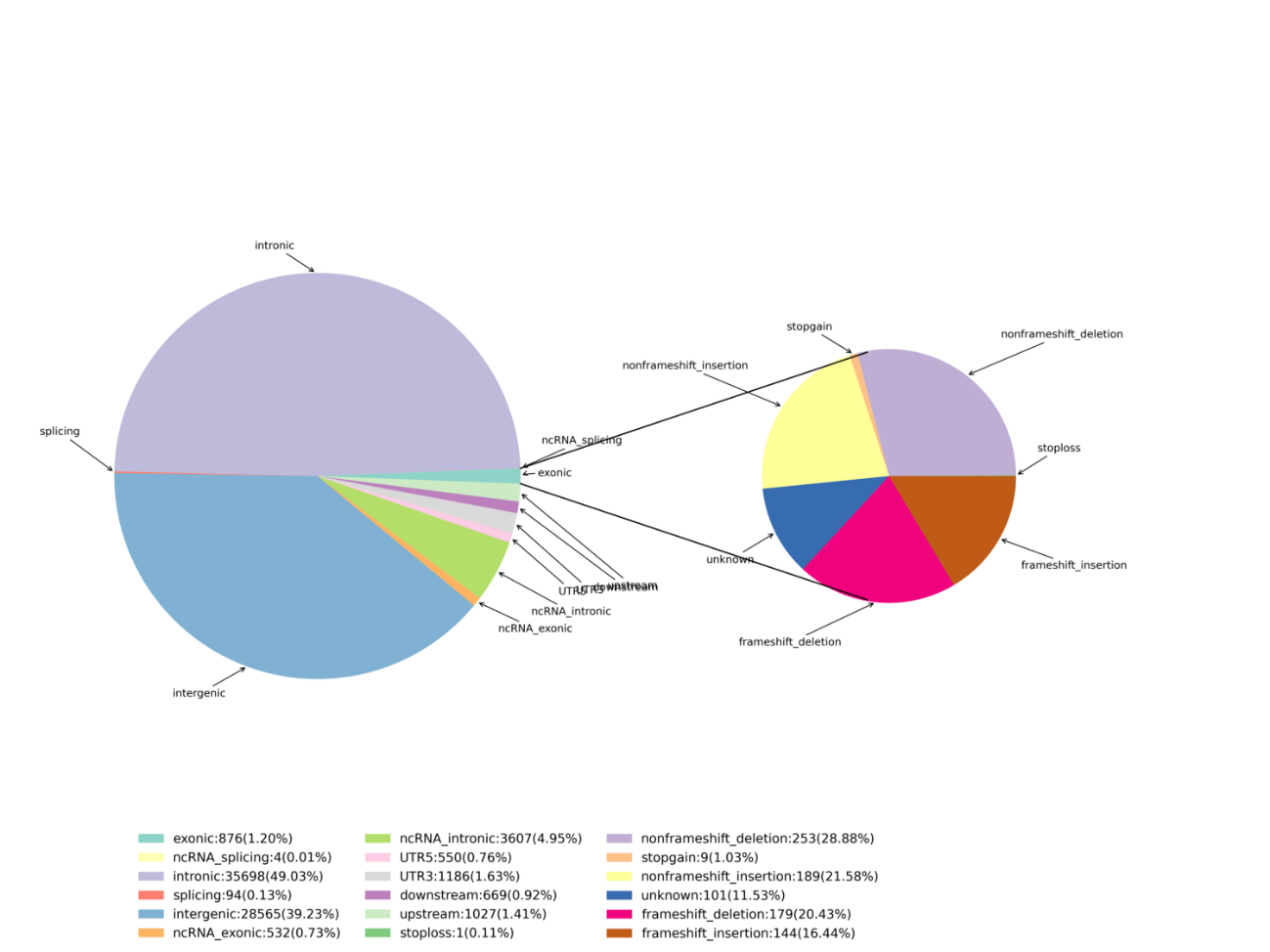
Variant identifikasie – InDels
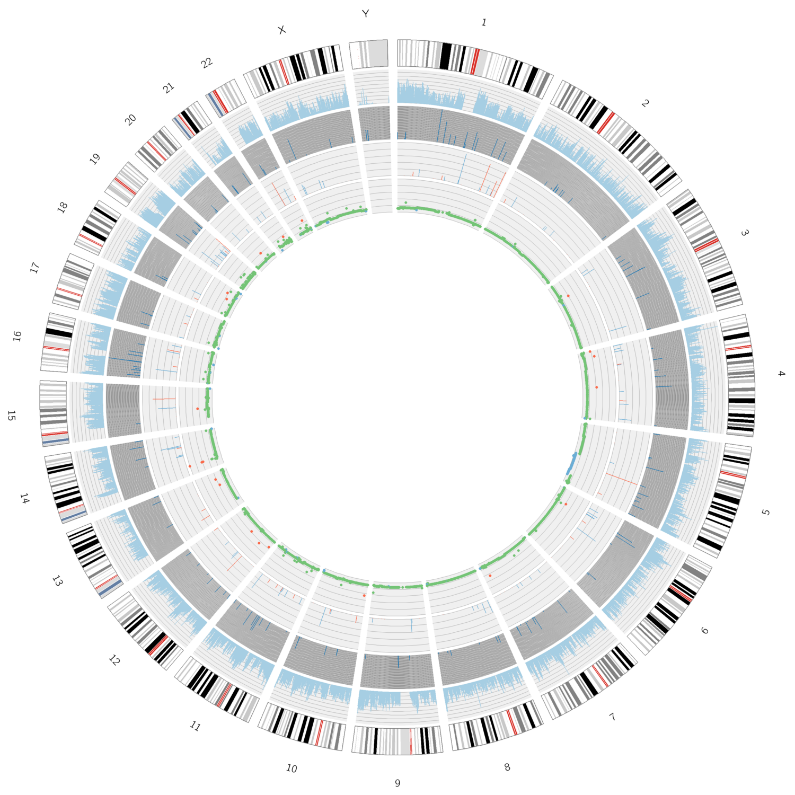
Gevorderde analise: identifikasie en verspreiding van skadelike SNPs/InDels – Circos plot
Tumoranalise: identifikasie en verspreiding van somatiese mutasies – Circos plot
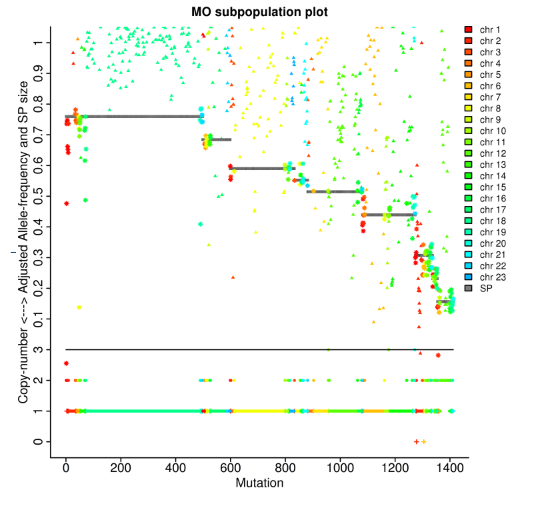
Tumoranalise: klonale afstammelinge