Hi-C-gebaseerde genoomsamestelling
Dienskenmerke
● Opeenvolging op Illumina NovaSeq met PE150.
● Diens vereis weefselmonsters, in plaas van onttrekte nukleïensure, om met formaldehied te kruisbind en die DNA-proteïen-interaksies te bewaar.
● Die Hi-C-eksperiment behels beperking en eindherstel van die taai punte met biotien, gevolg deur sirkulasie van die gevolglike stomp punte, terwyl die interaksies bewaar word. Die DNA word dan met streptavidien-krale afgetrek en gesuiwer vir daaropvolgende biblioteekvoorbereiding.
Diensvoordele

Oorsig van Hi-C
(Lieberman-Aiden E et al.,Wetenskap, 2009)
●Elimineer die behoefte aan genetiese bevolkingsdata:Hi-C vervang die noodsaaklike inligting wat benodig word vir aaneenlopende anker.
●Hoë merkerdigtheid:lei tot 'n hoë aaneenlopende ankerverhouding bo 90%.
●Uitgebreide kundigheid en publikasie rekords:BMKGene het groot ondervinding met meer as 2000 gevalle van Hi-C Genome Assembly van 1000 verskillende spesies en verskeie patente. Meer as 200 gepubliseerde gevalle het 'n akkumulatiewe impakfaktor van meer as 2000.
●Hoogs vaardige bioinformatika-span:met interne patente en sagteware-kopieregte vir Hi-C-eksperimente en data-analise, maak die self-ontwikkelde visualiseringsdata-sagteware dit moontlik om blokke met die hand te verskuif, om te keer, te herroep en oor te doen.
●Ondersteuning na verkope:Ons verbintenis strek verder as projekvoltooiing met 'n 3-maande na-verkope dienstydperk. Gedurende hierdie tyd bied ons projekopvolging, probleemoplossingshulp en V&A-sessies aan om enige navrae wat met die resultate verband hou, aan te spreek.
●Omvattende aantekening: ons gebruik veelvuldige databasisse om die gene funksioneel met geïdentifiseerde variasies te annoteer en die ooreenstemmende verrykingsanalise uit te voer, wat insigte oor veelvuldige navorsingsprojekte verskaf.
Diensspesifikasies
| Biblioteekvoorbereiding | Opeenvolgingstrategie | Aanbevole data-uitvoer | Gehaltebeheer |
| Hi-C biblioteek | Illumina NovaSeq PE150 | 100x | Q30 ≥ 85% |
Voorbeeldvereistes
| Weefsel | Vereiste bedrag |
| Diere Viscera | ≥ 2 g |
| Dierespier | |
| Soogdier Bloed | ≥ 2 ml |
| Pluimvee/Visbloed | |
| Plant- Vars Blaar | ≥ 3 g |
| Gekweekte selle | ≥ 1x107 |
| Insek | ≥ 2 g |
Dienswerkvloei

Eksperiment ontwerp

Voorbeeld aflewering

Biblioteek konstruksie

Opeenvolging

Data-analise

Na-verkope dienste
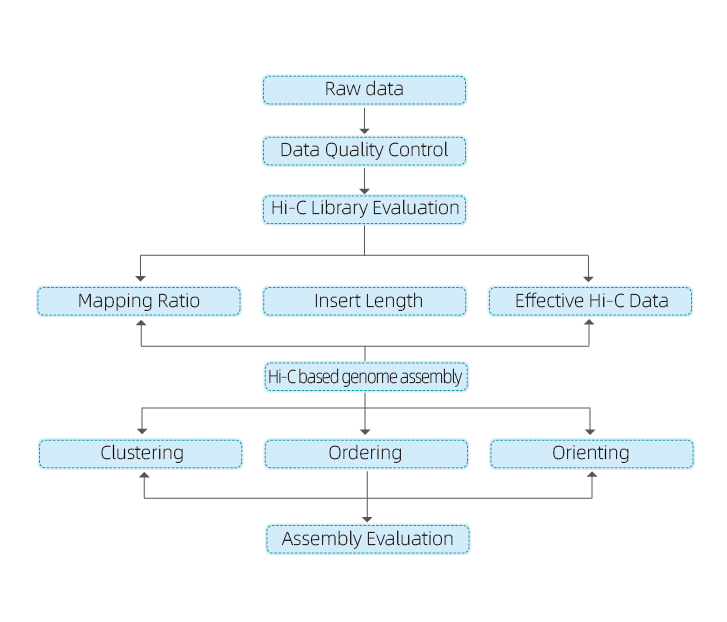
1) Rou data QC
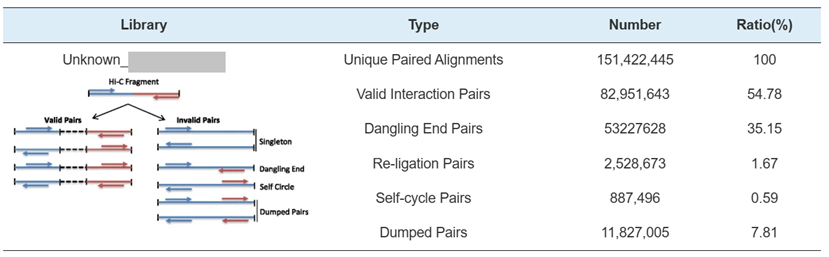
2) Hi-C biblioteek QC: skatting van geldige Hi-C interaksies
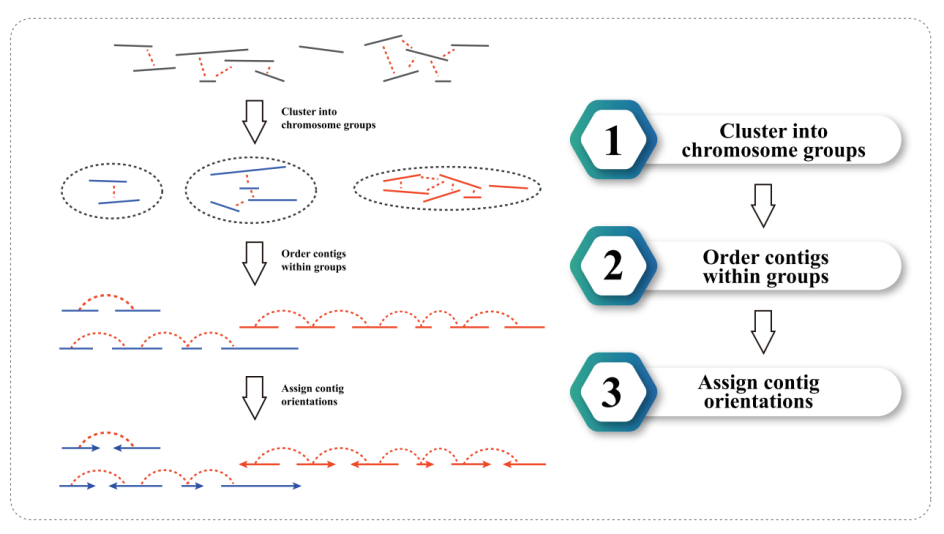
3) Hi-C-samestelling: groepering van contigs in groepe, gevolg deur contig-ordening binne elke groep en toewysing van contig-oriëntasie
4) Hi-C evaluering
Hi-C Library QC – skatting van Hi-C geldige interaksiepare
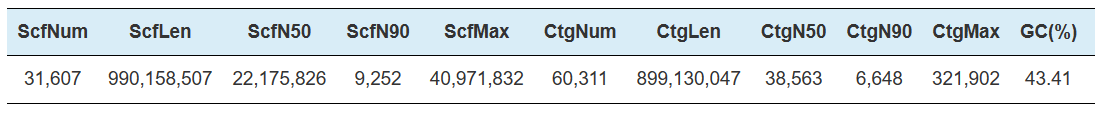
Hi-C Assembly – statistieke
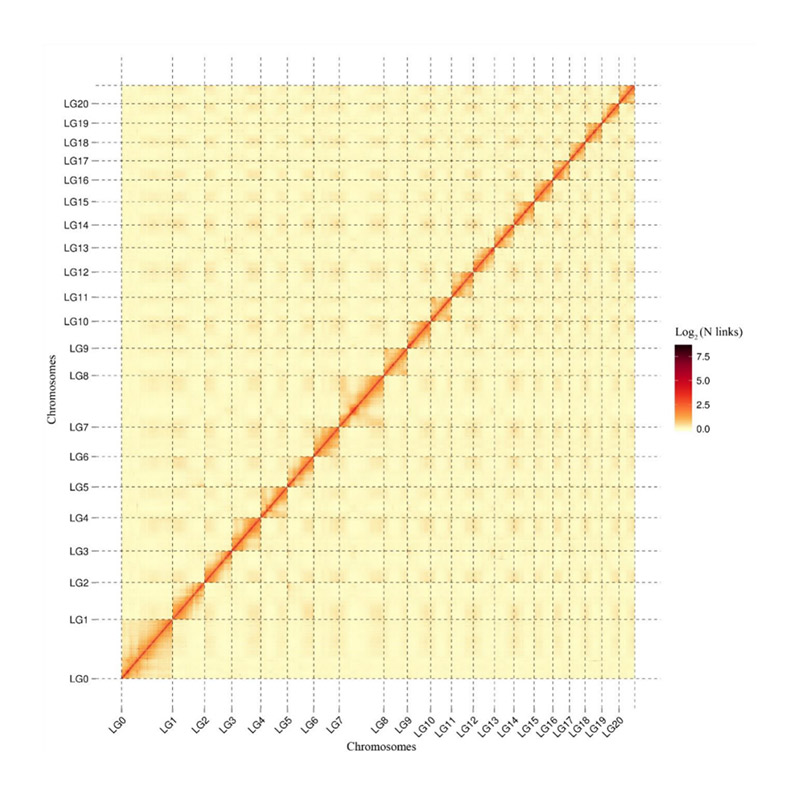
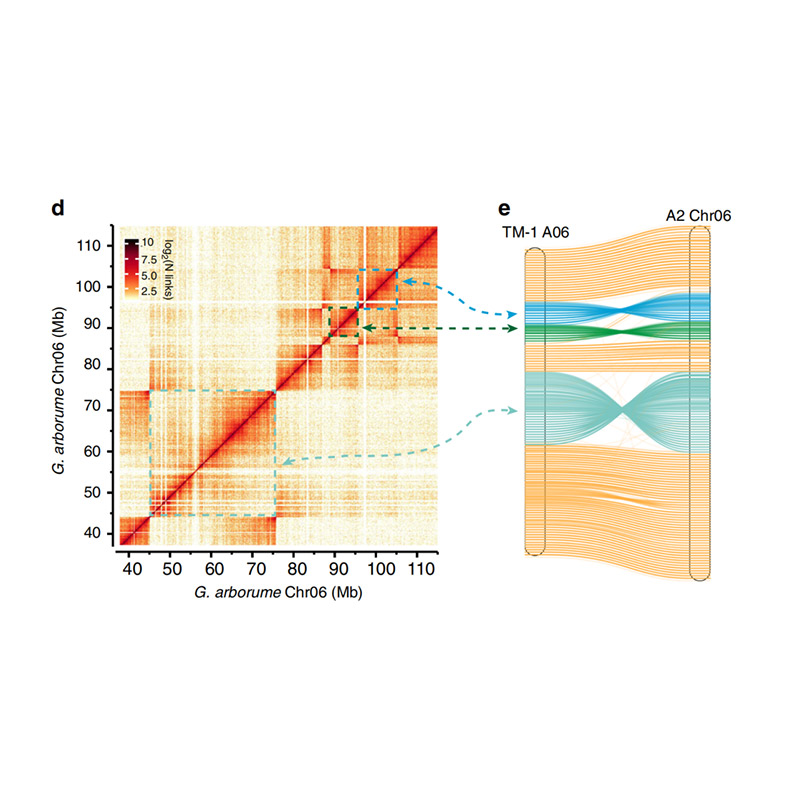
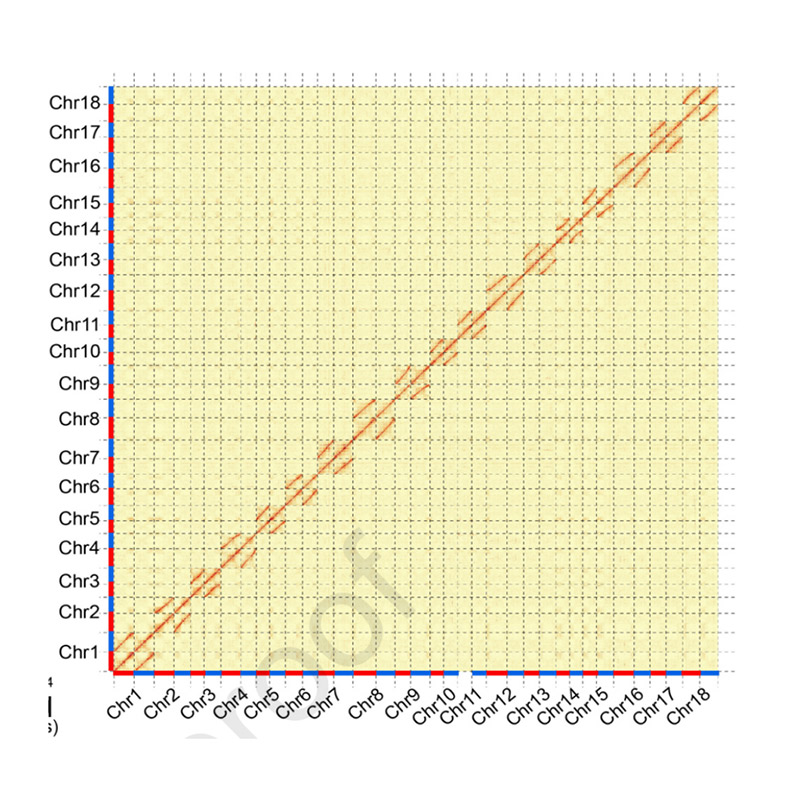
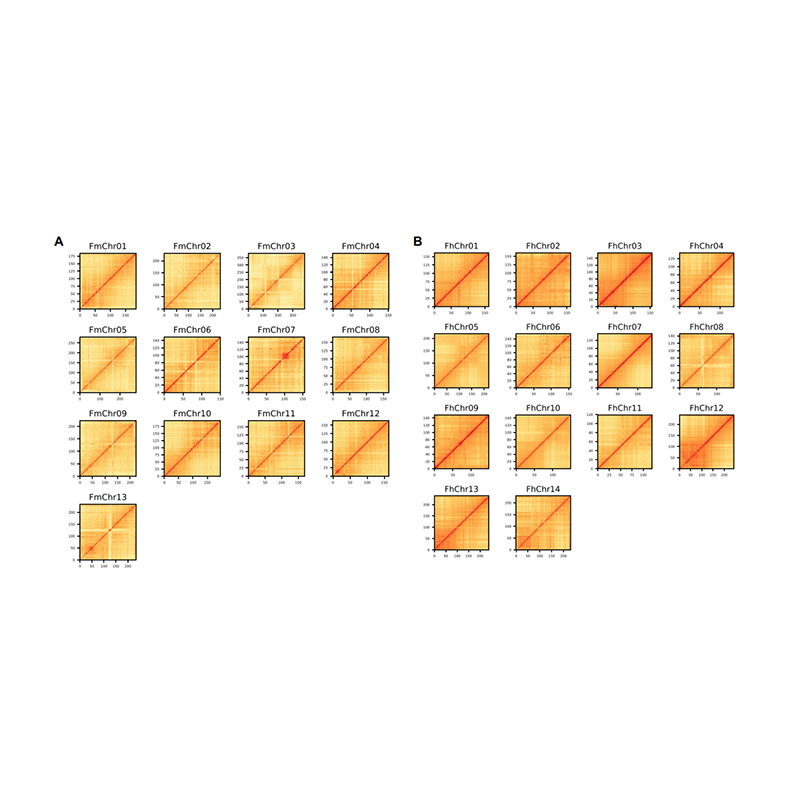
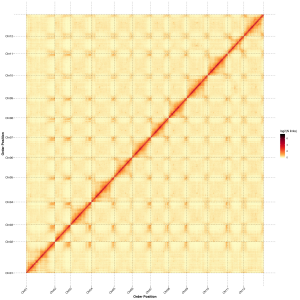
Na-samestelling evaluering – hittekaart van seinintensiteit tussen houers
Verken die vooruitgang wat deur BMKGene se Hi-C-monteerdienste gefasiliteer word deur 'n saamgestelde versameling publikasies.
Tian, T. et al. (2023) 'Genoomsamestelling en genetiese disseksie van 'n prominente droogtebestande mieliekiemplasma', Nature Genetics 2023 55:3, 55(3), pp. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'A Chromosome-Scale Assembly of the Asian Heuningby Apis cerana Genome', Frontiers in Genetics, 11, p. 524140. doi: 10.3389/FGENE.2020.00279/BIBTEX.
Zhang, F. et al. (2023) 'Openbaring van evolusie van tropaanalkaloïedbiosintese deur twee genome in die Solanaceae-familie te ontleed', Nature Communications 2023 14:1, 14(1), pp. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) 'Genome van die Banyan-boom en bestuiwer-wesp bied insig in Fig-Wesp Co-evolution', Sel, 183(4), pp. 875-889.e17. doi: 10.1016/J.CELL.2020.09.043